Index >
Results for CNB 124950
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
124950_ENSG00000164600_HUMAN_8350_8467/1-120 GATGCCAGATGATCAGTGCAGAATGAAGGTCCCAAGAGA-GAGGATCACATGGCTCTCTC
124950_ENSRNOG00000026055_RAT_7205_7320/1-120 GATGCCAGATGATCAGTGCAGAATGAAGGTCCCAAGAGA-G--GATCACATGGCTCTCTC
124950_ENSMUSG00000037984_MOUSE_8436_8550/1-120 -ATGCCAGATGATCAGTGCAGAATGAAGGTCCCAAGAGA-G--GATCACATGGCTCTCTC
124950_SINFRUG00000142404_FUGU_9535_9652/1-120 GCTGCCAGATGATCGGTGCGGCGCGGAGGTCCCGTGGGACGCCGAACACGTGGTCCCGGC
************ **** * * ******* * ** * ** *** *** * *
124950_ENSG00000164600_HUMAN_8350_8467/1-120 TGCCCTGTGACGTCACTAGCAGATGGCATGGGTACCAGCT-CTGGCAGTTGGCATCAATG
124950_ENSRNOG00000026055_RAT_7205_7320/1-120 TGCCCTGTGACGTCACTAGCAGATGGCATGGGTACCAGCT-CTGGCAGTTGGCATCAATG
124950_ENSMUSG00000037984_MOUSE_8436_8550/1-120 TGCCCTGTGACGTCACTAGCAGATGGCATGGGTACCAGCT-CTGGCAGTTGGCATCAATG
124950_SINFRUG00000142404_FUGU_9535_9652/1-120 TGCCACAAGACGTCAGCGGCAGATGGCATCGGTACCCGCTACTGGCA-TGAGTGTCATT-
**** ******* *********** ****** *** ****** * * *** *
//
124950.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 120
Mean pairwise identity: 83.43
Mean single sequence MFE: -44.83
Consensus MFE: -30.59
Energy contribution: -30.27
Covariance contribution: -0.31
Mean z-score: -1.70
Structure conservation index: 0.68
SVM decision value: 0.23
SVM RNA-class probability: 0.646642
Prediction: RNA
######################################################################
>124950_ENSG00000164600_HUMAN_8350_8467/1-120
GAUGCCAGAUGAUCAGUGCAGAAUGAAGGUCCCAAGAGAGAGGAUCACAUGGCUCUCUCUGCCCUGUGACGUCACUAGCAGAUGGCAUGGGUACCAGCUCUGGCAGUUGGCAUCAAUG
((((((((.((.((((.((........(((((((((((((((..........))))))))(((((((..........))))..))).)))).))).)).)))))).)))))))).... ( -46.20)
>124950_ENSRNOG00000026055_RAT_7205_7320/1-120
GAUGCCAGAUGAUCAGUGCAGAAUGAAGGUCCCAAGAGAGGAUCACAUGGCUCUCUCUGCCCUGUGACGUCACUAGCAGAUGGCAUGGGUACCAGCUCUGGCAGUUGGCAUCAAUG
((((((((.((.((((.((........(((((((((((((..((....))..))))))(((((((..........))))..))).)))).))).)).)))))).)))))))).... ( -45.40)
>124950_ENSMUSG00000037984_MOUSE_8436_8550/1-120
AUGCCAGAUGAUCAGUGCAGAAUGAAGGUCCCAAGAGAGGAUCACAUGGCUCUCUCUGCCCUGUGACGUCACUAGCAGAUGGCAUGGGUACCAGCUCUGGCAGUUGGCAUCAAUG
(((((((.((.((((.((........(((((((((((((..((....))..))))))(((((((..........))))..))).)))).))).)).)))))).)))))))..... ( -40.90)
>124950_SINFRUG00000142404_FUGU_9535_9652/1-120
GCUGCCAGAUGAUCGGUGCGGCGCGGAGGUCCCGUGGGACGCCGAACACGUGGUCCCGGCUGCCACAAGACGUCAGCGGCAGAUGGCAUCGGUACCCGCUACUGGCAUGAGUGUCAUU
..((((((.((.((((((...(((((.....)))))...)))))).))((.(((.((((.(((((.....((....)).....))))))))).)))))...))))))........... ( -46.80)
>consensus
GAUGCCAGAUGAUCAGUGCAGAAUGAAGGUCCCAAGAGA_G__GAUCACAUGGCUCUCUCUGCCCUGUGACGUCACUAGCAGAUGGCAUGGGUACCAGCU_CUGGCAGUUGGCAUCAAUG
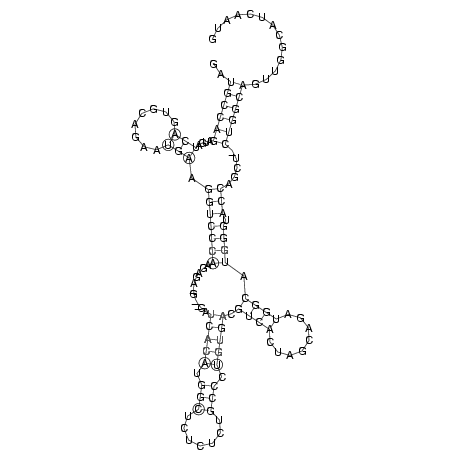
..((((((....(((........))).(((((((...........(((((.(((.......))).))))).((((........)))).)))).))).....))))))............. (-30.59 = -30.27 + -0.31)
124950.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004