Index >
Results for CNB 118451-rev
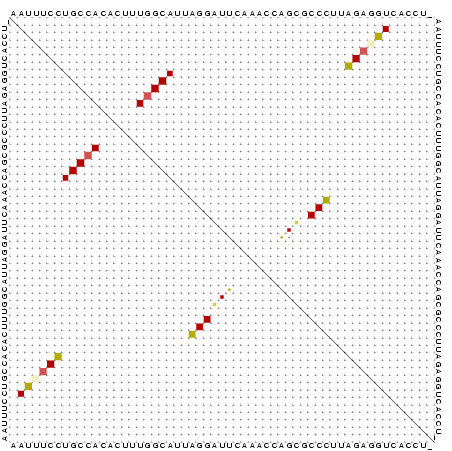
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
118451_SINFRUG00000145244_FUGU_2793_2847/1-57 -ATCTCCTGCCACACCTTGGCATTGGGGTTCAAACCGGCACCCTTGGAGGTGACCT-
118451_ENSMUSG00000046236_MOUSE_9818_9873/1-57 AATTTCTTGCCATACTTTGGCATTAGGATTTAAACCAGTGCCTTTAGATGTCACCT-
118451_ENSRNOG00000013146_RAT_4861_4917/1-57 AATTTCTTGCCATACTTTGGCATTAGGATTTAAACCAGTGCCTTTAGATGTCACGTG
118451_ENSDARG00000028567_ZEBRAFISH_8408_8464/1-57 CACTTCCTGCCACACTTTAGCATTGGGATTCAGACCAGCCCCCTTGGTGGTCACCTT
* ** ***** ** ** ***** ** ** * *** * ** ** * ** ** *
118451-rev.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 57
Mean pairwise identity: 77.42
Mean single sequence MFE: -13.57
Consensus MFE: -10.01
Energy contribution: -9.58
Covariance contribution: -0.44
Mean z-score: -1.57
Structure conservation index: 0.74
SVM decision value: 0.37
SVM RNA-class probability: 0.707521
Prediction: RNA
######################################################################
>118451_SINFRUG00000145244_FUGU_2793_2847/1-57
AUCUCCUGCCACACCUUGGCAUUGGGGUUCAAACCGGCACCCUUGGAGGUGACCU
(((((((((((.....)))))..(((((((.....)).))))).))))))..... ( -18.80)
>118451_ENSMUSG00000046236_MOUSE_9818_9873/1-57
AAUUUCUUGCCAUACUUUGGCAUUAGGAUUUAAACCAGUGCCUUUAGAUGUCACCU
.......((.(((.((..((((((.((.......))))))))...))))).))... ( -11.80)
>118451_ENSRNOG00000013146_RAT_4861_4917/1-57
AAUUUCUUGCCAUACUUUGGCAUUAGGAUUUAAACCAGUGCCUUUAGAUGUCACGUG
.......((.(((.((..((((((.((.......))))))))...))))).)).... ( -11.80)
>118451_ENSDARG00000028567_ZEBRAFISH_8408_8464/1-57
CACUUCCUGCCACACUUUAGCAUUGGGAUUCAGACCAGCCCCCUUGGUGGUCACCUU
...(((((((.........)))..))))....(((((.((.....)))))))..... ( -11.90)
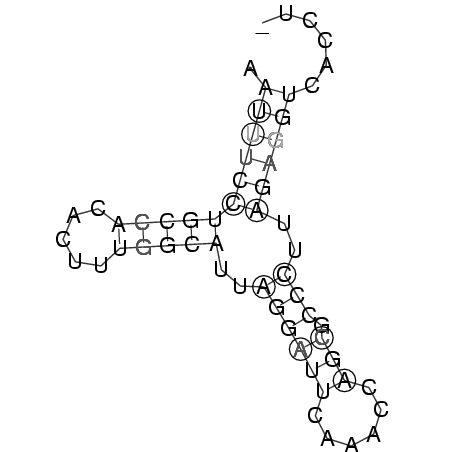
>consensus
AAUUUCCUGCCACACUUUGGCAUUAGGAUUCAAACCAGCGCCCUUAGAGGUCACCU_
.(((((((((((.....)))))..((((((......))).)))..))))))...... (-10.01 = -9.58 + -0.44)
118451-rev.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004