Index >
Results for CNB 110345
Input Alignment
CLUSTAL W(1.81) multiple sequence alignment
110345_ENSG00000128191_HUMAN_9063_9149/1-87 AAGCTGAGTGCATTGTGATTTCCAATAATTGAGGCAGTGGTTCTAAAAGCTGTCTACATT
110345_ENSMUSG00000022718_MOUSE_6118_6204/1-87 AAGCTGAGTGCATTGTGATTTCCAATAATTGAGGCAGTGGTTCTAAAAGCTGTCTACATT
110345_ENSRNOG00000001886_RAT_9769_9855/1-87 AAGCTGAGTGCATTGTGATTTCCAATAATTGAGGCAGTGGTTCTAAAAGCTGTCTACATT
110345_SINFRUG00000153636_FUGU_1736_1820/1-87 -AGCTGAATGCATTGTGATTTCCAATAATTGAGACAGTGATTCTAAAAGCTGTCTACATT
****** ************************* ***** ********************
110345_ENSG00000128191_HUMAN_9063_9149/1-87 AATGAAAAGAGCAATGTGGCCAGCTTG
110345_ENSMUSG00000022718_MOUSE_6118_6204/1-87 AATGAAAAGAGCAATGTGGCCAGCTTG
110345_ENSRNOG00000001886_RAT_9769_9855/1-87 AATGAAAAGAGCAATGTGGCCAGCTTG
110345_SINFRUG00000153636_FUGU_1736_1820/1-87 AATGAAAAGAACAATGTAGTCAGCTT-
********** ****** * ******
//
110345.aln
RNAz output
############################ RNAz 0.1 ##############################
Sequences: 4
Columns: 87
Mean pairwise identity: 95.40
Mean single sequence MFE: -29.37
Consensus MFE: -28.99
Energy contribution: -28.67
Covariance contribution: -0.31
Mean z-score: -4.49
Structure conservation index: 0.99
SVM decision value: 1.98
SVM RNA-class probability: 0.984670
Prediction: RNA
######################################################################
>110345_ENSG00000128191_HUMAN_9063_9149/1-87
AAGCUGAGUGCAUUGUGAUUUCCAAUAAUUGAGGCAGUGGUUCUAAAAGCUGUCUACAUUAAUGAAAAGAGCAAUGUGGCCAGCUUG
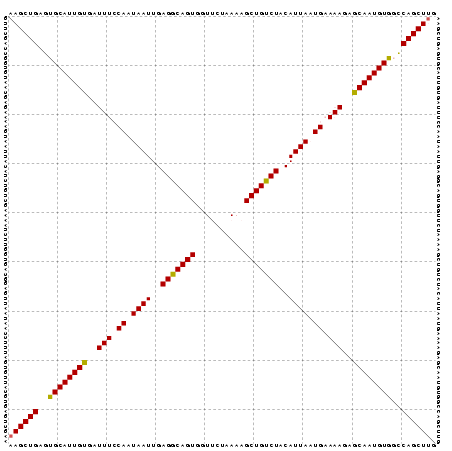
((((((..((((((((..(((.((.((((..(((((((..........)))))))..)))).)).)))..))))))))..)))))). ( -28.90)
>110345_ENSMUSG00000022718_MOUSE_6118_6204/1-87
AAGCUGAGUGCAUUGUGAUUUCCAAUAAUUGAGGCAGUGGUUCUAAAAGCUGUCUACAUUAAUGAAAAGAGCAAUGUGGCCAGCUUG
((((((..((((((((..(((.((.((((..(((((((..........)))))))..)))).)).)))..))))))))..)))))). ( -28.90)
>110345_ENSRNOG00000001886_RAT_9769_9855/1-87
AAGCUGAGUGCAUUGUGAUUUCCAAUAAUUGAGGCAGUGGUUCUAAAAGCUGUCUACAUUAAUGAAAAGAGCAAUGUGGCCAGCUUG
((((((..((((((((..(((.((.((((..(((((((..........)))))))..)))).)).)))..))))))))..)))))). ( -28.90)
>110345_SINFRUG00000153636_FUGU_1736_1820/1-87
AGCUGAAUGCAUUGUGAUUUCCAAUAAUUGAGACAGUGAUUCUAAAAGCUGUCUACAUUAAUGAAAAGAACAAUGUAGUCAGCUU
((((((.((((((((..(((.((.((((..(((((((..........)))))))..)))).)).)))..)))))))).)))))). ( -30.80)
>consensus
AAGCUGAGUGCAUUGUGAUUUCCAAUAAUUGAGGCAGUGGUUCUAAAAGCUGUCUACAUUAAUGAAAAGAGCAAUGUGGCCAGCUUG
((((((..((((((((..(((.((.((((..(((((((..........)))))))..)))).)).)))..))))))))..)))))). (-28.99 = -28.67 + -0.31)
110345.rnaz
RNAalifold consensus structure
Stefan Washietl
Last modified: Thu Aug 12 14:35:44 CEST 2004