Pseudoknoten
pknots
Das pknots-Programm der Universitaet Bielefeld faltet auf Grundlage einer RNA-Sequenz eine Sekundaerstruktur unter Beruecksichtigung moeglicher einfacher, rekursiver Pseudoknoten. Hier wurde die Version pknotsRG-mfe verwendet, welche Strukturen unter Minimierung der freien Energie erstellt. Maximal 800 bp einer Sequenz koennen gefaltet werden. Das Programm faltete die gekuerzte Sequenz von Homo sapiens (links) ohne Pseudoknoten, waehrend bei der ungekuerzten 3'UTR-Sequenz ein Pseudoknoten (rechts, gelb markiert in folgender Abbildung) ausgebildet wurde. Die Aktivatorsequenz wird laut pknots als durch Osman et. al vorhergesagte Stem-Loop-Struktur gefaltet (umrahmt).
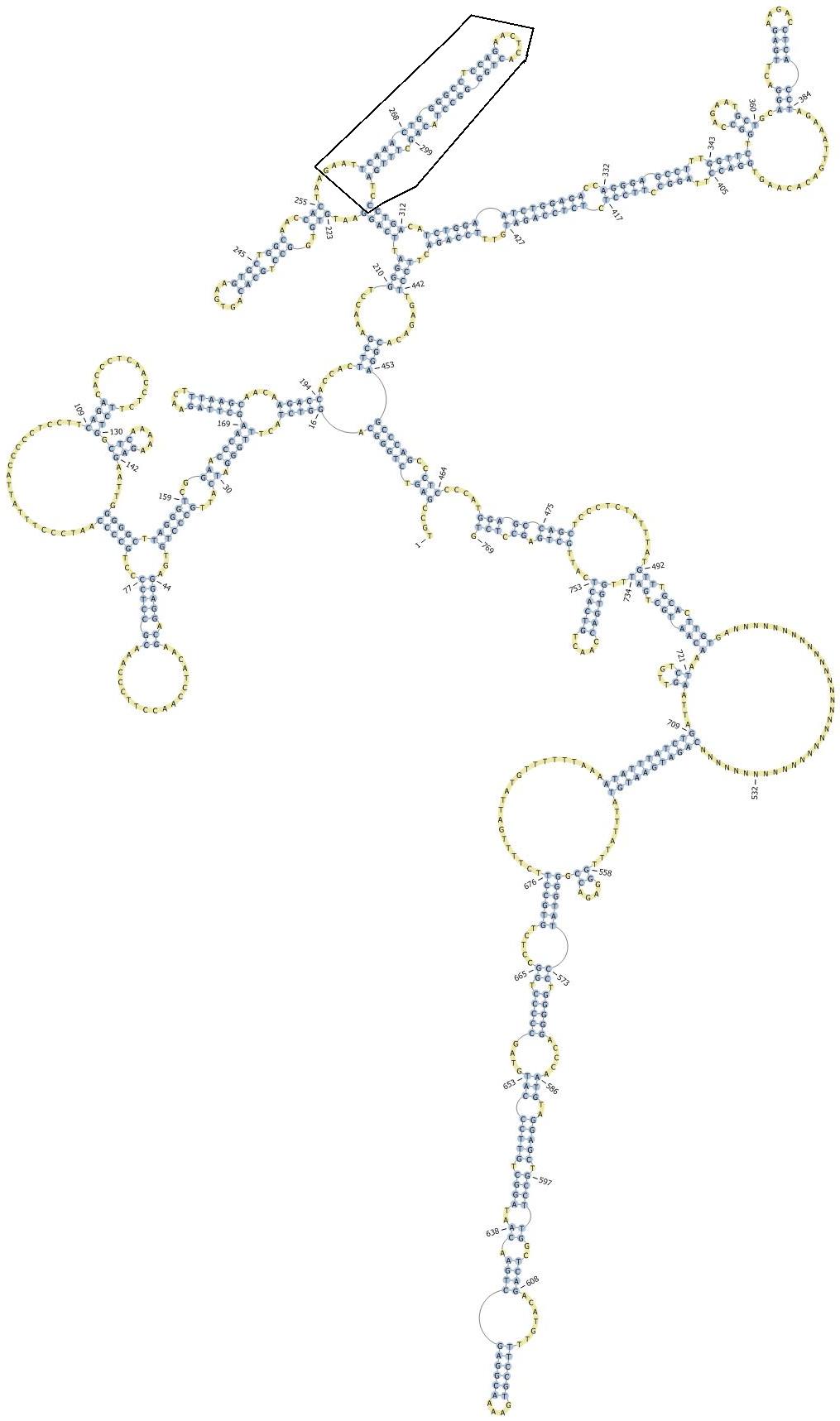
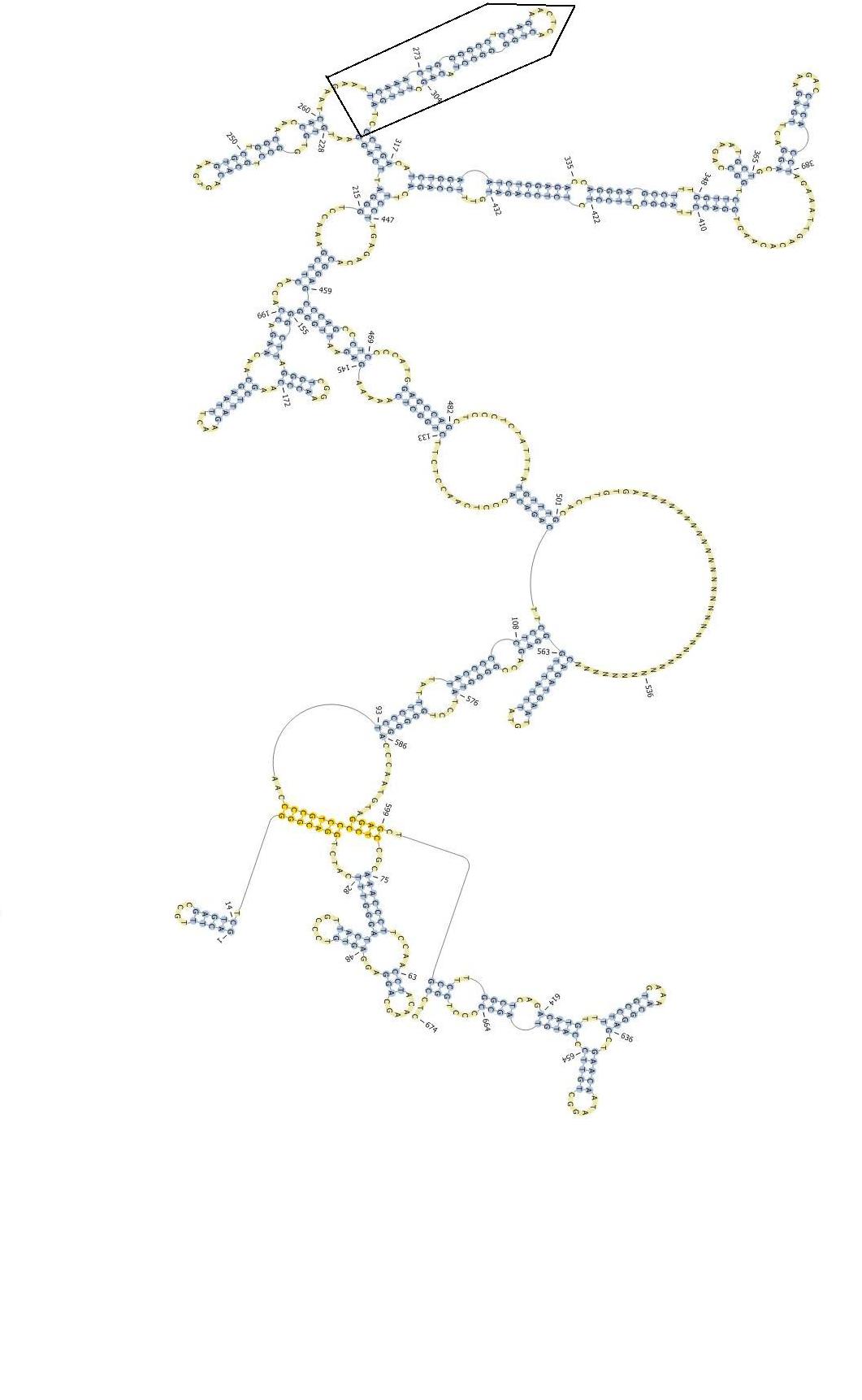
Zudem wurden die gekuezten UTR-Sequenzen der naechsten Verwandten von Homo sapiens Pan troglodytes und Gorilla gorilla mittels pknots gefaltet.
Bei beiden Arten paarten die Basen 1 bis 4 bzw. 7 bis 13 mit verschiedenen Basenabschnitten zu einem Pseudoknoten. Im Gegensatz dazu bildete der gleiche Sequenzausschnitt von Homo sapiens keinen Pseudoknoten aus. Die Sequenz von Felis catus wies bei Faltung 2 Pseudoknoten auf.
Andere Pseudoknoten-Programme
Andere verwendete Programme wie HotKnots und vsfold5 konnten maximal Sequenzlaengen von 100 bp bzw. 400 bp falten, sodass die oben gezeigten Stem-Loop-Strukturen nicht in gleicher Weise reproduziert werden konnten.
Da nach Osman et al. keine Pseudoknoten in der 3'UTR bekannt sind, gab es keinen konkreten Ansatz, bestimmte Sequenzabschnitte zu testen.
Somit wurde kein konservierter Pseudoknoten gefunden. Es kann jedoch nicht ausgeschlossen werden, dass Pseudoknoten in der 3'UTR existieren, da nicht alle moeglichen Sequenzabschnitte getestet wurden und die Pseudoknoten-Programme aufgrund unterschiedlicher Sequenzlaengen unterschiedliche Ergebnisse liefern.
Startseite