Ergebnisse
Ergebnisse
1. Chrakteristik des
Datensatzes
2. Annotation mit
Datenbanken, RNAmicro und tRNAScan
3. Cluster-Analyse auf
Sequenzebene
4. Cluster-Analyse
hinsichtlich der Sekundaerstruktur
5. Manuelle Annotation
der Ergebnissen der Clusteranalyse
6. Sekundärstrukturen
1. Chrakteristik des Datensatzes
Locus-Laengen
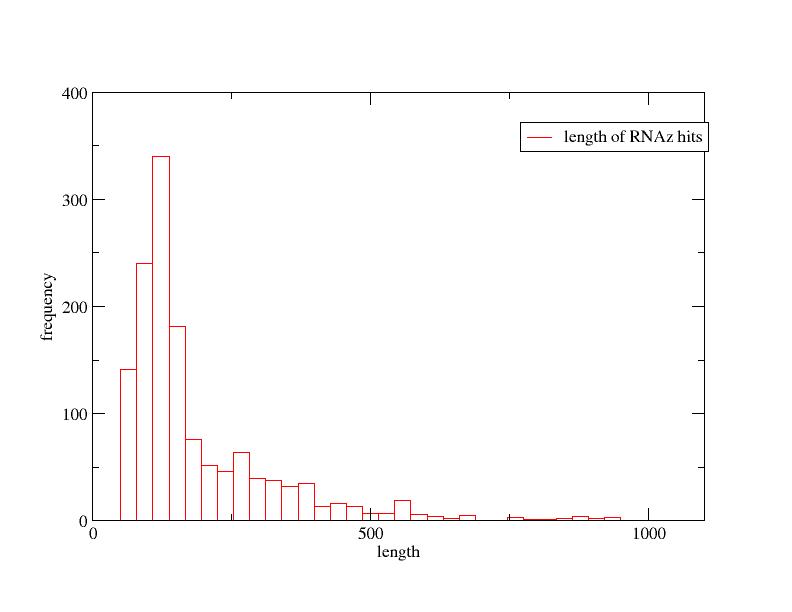
Die Haeufung der bei 120 ist mit der Windowslaenge von eben diesem Wert zu erklaeren.
P-value fuer die Originaldaten und den zufaellig erzeugten
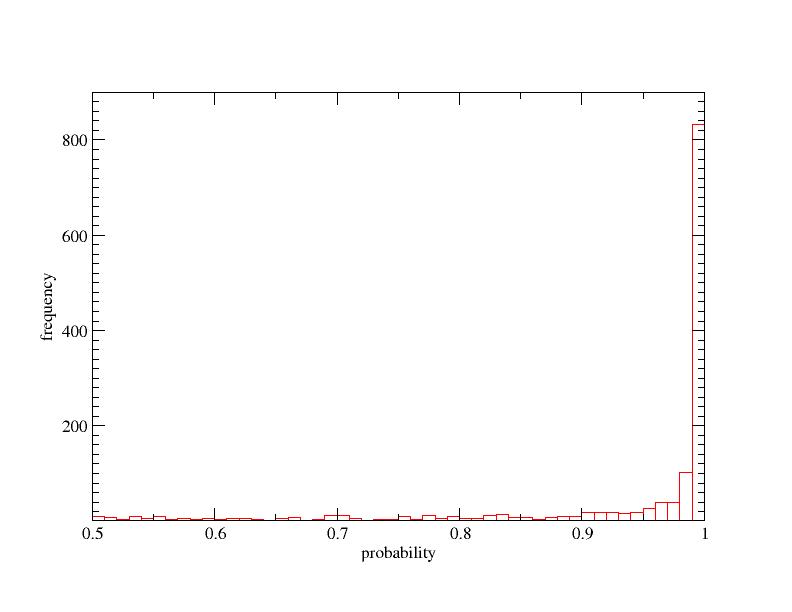
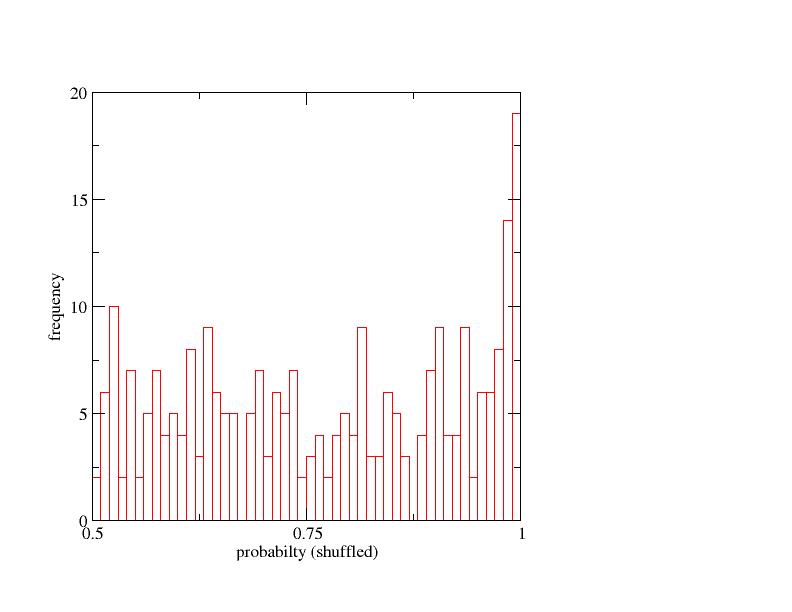
Im Originaldatensatz werden sehr viele Kandidaten mit hoher Signifikanz gefunden, waehrend im zufaellig erzeugten zum einen insgesamt weniger als ncRNA eingestuft werden und ausserdem werden den gefunden Kandidaten geringere Signifikanzen zugewiesen.
SCI und z-score im Vergleich mit den zufaellig erzeugten Daten
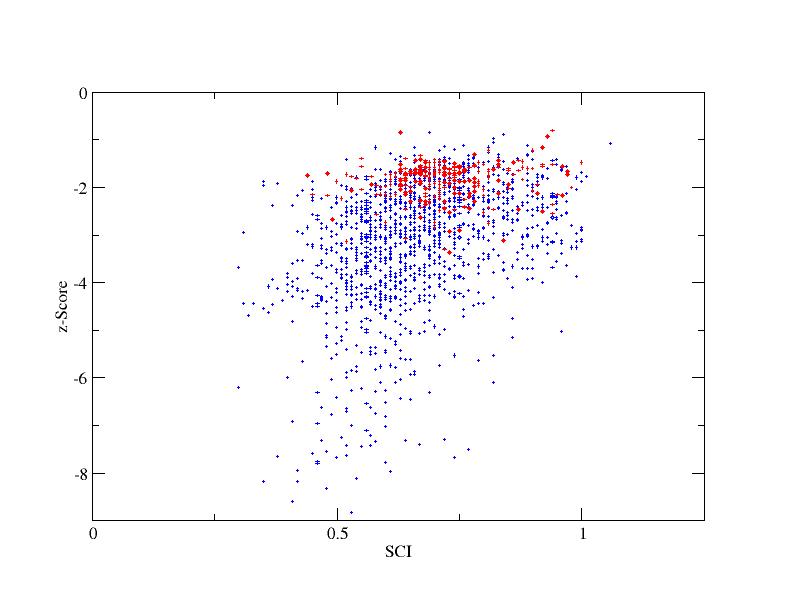
blau: Orginial-loci
rot: zufaellig gemischten loci
Loci mit einem z-Score mit einem unter -3 und einem SCI-Werte nahe 1 gelten als gute ncRNA-Kandidaten. Unter den Orginalloci erfuellen sichtbar mehr loci die Anforderungen.
False-Positive-Rate
False-Positive-Rate = 268/1392 = 0.1925
Erwartungsgemaess faellt die FPRate einigemassen gering aus, was darauf hindeutet, dass RNAz relativ gut trainiert ist und aus einem in Rahmen zufaellig erzeugten Datensatz
erheblich weniger ncRNAs vorhersagt.
2. Annotation mit Datenbanken, RNAmicro und tRNAScan
Der Vergleich mit den Datenbanken ergab folgende Ergebnisse:
noncode 3 hits
Rfam 120 hits
miRBase 9 hits
ncRNAdb 27 hits
tRNAScan 110 hits
RNAmicro 305 hits
gesamt annotiert: 426
Anzahl aller loci: 1392
nicht-annotierte mit p-wert > .9: 724
nicht-annotierte mit p-wert > .99: 459
3. Clusteranalyse auf Sequenzebene
1. Cluster-size:63; Anotated:58; RNAmicro:58;
2. Cluster-size:32; Anotated:31; RNAmicro:31;
3. Cluster-size:26; Anotated:17; RNAmicro:17;
4. Cluster-size:25; Anotated:0;
5. Cluster-size:24; Anotated:0;
6. Cluster-size:21; Anotated:17; RNAmicro:17;
7. Cluster-size:21; Anotated:2; RNAmicro:2;
8. Cluster-size:21; Anotated:18; RNAmicro:18;
9. Cluster-size:21; Anotated:1; RNAmicro:1;
10. Cluster-size:19; Anotated:0;
11. Cluster-size:18; Anotated:2; RNAmicro:2;
12. Cluster-size:17; Anotated:0;
13. Cluster-size:14; Anotated:0;
14. Cluster-size:13; Anotated:7; RNAmicro:7;
15. Cluster-size:13; Anotated:8; RNAmicro:8;
16. Cluster-size:12; Anotated:2; RNAmicro:2;
17. Cluster-size:12; Anotated:0;
18. Cluster-size:12; Anotated:12; Rfam:12;tRNAScan:12;
19. Cluster-size:11; Anotated:0;
20. Cluster-size:11; Anotated:11; Rfam:11;tRNAScan:11;
...
Verteilung der Loci selbst, sowie der fuenf groessten Cluster im untersuchten Genomabschnitt
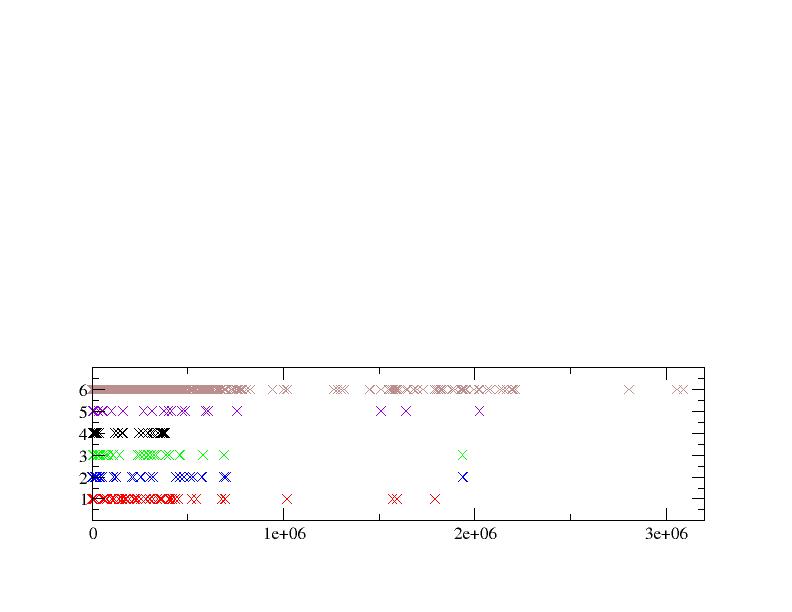
1: Cluster Nr. 1 (s.o.)
2: Cluster Nr. 2 (s.o.)
3: Cluster Nr. 3 (s.o.)
4: Cluster Nr. 4 (s.o.)
5: Cluster Nr. 5 (s.o.)
6: alle Kandidaten (1392 loci)
Deutlich wird mit diese Grafik, dass sich im Allgemeinen die RNAz-Kandidaten eher im vorderen Bereich des Genoms befinden und somit auch in diesem Bereich viele Cluster sich bilden koennen.
5. Manuelle Annotation der Ergebnissen der Clusteranalyse
| Locus | Annotation | S-Score | e-Value |
|---|---|---|---|
| locus1315 | Non-LTR Retrotransposon | 216 | e-53 |
| locus138 | Non-LTR Retrotransposon | 218 | e-54 |
| locus39 | Non-LTR Retrotransposon | 222 | e-55 |
| Fugu T-cell-receptor | 50 | 0.002 | |
| locus437 | Non-LTR Retrotransposon | 222 | e-55 |
| Fugu T-cell-receptor | 50 | 0.001 | |
| locus1002 | Fugu T-cell-receptor | 234 | e-59 |
| Non-LTR Retrotransposon | 46 | 0.03 | |
| locus676 | Fugu T-cell-receptor | 224 | e-56 |
| Non-LTR Retrotransposon | 54 | e-04 |
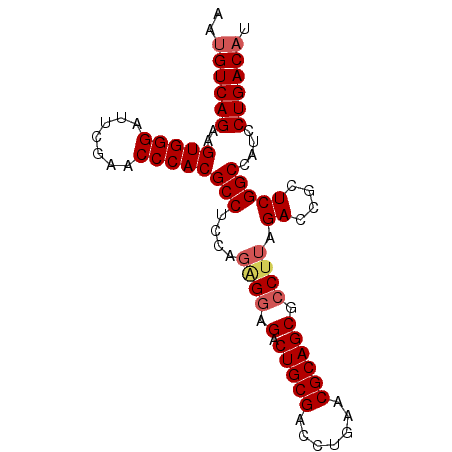
6. Sekundärstrukturen
Mikro-RNA

t-RNA