4. Auswertung
Einleitend ist zu sagen, daß die Untersuchung auf konservierte Sekundärstrukturen
relativ ergebnislos verlief. Um eine gewisse evolutionäre Distanz zwischen den Sequenzen zu
gewährleisten, entschlossen wir uns die Gruppen
untereinander zu vergleichen.
Begonnen wurde mit einem Vergleich zwischen allen Gruppen:
| Anzahl der Sequenzen |
11 |
| minimale Sequenzlänge |
7434 bp |
| maximale Sequenzlänge |
9895 bp |
| Alignmentlänge |
10329 bp |
| Durchschnittliche Sequenzähnlichkeit |
49% |
|
|
|
|
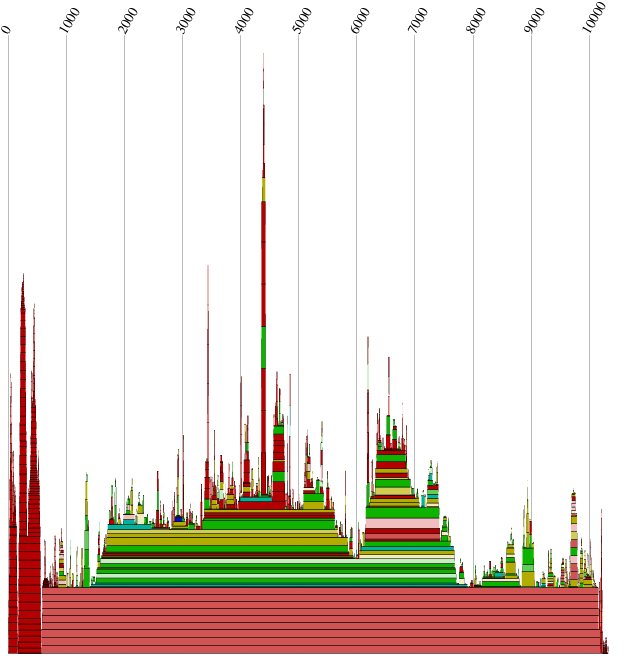
Abb.8: der Mountainplot aller Gruppen
|
Zwischen den Basenpaaren 4000 und 5000 sind mehrere Peaks zu erkennen, die eine interessante
Sekundärstruktur vermuten lassen. Aber selbst das beste Ergebnis:
|
|
|
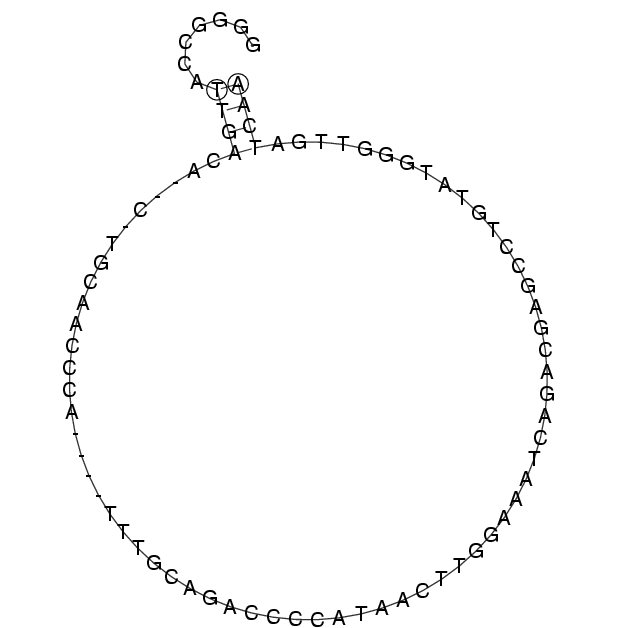
Abb.9: vorhergesagte Sekundärstruktur
für den Bereich von 4350 bis 4430
|
läßt erkennen, daß in diesem Bereich die Erwartungen nicht erfüllt werden.
Die folgenden Untersuchungen konzentrierten sich auf den Vergleich zwischen Gruppe 1 und 2. Hier sind
die Längenunterschiede zwischen den Sequenzen nicht mehr ganz so groß (maximal 1160 bp).
| Anzahl der Sequenzen |
7 |
| minimale Sequenzlänge |
7434 bp |
| maximale Sequenzlänge |
8557 bp |
| Alignmentlänge |
8864 bp |
| Durchschnittliche Sequenzähnlichkeit |
43% |
|
|
|
|
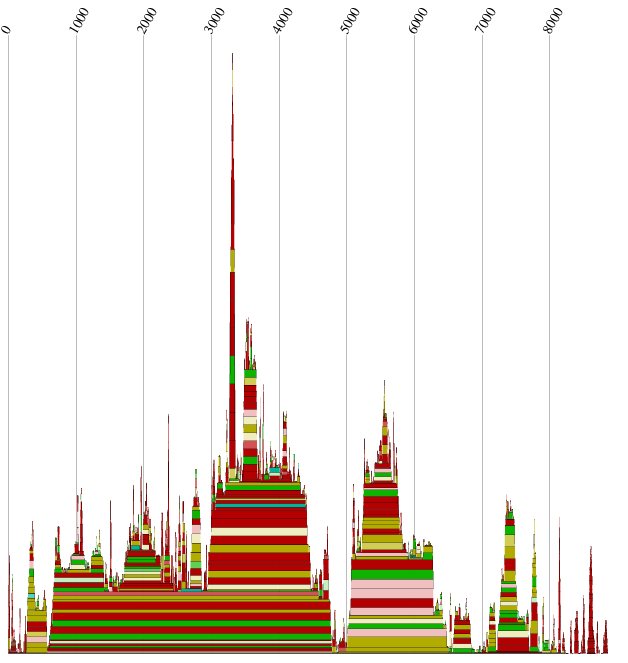
Abb.10: der Mountainplot der Gruppen 1 & 2
|
Die Untersuchung der einzelnen Sequenzintervalle ergab folgende Sekundärstrukturen:
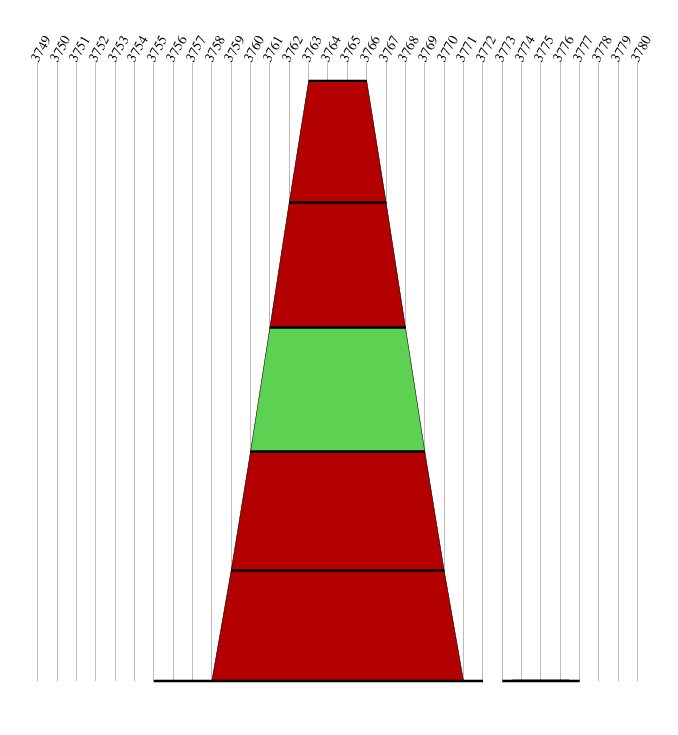
Bereich von 3750 bis 3780
|
|
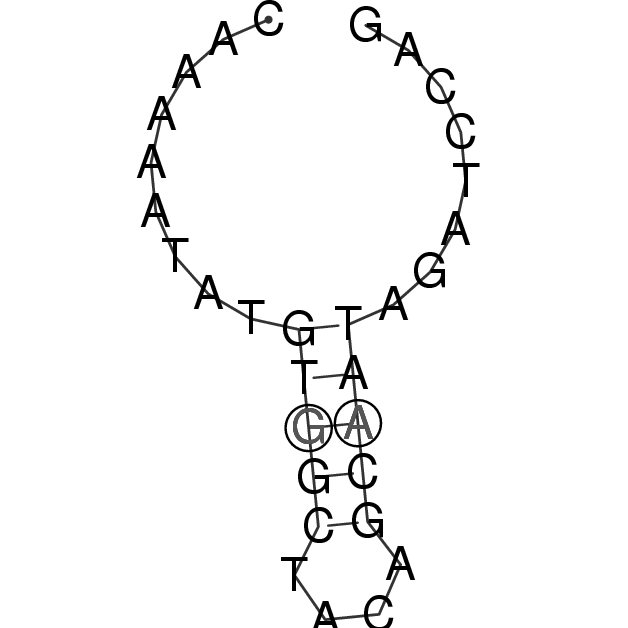
|
|
|
|
Abb.11: Bereich von 3750 bis 3780 aus den Gruppen 1 & 2
|
|
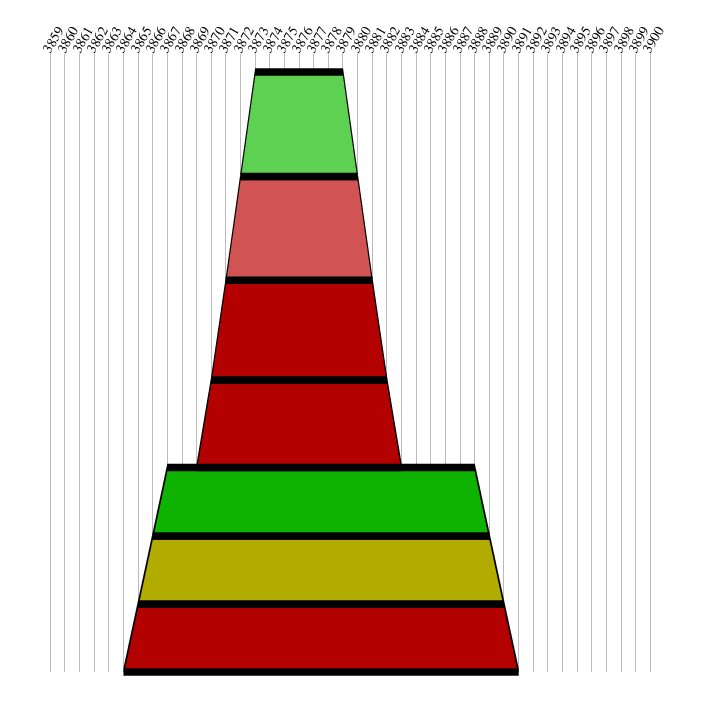
Bereich von 3860 bis 3900
|
|
|
|
|
|
Abb.12: Bereich von 3860 bis 3900 aus den Gruppen 1 & 2
|
|
Bereich von 7830 bis 7850
|
|
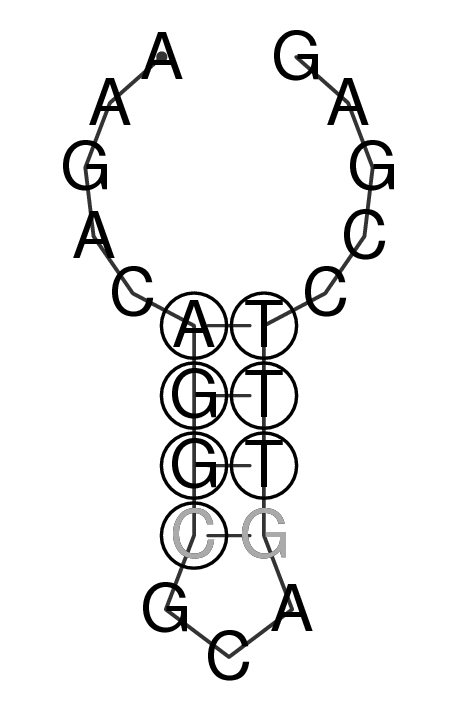
|
|

|
|
Abb.13: Bereich von 7830 bis 7850 aus den Gruppen 1 & 2
|
|
Gruppe 1
| Anzahl der Sequenzen |
3 |
| minimale Sequenzlänge |
7434 bp |
| maximale Sequenzlänge |
7462 bp |
| Alignmentlänge |
7470 bp |
| Durchschnittliche Sequenzähnlichkeit |
87% |
|
|
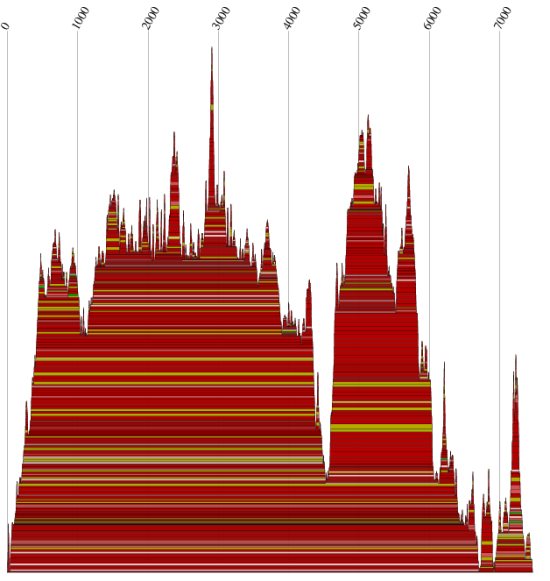
|
|
Abb.14: der Mountainplot der Gruppe 1
|
Gruppe 2
| Anzahl der Sequenzen |
4 |
| minimale Sequenzlänge |
7759 bp |
| maximale Sequenzlänge |
8557 bp |
| Alignmentlänge |
8812 bp |
| Durchschnittliche Sequenzähnlichkeit |
81% |
|
|
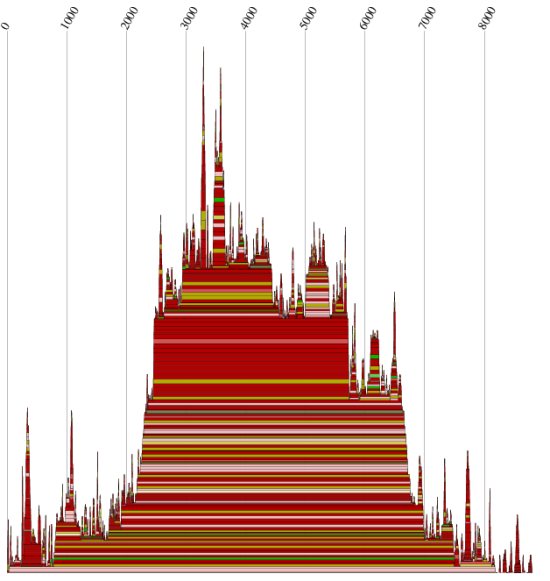
|
|
Abb.15: der Mountainplot der Gruppe 2
|
Im Gegensatz zu den Gruppen 1 und 3 läßt die Färbung der Gruppe 2 einige konservierte Sekundärstrukturen
vermuten. Der linke große "Peak" im Bereich 3000 bis 4000 des Mountainplots repräsentiert die folgende
Sekundürstruktur:
Bereich von 3240 bis 3330
|
|

|
|
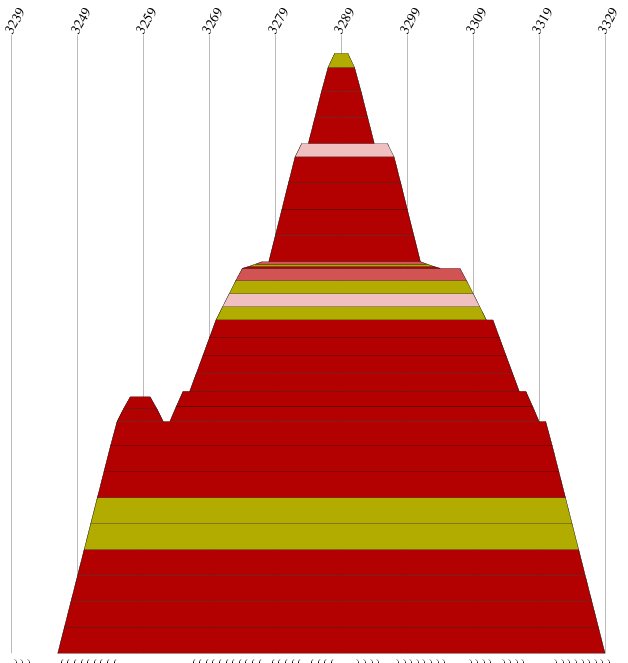
|
|
Abb.16: Bereich von 3240 bis 3330 aus den Gruppen 2
|
|
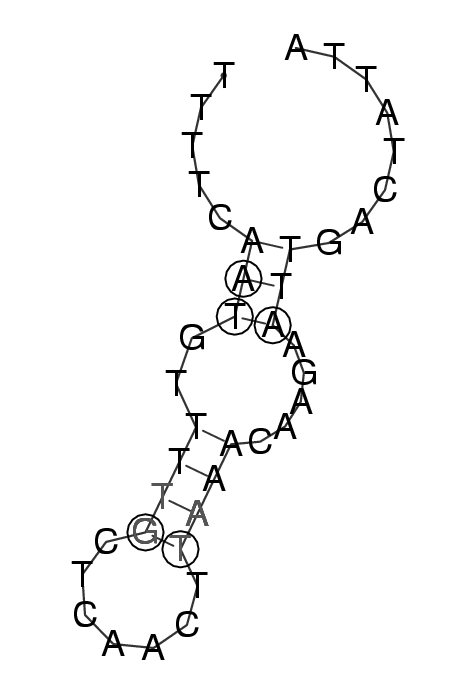
Die Struktur verfügt über eine geringe Anzahl konservierte Basenpaare. Es finden sich auch wenige instabile (hellgrau
markiert) Basenpaarungen, die aber die Struktur aufgrund ihrer Länge nicht destabilisieren.
Gruppe 3
Der Austausch der Sequenzen AF033807 mit der Sequenz M15122 erbrachte nicht die erhoffte Verbesserung.
Mit AF033807:
| Anzahl der Sequenzen |
4 |
| minimale Sequenzlänge |
8603 bp |
| maximale Sequenzlänge |
9895 bp |
| Alignmentlänge |
10001 bp |
| Durchschnittliche Sequenzähnlichkeit |
93% |
|
|
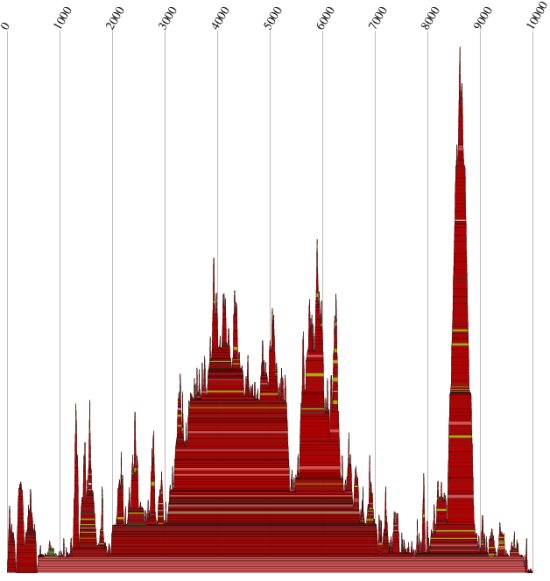
|
|
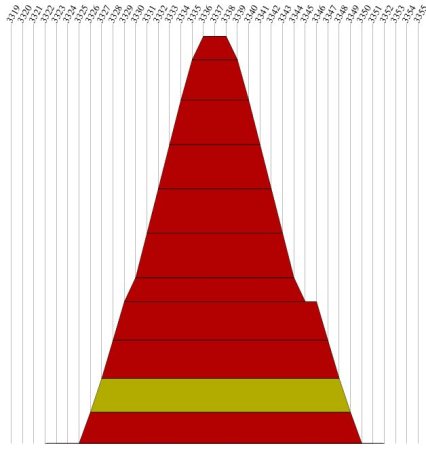
Abb.17: der Mountainplot der Gruppe 3
|
Aufgrund seiner starken Rotfärbung läßt der Mountainplot der Gruppe 3 auf keine große Anzahl konservierter
Sekundärstrukturen hoffen. Dies hat seine Ursache in der extrem hohen Ähnlichkeit (93%) der verglichenen Sequenzen. Aus
diesem Grund ist die Gruppe 3 im Rahmen der gestellten Praktikumsaufgabe ungeeignet.
Trotzdem hier eine kleine Auswahl an interessanten Strukturen:
Bereich von 8300 bis 8900
|
|

|
|
Abb.18: Bereich von 8300 bis 8900
aus der Gruppe 3
|
|
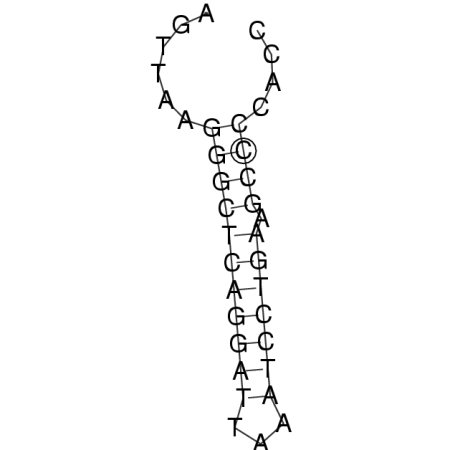
Bereich von 3320 bis 3355
|
|
|
|
|
|
Abb.19: Bereich von 3320 bis 3355 aus der Gruppe 3
|
|






