Praktikum Bioinformatik 1 - Auswertung der Analysen
4. Analyse und Auswertung der Untersuchungen
Analyse der Daten aus den ClustAlW-Alignments
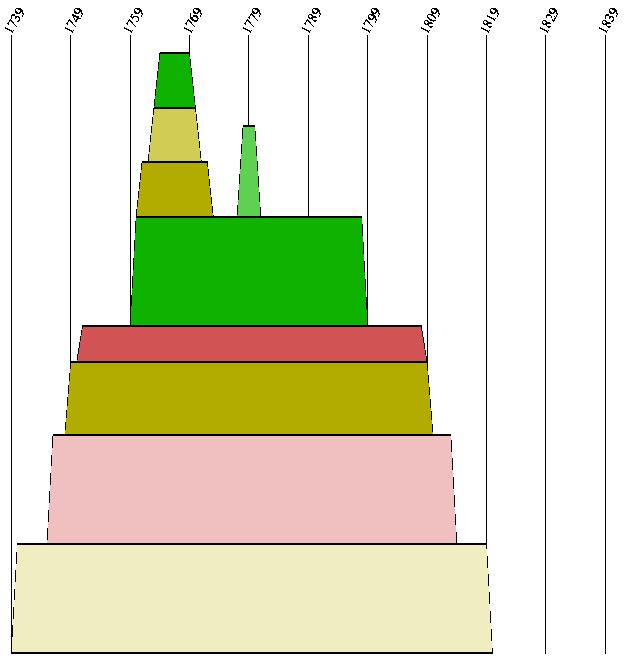
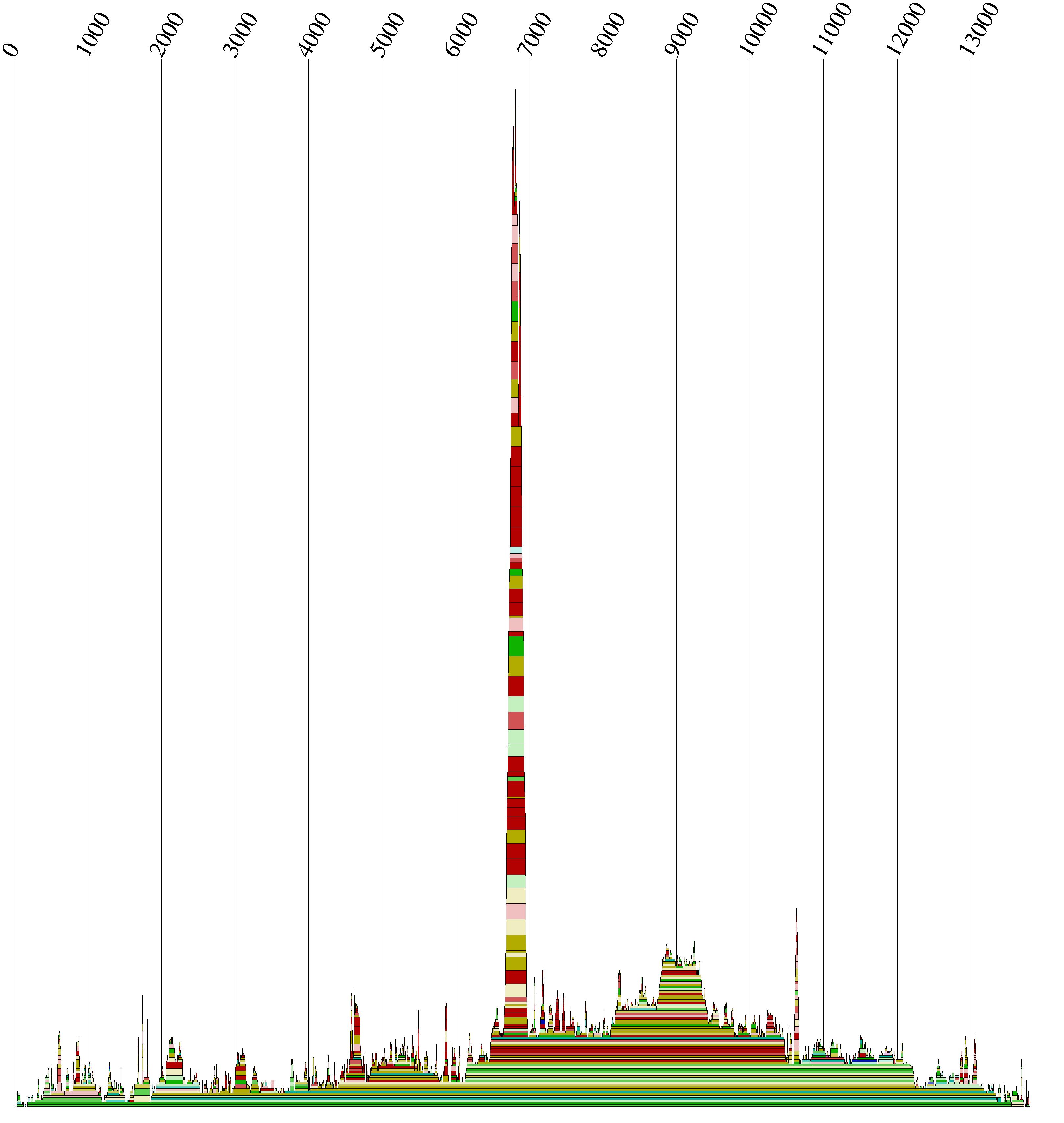
Die Sekundärstruktur aus den clustalw-Alignments in Mountain-Plot Darstellung:
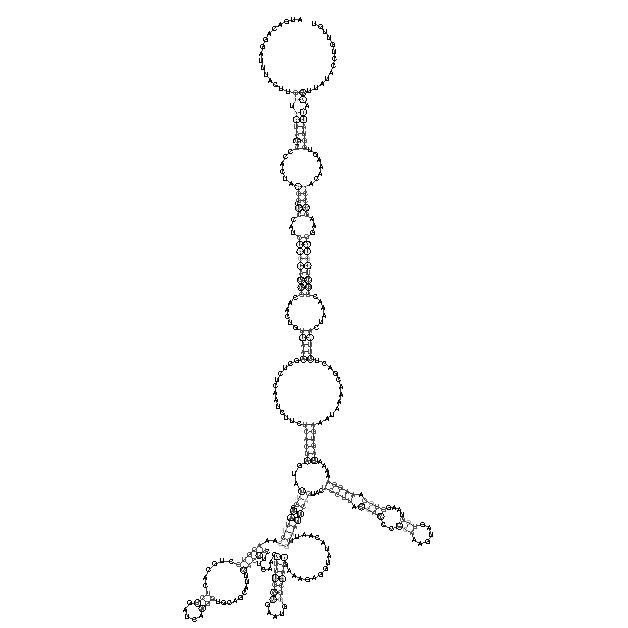
Sequenzabschnitt : 1770 - 1840
Gut zu erkennen sind die Haarnadelstrukturen und die stems. An den stems sind konservierte und kompensatorische Mutationen vorhanden, welche leider wenig aussagekräftig sind, da sie zu schwach ausgeprägt sind (zu graue Farbwerte).
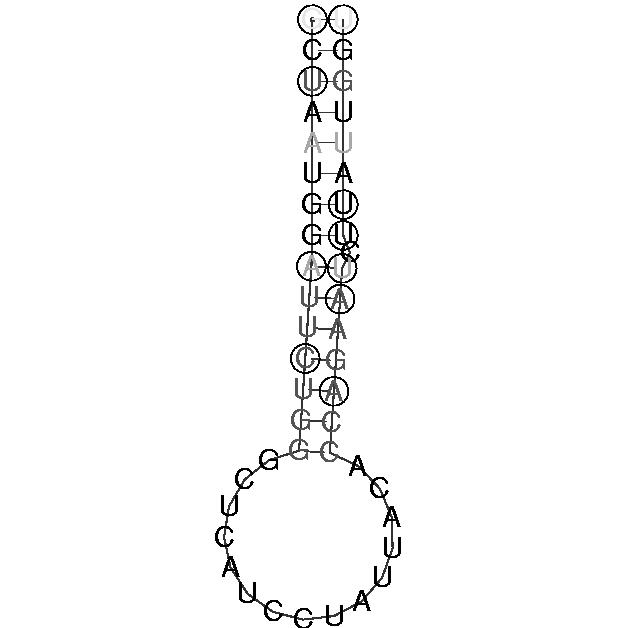
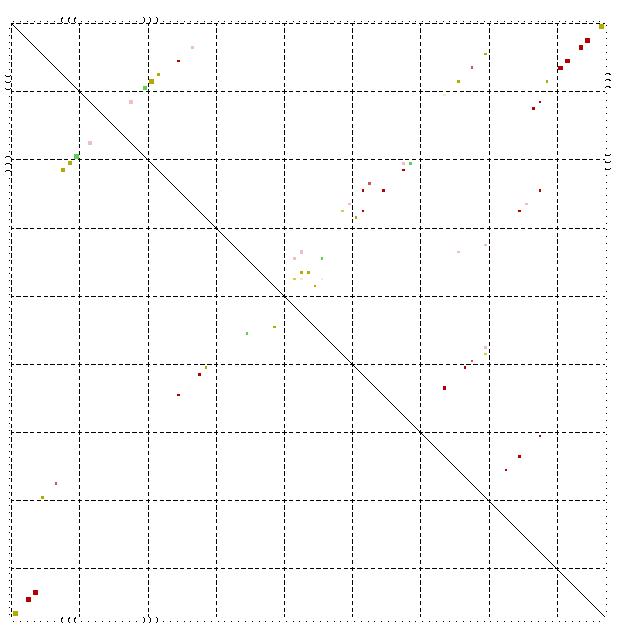
Sequenzabschnitt : 4708 - 4753
 |
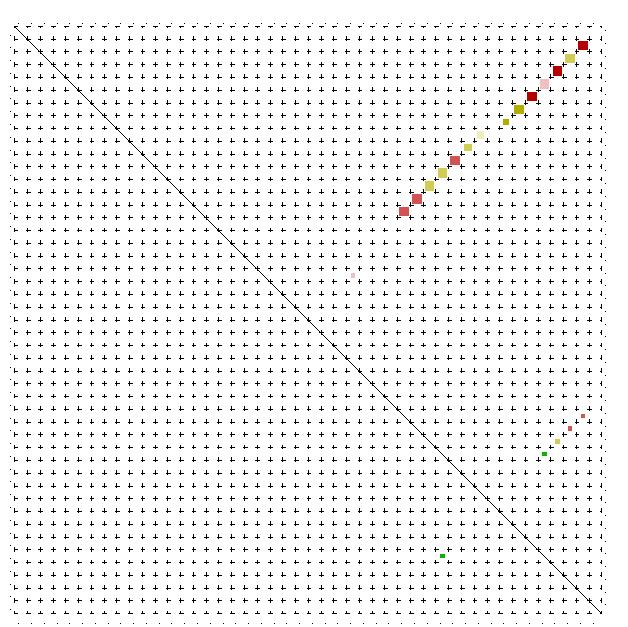
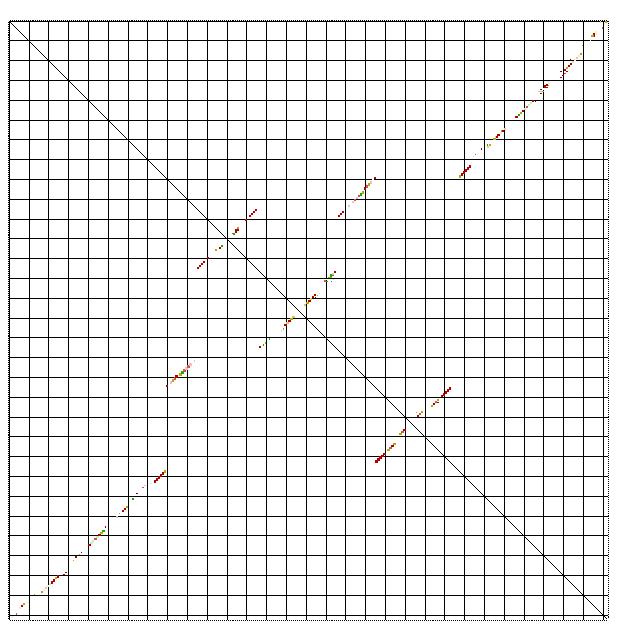
Der Bereich 4500 - 4800, in dem der betrachtete Bereich 4708 - 4753 eingebettet ist, weist einige Stellen auf, an denen
alidot konservierte Abschnitte vorhersagt.
|
Auch hier ist der stem mit dem anschließenden loop schön ausgeprägt. Jedoch tritt das selbe Problem wie beim Sequenzabschnitt 1770 - 1840 auf; die erkennbaren konservierten und kompensatorischen Abschnitte sind zu schwach ausgeprägt.
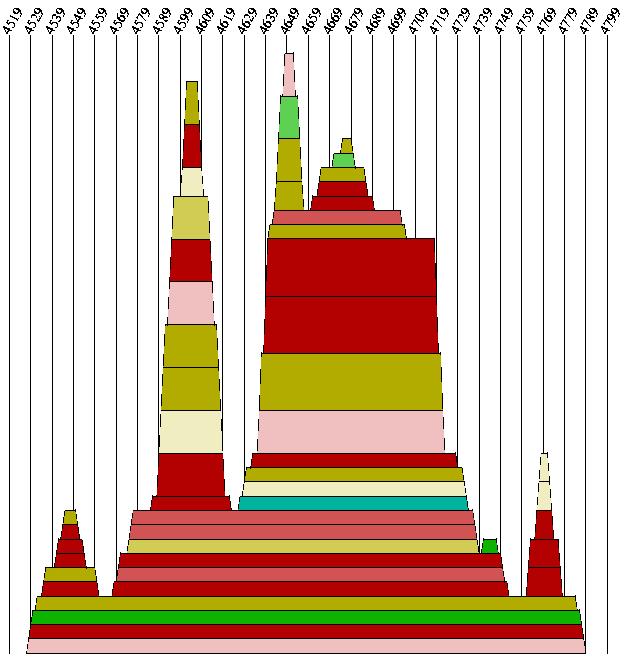
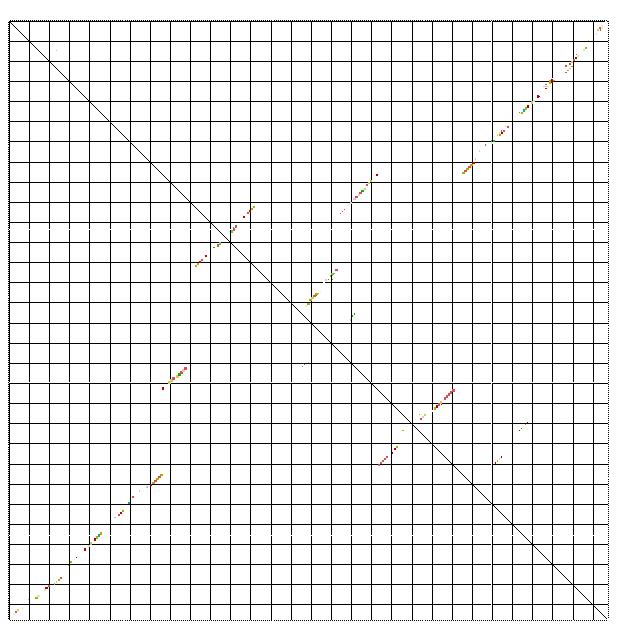
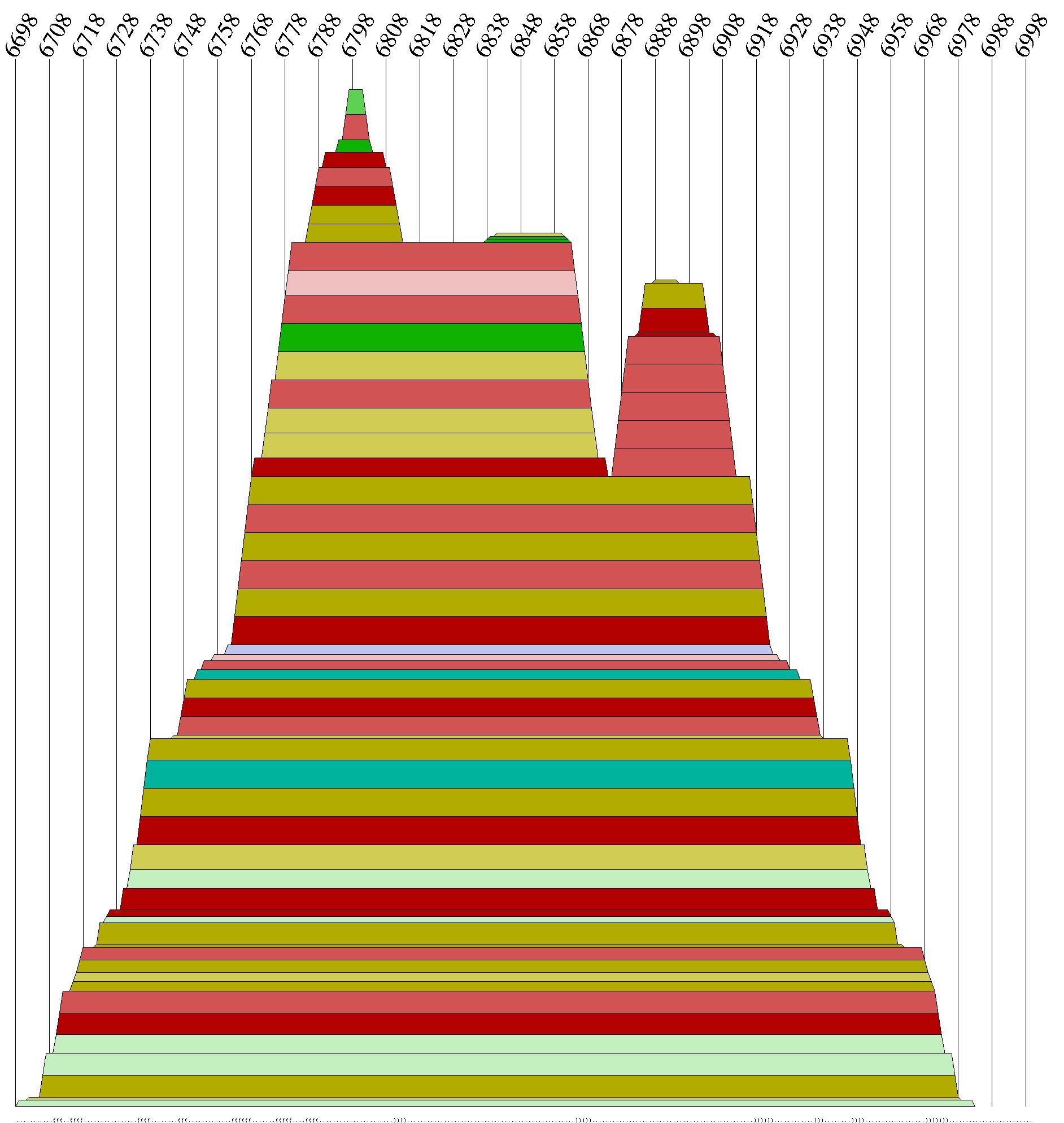
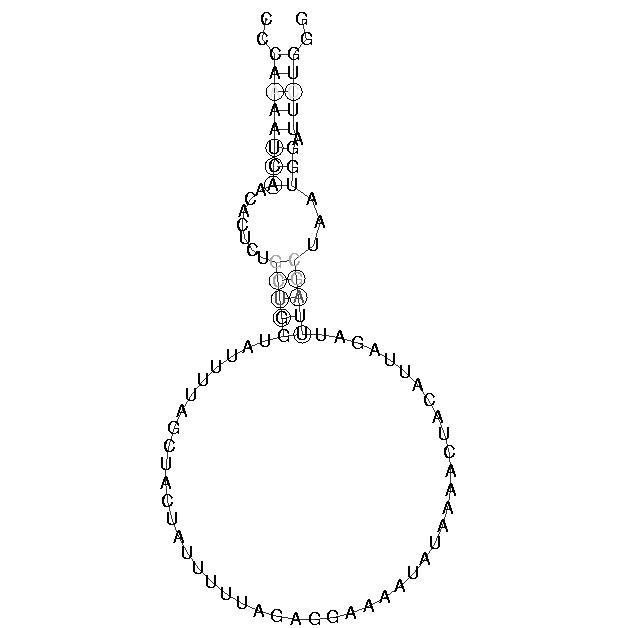
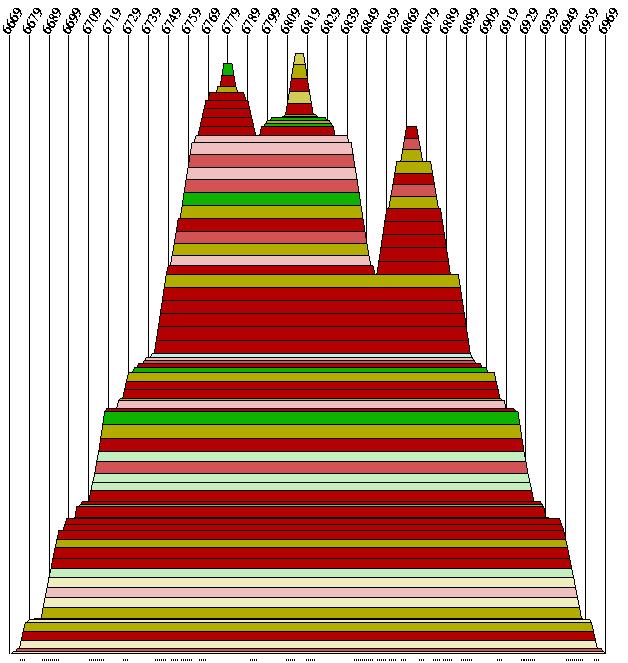
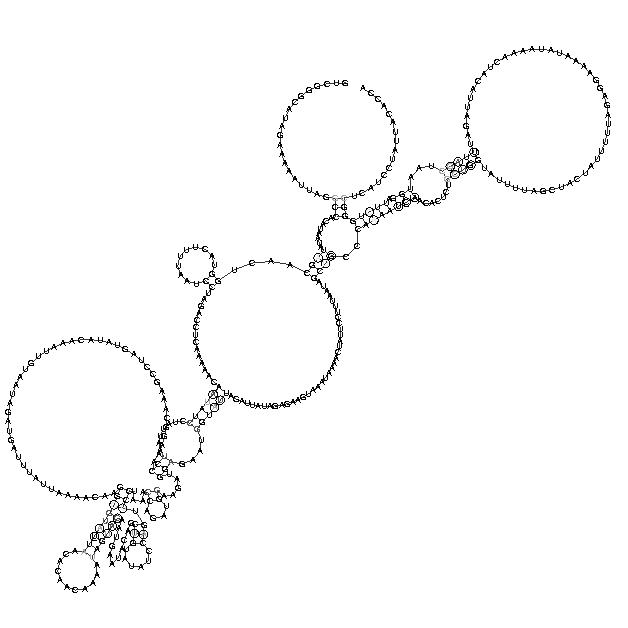
Sequenzabschnitt : 6693 - 6989
In diesem Abschnitt ist eine schöne Sekundärstruktur mit schönen stems und loops zu sehen. Leider sind auch hier wieder die konsistenten und kompensatorischen Mutationen zu schwach ausgeprägt.
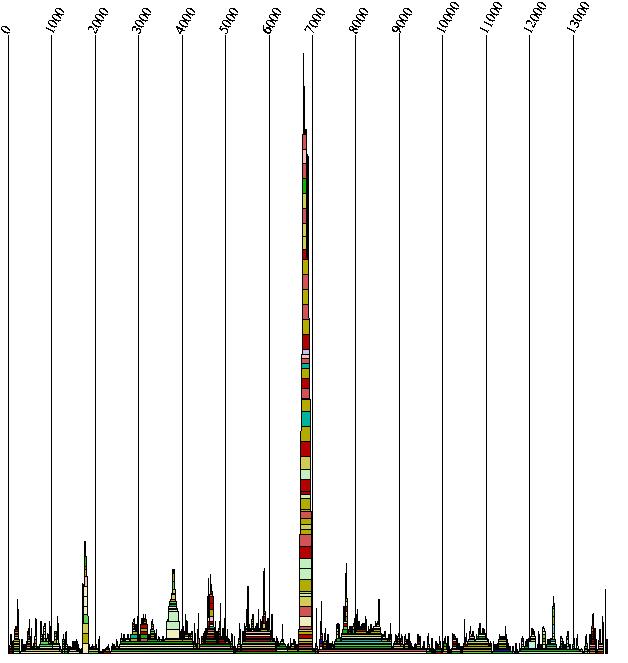
Zu diesem Sequenzabschnitt ist noch festzuhalten, dass es sich um die am meisten gefaltete Region im multiplen
Alignment handelt. Diese Region zeichnet sich durch einen auffallend hohen Peak im Mountain-Plot aus. Diese Region befindet in einem
Abschnitt der Nukeotidsequenz, die im Bereich des env – Gens liegt.
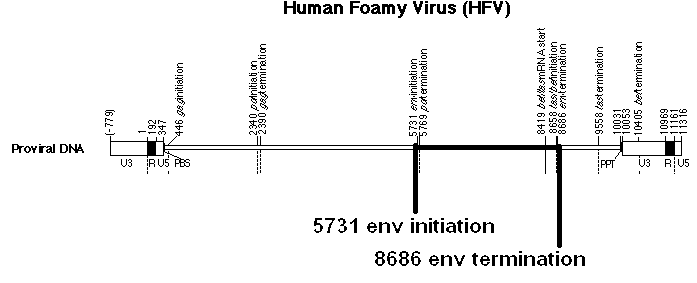
Es scheint so, als ob dieser Bereich der viralen RNA mit seiner starken Faltung bestimmte Aufgaben im Lebenzyklus der Spumaviren
übernimmt.
Analyse der Daten aus den code2aln-Alignments
Bei beiden unten abgebildeten Strukturten ist das selbe Problem wie bei den ClustalW - Auswertungen zu sehen - die konservierten und kompensatorischen Mutationen sind nicht stark genug ausgeprägt.
Darstellung des Mountain-Plots mit den Alignments aus code2aln :
Sequenzabschnitt : 4618 - 4704
Sequenzabschnitt : 6670 - 6971
Bei diesem Abschnitt handelt es sich um den auffallend großen Peak in der Mountain-Plot-Darstellung. Dabei ist zu erwähnen,
dass sich bei beiden Alignments ( code2aln vs. clustalw ) an dieser Stelle diese Peaks ergeben.
Zurück