Das zu untersuchende CASP5 Target T0157 beschreibt das Protein yqgF des Bakteriums E. coli und besitzt eine Länge von 138 Aminosären.
genutzte Server: blast
Es werden keine signifikanten Domainen in der Blast-Datenbank gefunden. Somit erübrigt sich auch die Suche nach Sequenz-Sequenz Homologen.
genutzte Software: Fugue
Es werden 3 signifikante Alignments gefunden.
genutzte Software: Modeller 6v2
Die für den Modeller benötigten Eingabedaten
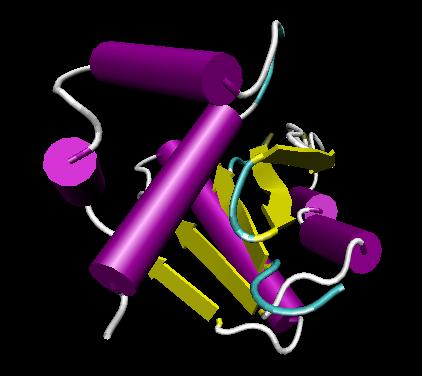
Kontrolle des erhaltenen Ergebnisses auf seine Glaubwürdigkeit:
genutzte Software: ProFit v2.2
Der Vergleich der Aminosäuren 6 bis 143 der Modellstruktur 1HJR (pdb-file) mit der Targetstruktur T0157 ergibt einen RMS-Wert von 11.46. Da der RMS-Wert grösser als der Grenzwert 10 ist, wird dieser Modellansatz wieder verworfen.
genutzte Software: 3dpssm
Es werden 2 signifikante Alignments gefunden.
Nachdem mit Blast kein signifikantes Sequenzalignment gefunden wurde, ergaben die Strukturalignments sowohl mit Fugue als auch mit 3dpssm die grösste Übereinstimmung mit 1HJR. Dieses Modell erwies sich aber als ungenügend für die Strukturvorhersage der Targets T0157.