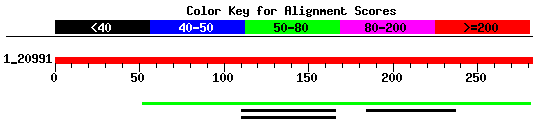
Das zu untersuchende CASP5 Target T0142 beschreibt das Protein Nitrophorin des Organismus Cimex lectularius und besitzt eine Länge von 282 Aminosären.
genutzte Server: blast
3 vermeintlich konservierte Domainen wurden gefunden:

genutzte Server: blastp
Die Nutzung der Domaine IPPc erzeugt 4 signifikante Alignments.
genutzte Software: Modeller 6v2
Die für den Modeller benötigten Eingabedaten
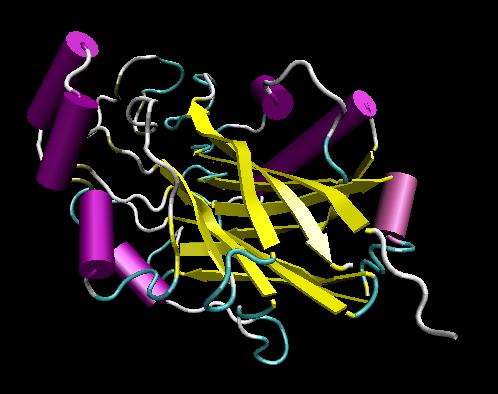
Kontrolle des erhaltenen Ergebnisses auf seine Glaubwürdigkeit:
genutzte Software: ProFit v2.2
Der Vergleich der Aminosären 552 bis 838 der Modellstruktur 1I9Y (pdb-file) mit der Targetstruktur T0142 ergibt einen RMS-Wert von 19.512. Dieser ist bedeutend grösser als der Grenzwert 10, weswegen auf ein schlechtes Modell zu schliessen ist.
Das Modell 1I9Y wird verworfen.
genutzte Software: 3dpssm
Es werden 2 signifikante Alignments gefunden.
genutzte Software: Modeller 6v2
Die für den Modeller benötigten Eingabedaten
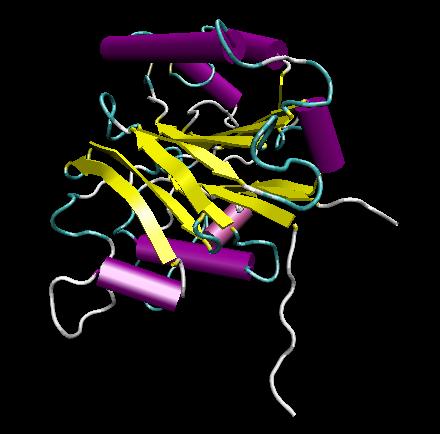
Kontrolle des erhaltenen Ergebnisses auf seine Glaubwürdigkeit:
genutzte Software: ProFit v2.2
Der Vergleich der Modellstruktur 1AKO (pdb-file) mit den Aminosären 15 bis 282 der Targetstruktur T0142 ergibt einen RMS-Wert von 12.042. Obwohl dieses Modell somit besser als das erste Modell ist, ist der RMS-Wert immer noch grösser als der Grenzwert 10.
Es konnte sowohl durch das Sequenz-Sequenz- als auch durch das Sequenz-Struktur-Alignment mit den Proteindatenbanken von Blast und 3dpssm kein genügend gutes Modell zur Vorhersage der Proteinstruktur des Targets T0142 gefunden werden.