
| Sequence ID | pao.2374 |
|---|---|
| Location | 4,444,680 – 4,444,919 |
| Length | 239 |
| Max. P | 0.997388 |

| Location | 4,444,680 – 4,444,762 |
|---|---|
| Length | 82 |
| Sequences | 6 |
| Columns | 83 |
| Reading direction | reverse |
| Mean pairwise identity | 70.88 |
| Mean single sequence MFE | -35.38 |
| Consensus MFE | -22.33 |
| Energy contribution | -21.83 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.45 |
| Mean z-score | -3.09 |
| Structure conservation index | 0.63 |
| SVM decision value | 2.85 |
| SVM RNA-class probability | 0.997388 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
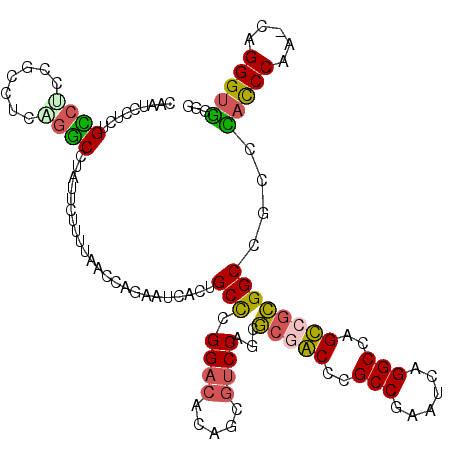
>pao.2374 4444680 82 - 6264404 GUGAGCCGUCGCCAACAGCGCCGAGCCAGGACAGUUAC-CCCAGAAGGGAGGUCGACGGGCCUCCCUUUUUCUUUGCCCGCGA (((.((((.(((.....))).)).))..((.(((....-...((((((((((((....))))))))))))...)))))))).. ( -30.20) >pf5.3295 6190030 80 - 7074893 GUGAGCCGGCGUCAAAAGCGCCGAGCACGGACAAAA---UCUCAAAGGGCGGCCGACGGGCCGCCCUUUUUUAUUGCCUGCGA ......((((((.....)))))).(((.((.(((..---....(((((((((((....)))))))))))....)))))))).. ( -39.00) >pel.2759 5099022 80 - 5888780 GUGAGCCGGUGUCACAAGCGCCGAGCACGGACGACAAU-UCCCGAAGGGAGGCCGCAAGGCCU-CCCUUUUUUAUACCUGCG- ......((((((.....)))))).(((.((.((.....-...))((((((((((....)))))-))))).......))))).- ( -37.70) >pfo.2931 5565622 82 - 6438405 GUGAGCCGACGUCAAAAGCGUCGAGCCAGGACGAAGUUCUCUAGAAGGGAGGCCGACGGGUCUCCCUUUUUUCUUGCCCGCG- (((.((((((((.....)))))).))(((((.((.....)).((((((((((((....)))))))))))).)))))..))).- ( -34.20) >pss.2638 5186776 77 - 6093698 GUGAGCCAGCGUCAAAAGCGCUGCGCCAGGACGAAG---UAUCUCAAGGGGGCCAUUUGGUC--CCCUUUUCUUUGUCUGCU- .((.((((((((.....)))))).))))((((((((---......(((((((((....))))--)))))..))))))))...- ( -37.10) >psp.2565 4998991 77 - 5928787 GUGAGCCAACGUCAAAAGCGUUGCGCCAGGGCGAAG---UAUCUCAAGGGGGCCAUUUGGUC--CCCUUUUCUUUGUCUGCU- .((.((((((((.....)))))).))))((((((((---......(((((((((....))))--)))))..))))))))...- ( -34.10) >consensus GUGAGCCGGCGUCAAAAGCGCCGAGCCAGGACGAAG___UCUCGAAGGGAGGCCGACGGGCCU_CCCUUUUCUUUGCCUGCG_ (((.((((((((.....)))))).)).................(((((((((((....))))).))))))........))).. (-22.33 = -21.83 + -0.50)

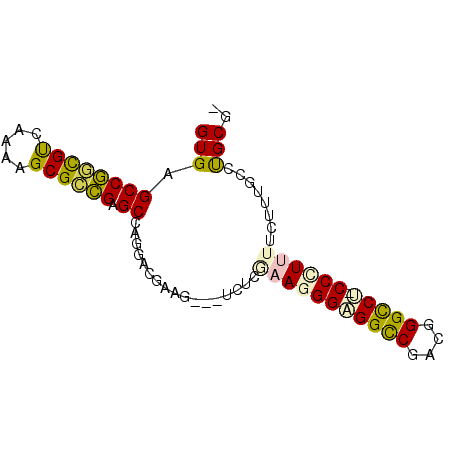
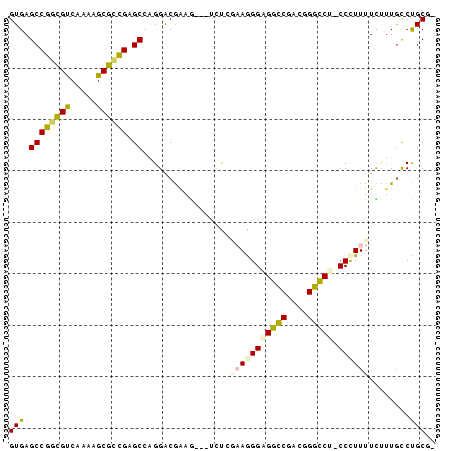
| Location | 4,444,762 – 4,444,881 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.75 |
| Mean single sequence MFE | -60.10 |
| Consensus MFE | -46.31 |
| Energy contribution | -45.85 |
| Covariance contribution | -0.46 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.900534 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2374 4444762 119 + 6264404 CUCACUCCACCUUCUGGGCGGUGUGCAACCCGGAGGCCGGCGCAACCCUG-UAGGGUCGGCGGCCACGGCUGGCCUGAUUGGGCGAGCUGUGAUGGACGAGAUGUCCGGGCCGUGAAUCU .((((.......((((((..(....)..))))))(((((.(((.((((..-..))))..))).)(((((((.((((....)))).))))))).(((((.....))))))))))))).... ( -59.40) >pss.2638 5186853 119 + 6093698 CUCAUGCCGUCUUCUGGACAGUAUGUAGCCCGGGGGCCUGACACACCCUG-UCGGGUGGGAGGCCGCGACUGGCCUGAUUCGGCGGGUCGCGCUGGACGCUGUGUCCGGGCAGUGAUUCU ..(((((.(((.....))).)))))..((((((((((((..(.(((((..-..)))))).)))))((((((.(((......))).))))))((.....))....)))))))......... ( -59.70) >pel.2759 5099102 118 + 5888780 CUCGCGCCGUCUUCUGGACAGUGCGUAGCCCGGGGGCCUGGCGGAUCCUG-UUGGAUGGCCGGCCGCGACCGCCCGUCUGGGGCGCGUCGCCGUGGACUG-AUGUCCGGGCAGUGAUUCU ..(((((.(((.....))).)))))..(((((((((((.(((.(.(((..-..)))).)))))))((((((((((.....))))).))))).........-...)))))))......... ( -62.90) >ppk.2774 5437709 117 + 6181863 CUCACACCGUCUUCUGGAGGGUGUGUAGCCCGGGGGCCUG-CAGAUCCUG-UUGGAUGGCCGGCCGCGACCGCCCGCCUAGGGCGCGUCGCCGUGGACUG-AUGUCCGGGCAGUGAUUCU ..((((((.((.....)).))))))..(((((((((((.(-(..((((..-..)))).)).))))((((((((((.....))))).))))).........-...)))))))......... ( -63.50) >pf5.3295 6190110 119 + 7074893 CUCACUCCGUCUUCUGGACAGUGUGUAGCCCGGGGGCCGGCUCAACCCUG-UGGGAUUGGCGGCCGCGGCAGGCCUGAUUCGGCGGGCCGCAGUGGACUCUAUGUCCGGGCAGUGAUUCU .(((((..(((.....)))........((((((((((((.(.((((((..-.))).)))))))))(((((..(((......)))..))))).............)))))))))))).... ( -54.80) >pfo.2931 5565704 120 + 6438405 CUCACGCCGUCUUCUGGACAGUGUGCAGCUCGGGGGCCGGCACAACCCCGGUGGGGGUGGCGGCCGCGGCGGGCCGGAUUGGGCGGGCCGCAGUGGACUGUGUUACCGAGCAGUGAUUCU ..(((((.(((.....))).)))))..((((((.(((((.(((..((((...))))))).)))))(((((..(((......)))..)))))..............))))))......... ( -60.30) >consensus CUCACGCCGUCUUCUGGACAGUGUGUAGCCCGGGGGCCGGCCCAACCCUG_UGGGAUGGGCGGCCGCGACCGGCCGGAUUGGGCGGGCCGCAGUGGACUGUAUGUCCGGGCAGUGAUUCU ..(((((.(((.....))).)))))..(((((((((((.(((...(((.....)))..)))))))(((((..(((......)))..))))).............)))))))......... (-46.31 = -45.85 + -0.46)
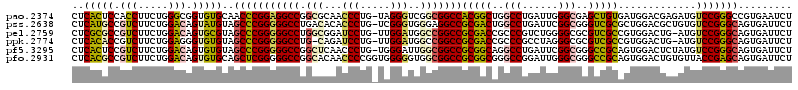

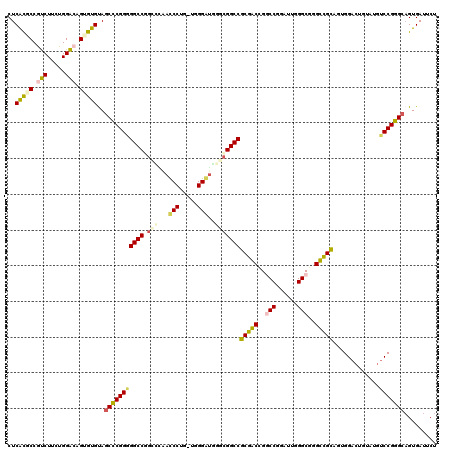
| Location | 4,444,762 – 4,444,881 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.75 |
| Mean single sequence MFE | -56.75 |
| Consensus MFE | -47.70 |
| Energy contribution | -45.98 |
| Covariance contribution | -1.71 |
| Combinations/Pair | 1.44 |
| Mean z-score | -2.79 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.79 |
| SVM RNA-class probability | 0.977262 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2374 4444762 119 - 6264404 AGAUUCACGGCCCGGACAUCUCGUCCAUCACAGCUCGCCCAAUCAGGCCAGCCGUGGCCGCCGACCCUA-CAGGGUUGCGCCGGCCUCCGGGUUGCACACCGCCCAGAAGGUGGAGUGAG .......(((((((((.............((.(((.(((......))).))).))((((((((((((..-..)))))).).))))))))))))))(((.(((((.....))))).))).. ( -55.90) >pss.2638 5186853 119 - 6093698 AGAAUCACUGCCCGGACACAGCGUCCAGCGCGACCCGCCGAAUCAGGCCAGUCGCGGCCUCCCACCCGA-CAGGGUGUGUCAGGCCCCCGGGCUACAUACUGUCCAGAAGACGGCAUGAG .........((((((.....((.....))(((((..(((......)))..)))))(((((.((((((..-..))))).)..))))).))))))..(((.(((((.....))))).))).. ( -54.40) >pel.2759 5099102 118 - 5888780 AGAAUCACUGCCCGGACAU-CAGUCCACGGCGACGCGCCCCAGACGGGCGGUCGCGGCCGGCCAUCCAA-CAGGAUCCGCCAGGCCCCCGGGCUACGCACUGUCCAGAAGACGGCGCGAG .........((((((((..-..))))...(((((.(((((.....))))))))))(((((((.((((..-..))))..))).))))...))))..(((.(((((.....))))).))).. ( -60.40) >ppk.2774 5437709 117 - 6181863 AGAAUCACUGCCCGGACAU-CAGUCCACGGCGACGCGCCCUAGGCGGGCGGUCGCGGCCGGCCAUCCAA-CAGGAUCUG-CAGGCCCCCGGGCUACACACCCUCCAGAAGACGGUGUGAG .........((((((((..-..))))...(((((.(((((.....))))))))))((((.((.((((..-..))))..)-).))))...))))..((((((.((.....)).)))))).. ( -57.80) >pf5.3295 6190110 119 - 7074893 AGAAUCACUGCCCGGACAUAGAGUCCACUGCGGCCCGCCGAAUCAGGCCUGCCGCGGCCGCCAAUCCCA-CAGGGUUGAGCCGGCCCCCGGGCUACACACUGUCCAGAAGACGGAGUGAG ....(((((((((((((.....))))...(((((..(((......)))..)))))((((((((((((..-..)))))).).)))))...))))......(((((.....)))))))))). ( -57.90) >pfo.2931 5565704 120 - 6438405 AGAAUCACUGCUCGGUAACACAGUCCACUGCGGCCCGCCCAAUCCGGCCCGCCGCGGCCGCCACCCCCACCGGGGUUGUGCCGGCCCCCGAGCUGCACACUGUCCAGAAGACGGCGUGAG .........((((((..............(((((..(((......)))..)))))(((((.(((((((...))))..))).))))).))))))..(((.(((((.....))))).))).. ( -54.10) >consensus AGAAUCACUGCCCGGACAUACAGUCCACCGCGACCCGCCCAAUCAGGCCAGCCGCGGCCGCCCACCCAA_CAGGGUUGGGCAGGCCCCCGGGCUACACACUGUCCAGAAGACGGCGUGAG .........((((((..............(((((..(((......)))..)))))(((((..(((((.....)))))....))))).))))))..(((.(((((.....))))).))).. (-47.70 = -45.98 + -1.71)

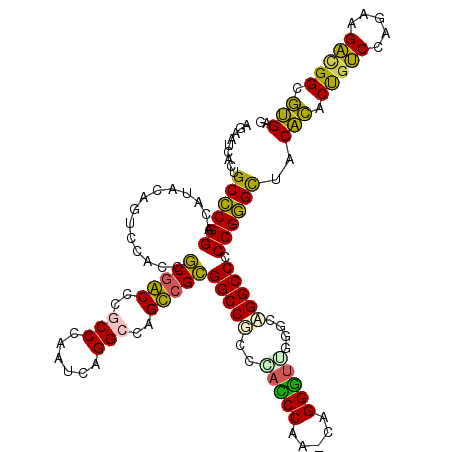
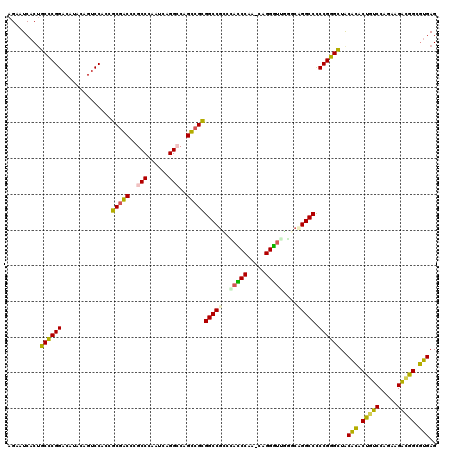
| Location | 4,444,802 – 4,444,919 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 77.16 |
| Mean single sequence MFE | -49.12 |
| Consensus MFE | -34.58 |
| Energy contribution | -35.30 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.91 |
| SVM RNA-class probability | 0.879599 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2374 4444802 117 + 6264404 CGCAACCCUG-UAGGGUCGGCGGCCACGGCUGGCCUGAUUGGGCGAGCUGUGAUGGACGAGAUGUCCGGGCCGUGAAUCUGGGUAAAAGAAUACGUCGCCAGGCGCGACCAAUGAUUG (((.((((..-..((((..((((((((((((.((((....)))).))))))..(((((.....)))))))))))..))))))))..........(((....))))))........... ( -49.40) >pss.2638 5186893 117 + 6093698 ACACACCCUG-UCGGGUGGGAGGCCGCGACUGGCCUGAUUCGGCGGGUCGCGCUGGACGCUGUGUCCGGGCAGUGAUUCUGUUUAAAAGAAUAGGCCUGAGGCUAAGGCAGAAGAUUG ...(((((..-..)))))(..(((.((((((.(((......))).)))))))))...)(((..((((((((....(((((.......)))))..))))).)))...)))......... ( -50.20) >pf5.3295 6190150 117 + 7074893 CUCAACCCUG-UGGGAUUGGCGGCCGCGGCAGGCCUGAUUCGGCGGGCCGCAGUGGACUCUAUGUCCGGGCAGUGAUUCUGGUGAUAAGAAUAGGCGCGCAGCGAAGACAGAGGAUUG ..(((.(((.-((...((.((((((......)))).......(((.(((((..(((((.....))))).))....(((((.......))))).))).))).)).))..)).))).))) ( -45.30) >pfo.2931 5565744 118 + 6438405 CACAACCCCGGUGGGGGUGGCGGCCGCGGCGGGCCGGAUUGGGCGGGCCGCAGUGGACUGUGUUACCGAGCAGUGAUUCUGGUGAUAAGAAUAGGCCCGGAGCCGCGACAGAUGAUUG .....((((...))))((.(((((.....((((((..((((..(((..(((((....)))))...)))..)))).(((((.......))))).))))))..))))).))......... ( -49.10) >psp.2565 4999108 117 + 5928787 GCACACCCUG-UUGGGUGGGAGGCCGCGACUGGCCUGAUUCGGCGAGUCGCGCUGGACGCUGUGUCCGGGCAGUGAUUCUGGUUAAAAGAAUAGGCCUGAGGCGAAGGCAGAGGAUUG ...(((((..-..)))))..(((((((((((.(((......))).))))))((..((((((((......))))))..))..))..........)))))...((....))......... ( -52.10) >pst.2860 5433146 115 + 6397126 GCACACCCUG-UUGGGUGGGAGGCCGCAACUGGCCUGAUUUGGCGAGU--CGCUGGACGCUGUGUCCGGGCAGUGAUUCUGUUUAAAAGAAUAGGCCUGAGGCAGUGGCAGGGGAUUG ((.(((((..-..)))))..((((((....))))))......)).(((--(.((...((((((.((.((((....(((((.......)))))..)))))).))))))...)).)))). ( -48.60) >consensus CCAAACCCUG_UGGGGUGGGAGGCCGCGACUGGCCUGAUUCGGCGAGCCGCGCUGGACGCUGUGUCCGGGCAGUGAUUCUGGUUAAAAGAAUAGGCCUGAAGCCAAGACAGAGGAUUG ...(((((.....)))))..(((((((((((.(((......))).))))))..(((((.....))))).......(((((.......))))).))))).................... (-34.58 = -35.30 + 0.72)

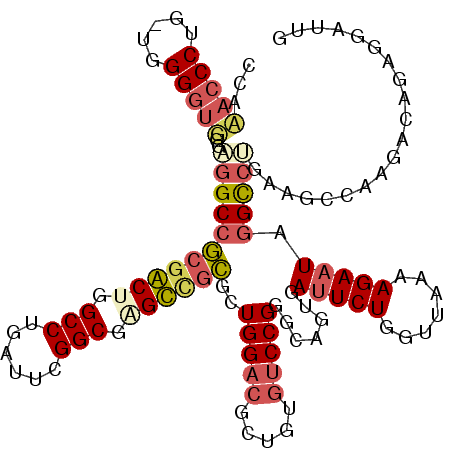
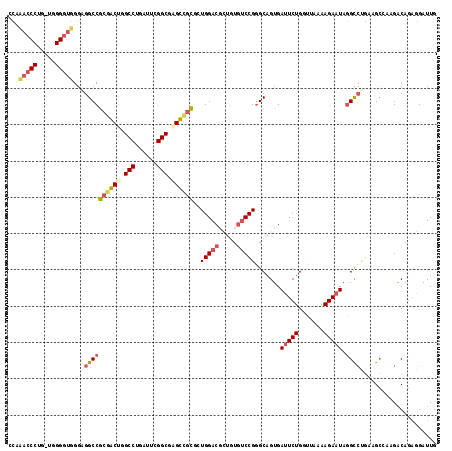
| Location | 4,444,802 – 4,444,919 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 77.16 |
| Mean single sequence MFE | -41.11 |
| Consensus MFE | -28.95 |
| Energy contribution | -28.23 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.42 |
| Mean z-score | -2.30 |
| Structure conservation index | 0.70 |
| SVM decision value | 1.82 |
| SVM RNA-class probability | 0.978584 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2374 4444802 117 - 6264404 CAAUCAUUGGUCGCGCCUGGCGACGUAUUCUUUUACCCAGAUUCACGGCCCGGACAUCUCGUCCAUCACAGCUCGCCCAAUCAGGCCAGCCGUGGCCGCCGACCCUA-CAGGGUUGCG .((((....(((((.....)))))(((......)))...))))..(((((.((((.....))))...((.(((.(((......))).))).))))))).((((((..-..)))))).. ( -42.60) >pss.2638 5186893 117 - 6093698 CAAUCUUCUGCCUUAGCCUCAGGCCUAUUCUUUUAAACAGAAUCACUGCCCGGACACAGCGUCCAGCGCGACCCGCCGAAUCAGGCCAGUCGCGGCCUCCCACCCGA-CAGGGUGUGU .........(((...((....(((..(((((.......)))))....))).((((.....)))).))(((((..(((......)))..))))))))....(((((..-..)))))... ( -39.50) >pf5.3295 6190150 117 - 7074893 CAAUCCUCUGUCUUCGCUGCGCGCCUAUUCUUAUCACCAGAAUCACUGCCCGGACAUAGAGUCCACUGCGGCCCGCCGAAUCAGGCCUGCCGCGGCCGCCAAUCCCA-CAGGGUUGAG (((((((.((........(((.(((.(((((.......)))))........((((.....))))...(((((..(((......)))..)))))))))))......))-.))))))).. ( -39.94) >pfo.2931 5565744 118 - 6438405 CAAUCAUCUGUCGCGGCUCCGGGCCUAUUCUUAUCACCAGAAUCACUGCUCGGUAACACAGUCCACUGCGGCCCGCCCAAUCCGGCCCGCCGCGGCCGCCACCCCCACCGGGGUUGUG .........((((((((...(((((.....(((((..(((.....)))...)))))..(((....))).)))))(((......)))..))))))))(((.(((((....))))).))) ( -46.60) >psp.2565 4999108 117 - 5928787 CAAUCCUCUGCCUUCGCCUCAGGCCUAUUCUUUUAACCAGAAUCACUGCCCGGACACAGCGUCCAGCGCGACUCGCCGAAUCAGGCCAGUCGCGGCCUCCCACCCAA-CAGGGUGUGC .........(((...((....(((..(((((.......)))))....))).((((.....)))).))((((((.(((......))).)))))))))....(((((..-..)))))... ( -41.30) >pst.2860 5433146 115 - 6397126 CAAUCCCCUGCCACUGCCUCAGGCCUAUUCUUUUAAACAGAAUCACUGCCCGGACACAGCGUCCAGCG--ACUCGCCAAAUCAGGCCAGUUGCGGCCUCCCACCCAA-CAGGGUGUGC .........(((.........(((..(((((.......)))))....))).((((.....)))).(((--(((.(((......))).)))))))))....(((((..-..)))))... ( -36.70) >consensus CAAUCCUCUGCCUCCGCCUCAGGCCUAUUCUUUUAACCAGAAUCACUGCCCGGACACAGCGUCCAGCGCGACCCGCCGAAUCAGGCCAGCCGCGGCCGCCCACCCAA_CAGGGUGGGG .........((((.......)))).......................(((.((((.....))))...(((((..(((......)))..))))))))....(((((.....)))))... (-28.95 = -28.23 + -0.72)

Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:12:09 2007