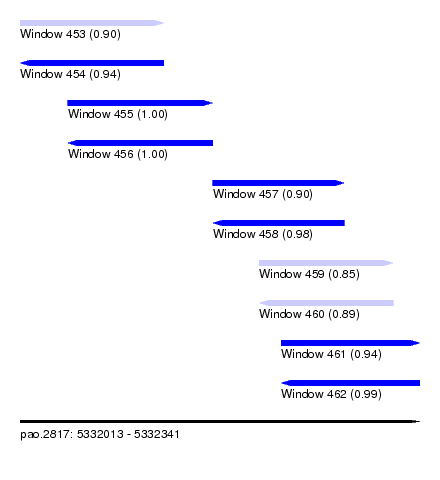
| Sequence ID | pao.2817 |
|---|---|
| Location | 5,332,013 – 5,332,341 |
| Length | 328 |
| Max. P | 0.999932 |

| Location | 5,332,013 – 5,332,131 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.13 |
| Mean single sequence MFE | -46.72 |
| Consensus MFE | -30.85 |
| Energy contribution | -33.64 |
| Covariance contribution | 2.78 |
| Combinations/Pair | 1.34 |
| Mean z-score | -3.05 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.899640 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
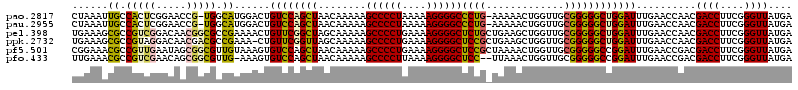
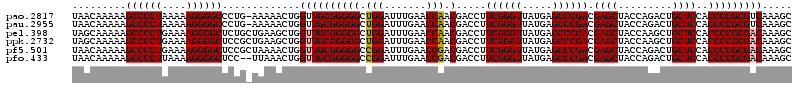
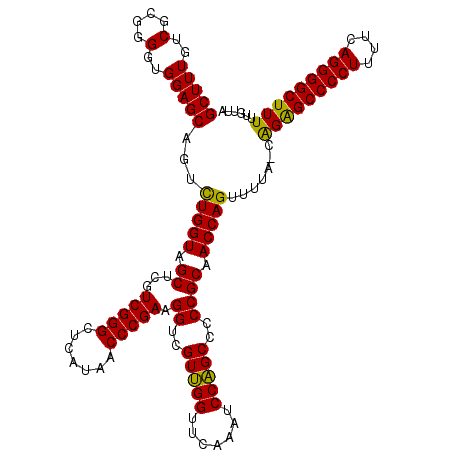
>pao.2817 5332013 118 + 6264404 CUAAAUUGCCACUCGGAACCG-UGGCAUGGACUGUCCAGCUAACAAAAAGCCCCUAAAAAGGGGCCCUG-AAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGA ......((((((........)-)))))(((...((((((((........((((((....))))))((((-...((....)).)))))))))))).....)))..((((....)))).... ( -46.50) >pau.2955 5603942 118 + 6537648 CUAAAUUGCCACUCGGAACCG-UGGCAUGGACUGUCCAGCUAACAAAAAGCCCCUAAAAAGGGGCCCUG-AAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGA ......((((((........)-)))))(((...((((((((........((((((....))))))((((-...((....)).)))))))))))).....)))..((((....)))).... ( -46.50) >pel.398 5066153 120 - 5888780 UGAAAGCGCCGUCGGACAACGGCGCCGAAAACUGUUCGGCUAGCAAAAAGCCCCUGAAAAGGGGCUCUGCUGAAGCUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGA .......(((.(((.(...((((((((((.....))))))(((((...(((((((....))))))).)))))..))))..).))).)))(((.......)))..((((....)))).... ( -52.20) >ppk.2732 5360839 119 + 6181863 UGAAAGCGCCGUAGGACAACGACGCCGAAA-CUGUUCGGUUAGCAAAAAGCCCCUGAAAAGGGGCUCCGCUGAAGCUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGA .....(((.(((......))).))).....-..(((((.(((((....(((((((....)))))))((((...........))))..)))))...)))))....((((....)))).... ( -42.00) >pf5.501 6103596 120 - 7074893 CGGAAACGCCGUUGAAUAGCGGCGUUGUAAAGUGUCCAGCUAACAAAAAGCCCCUGAAAAGGGGCUCCGCUAAAACUGGUUGCGGGGGCCGGAUUUGAACCGACGACCUUCGGGUUAUGA (((.((((((((......)))))))).......((((.(((.......(((((((....)))))))((((...........)))).))).)))).....)))..((((....)))).... ( -51.70) >pfo.433 5526746 117 - 6438405 UUGAAACGCCGUCGAACAGCGGCGUUG-AAAGUGUCCAGCUAACAAAAAGCCCCUUAAAAGGGGCUCC--UUAAACUGGUUGCGGGGGCCGGAUUUGAACCGACGACCUUCGGGUUAUGA ....((((((((......)))))))).-.....((((.(((..(....(((((((....)))))))((--.......))....)..))).))))..........((((....)))).... ( -41.40) >consensus CGAAAACGCCGUUGGACAACGGCGCCGUAAACUGUCCAGCUAACAAAAAGCCCCUGAAAAGGGGCUCCG_UAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGA ......((.(((((.....))))).))......((((((((........((((((....))))))((((.............))))))))))))..........((((....)))).... (-30.85 = -33.64 + 2.78)

| Location | 5,332,013 – 5,332,131 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.13 |
| Mean single sequence MFE | -44.03 |
| Consensus MFE | -34.57 |
| Energy contribution | -34.97 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.28 |
| Mean z-score | -3.01 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.31 |
| SVM RNA-class probability | 0.939979 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
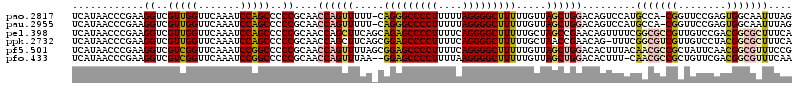
>pao.2817 5332013 118 - 6264404 UCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUU-CAGGGCCCCUUUUUAGGGGCUUUUUGUUAGCUGGACAGUCCAUGCCA-CGGUUCCGAGUGGCAAUUUAG ............((..(((((.......)))))..))....((((((...-.(((((((((....))))))))).....))))))........(((((-(........))))))...... ( -41.70) >pau.2955 5603942 118 - 6537648 UCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUU-CAGGGCCCCUUUUUAGGGGCUUUUUGUUAGCUGGACAGUCCAUGCCA-CGGUUCCGAGUGGCAAUUUAG ............((..(((((.......)))))..))....((((((...-.(((((((((....))))))))).....))))))........(((((-(........))))))...... ( -41.70) >pel.398 5066153 120 + 5888780 UCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGCUUCAGCAGAGCCCCUUUUCAGGGGCUUUUUGCUAGCCGAACAGUUUUCGGCGCCGUUGUCCGACGGCGCUUUCA .........(((((..(((((.......)))))..))((..........((((((((((((....))))))))..)))).((((((.....))))))((((((....)))))))).))). ( -48.80) >ppk.2732 5360839 119 - 6181863 UCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGCUUCAGCGGAGCCCCUUUUCAGGGGCUUUUUGCUAACCGAACAG-UUUCGGCGUCGUUGUCCUACGGCGCUUUCA ........((((((..(((((.......)))))..))((((...((....))(((((((((....)))))))))))))...........-.))))(((((((......)))))))..... ( -41.80) >pf5.501 6103596 120 + 7074893 UCAUAACCCGAAGGUCGUCGGUUCAAAUCCGGCCCCCGCAACCAGUUUUAGCGGAGCCCCUUUUCAGGGGCUUUUUGUUAGCUGGACACUUUACAACGCCGCUAUUCAACGGCGUUUCCG ............((..(((((.......)))))..))....((((((...(..((((((((....))))))))..)...)))))).........(((((((........))))))).... ( -43.40) >pfo.433 5526746 117 + 6438405 UCAUAACCCGAAGGUCGUCGGUUCAAAUCCGGCCCCCGCAACCAGUUUAA--GGAGCCCCUUUUAAGGGGCUUUUUGUUAGCUGGACACUUU-CAACGCCGCUGUUCGACGGCGUUUCAA .........((((((.(((((.......)))))........(((((((((--(((((((((....))))))))))))..))))))..)))))-)(((((((........))))))).... ( -46.80) >consensus UCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUA_CAGAGCCCCUUUUCAGGGGCUUUUUGUUAGCUGGACAGUUUACGACGCCGCUGUCCAACGGCGAUUUAA ............((..(((((.......)))))..))....((((((.....(((((((((....))))))))).....)))))).........(((((((........))))))).... (-34.57 = -34.97 + 0.39)
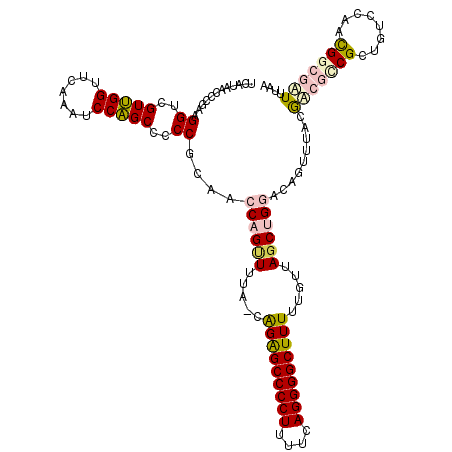
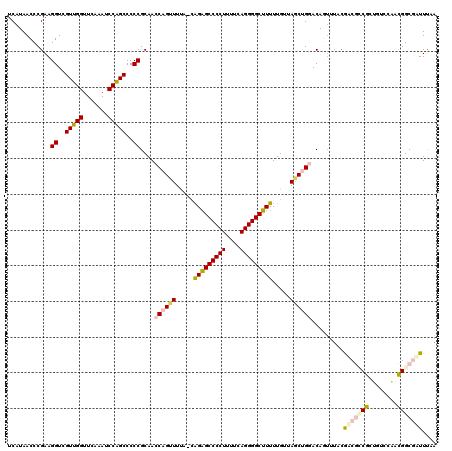
| Location | 5,332,052 – 5,332,171 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.49 |
| Mean single sequence MFE | -48.08 |
| Consensus MFE | -45.26 |
| Energy contribution | -45.90 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.12 |
| Structure conservation index | 0.94 |
| SVM decision value | 4.64 |
| SVM RNA-class probability | 0.999932 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2817 5332052 119 + 6264404 UAACAAAAAGCCCCUAAAAAGGGGCCCUG-AAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCCACCCCGCGUCAAAGC .........((((((....))))))....-.....((...((((((((.(((.......))).).....((((((.....)))))).((((.........))))..)))))))....)). ( -40.80) >pau.2955 5603981 119 + 6537648 UAACAAAAAGCCCCUAAAAAGGGGCCCUG-AAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCCACCCCGCGUCAAAGC .........((((((....))))))....-.....((...((((((((.(((.......))).).....((((((.....)))))).((((.........))))..)))))))....)). ( -40.80) >pel.398 5066193 120 - 5888780 UAGCAAAAAGCCCCUGAAAAGGGGCUCUGCUGAAGCUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAAGCUGCUCCACCCCGCGACAAAGC (((((...(((((((....))))))).)))))..(((.(((((((((..((((..............(.((((((.....)))))).)((((....))))..)))))))))))))..))) ( -55.80) >ppk.2732 5360878 120 + 6181863 UAGCAAAAAGCCCCUGAAAAGGGGCUCCGCUGAAGCUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAAGCUGCUCCACCCCGCGACAAAGC ((((....(((((((....)))))))..))))..(((.(((((((((..((((..............(.((((((.....)))))).)((((....))))..)))))))))))))..))) ( -54.10) >pf5.501 6103636 120 - 7074893 UAACAAAAAGCCCCUGAAAAGGGGCUCCGCUAAAACUGGUUGCGGGGGCCGGAUUUGAACCGACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCCACCCCGCGACAAAGC ........(((((((....)))))))............(((((((((..(((.......))).......((((((.....)))))).((((.........))))..)))))))))..... ( -49.80) >pfo.433 5526785 118 - 6438405 UAACAAAAAGCCCCUUAAAAGGGGCUCC--UUAAACUGGUUGCGGGGGCCGGAUUUGAACCGACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCCACCCCGCGACAAAGC ........(((((((....)))))))..--........(((((((((..(((.......))).......((((((.....)))))).((((.........))))..)))))))))..... ( -47.20) >consensus UAACAAAAAGCCCCUGAAAAGGGGCUCCG_UAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCCACCCCGCGACAAAGC .........((((((....)))))).............((((((((((.(((.......))).).....((((((.....)))))).((((.........))))..)))))))))..... (-45.26 = -45.90 + 0.64)
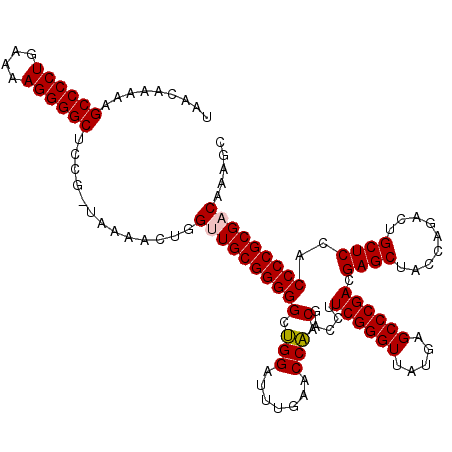
| Location | 5,332,052 – 5,332,171 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.49 |
| Mean single sequence MFE | -50.18 |
| Consensus MFE | -46.57 |
| Energy contribution | -45.43 |
| Covariance contribution | -1.14 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.93 |
| SVM decision value | 3.44 |
| SVM RNA-class probability | 0.999220 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2817 5332052 119 - 6264404 GCUUUGACGCGGGGUGGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUU-CAGGGCCCCUUUUUAGGGGCUUUUUGUUA ....((((((((((((((.....(..(..(((..(((((.......))))).)))..)..)......))))..))))))...........-.(((((((((....)))))))))..)))) ( -46.90) >pau.2955 5603981 119 - 6537648 GCUUUGACGCGGGGUGGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUU-CAGGGCCCCUUUUUAGGGGCUUUUUGUUA ....((((((((((((((.....(..(..(((..(((((.......))))).)))..)..)......))))..))))))...........-.(((((((((....)))))))))..)))) ( -46.90) >pel.398 5066193 120 + 5888780 GCUUUGUCGCGGGGUGGAGCAGCUUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGCUUCAGCAGAGCCCCUUUUCAGGGGCUUUUUGCUA ((((..((....))..))))((((.(((.((...(((((.......))))).((..(((((.......)))))..)))).)))))))..((((((((((((....))))))))..)))). ( -52.70) >ppk.2732 5360878 120 - 6181863 GCUUUGUCGCGGGGUGGAGCAGCUUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGCUUCAGCGGAGCCCCUUUUCAGGGGCUUUUUGCUA ((((..((....))..))))((((.(((.((...(((((.......))))).((..(((((.......)))))..)))).)))))))..((((((((((((....))))))))..)))). ( -52.00) >pf5.501 6103636 120 + 7074893 GCUUUGUCGCGGGGUGGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUCGGUUCAAAUCCGGCCCCCGCAACCAGUUUUAGCGGAGCCCCUUUUCAGGGGCUUUUUGUUA ((((..((....))..))))...(((((.((...(((((.......))))).((..(((((.......)))))..)))).)))))...(((((((((((((....))))))))..))))) ( -50.40) >pfo.433 5526785 118 + 6438405 GCUUUGUCGCGGGGUGGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUCGGUUCAAAUCCGGCCCCCGCAACCAGUUUAA--GGAGCCCCUUUUAAGGGGCUUUUUGUUA ((((..((....))..))))...(((((.((...(((((.......))))).((..(((((.......)))))..)))).)))))..(((--(((((((((....))))))))))))... ( -52.20) >consensus GCUUUGUCGCGGGGUGGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUA_CAGAGCCCCUUUUCAGGGGCUUUUUGUUA (((((..(....)..)))))...(((((.((...(((((.......))))).((..(((((.......)))))..)))).))))).......(((((((((....)))))))))...... (-46.57 = -45.43 + -1.14)
| Location | 5,332,171 – 5,332,279 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.62 |
| Mean single sequence MFE | -40.21 |
| Consensus MFE | -24.02 |
| Energy contribution | -24.60 |
| Covariance contribution | 0.58 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.04 |
| SVM RNA-class probability | 0.904466 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2817 5332171 108 + 6264404 UGGUGCAAGAUUAUACGACCGACACCC-UGAUGGGUCAA--CCGAACCU---UGCGGAGACAAACCCCGCA-GGACGGGGCUUGCU-----UAUAUGGUACCGAGGAGGGGACUCGAACC .(((((.....((..(((((...((((-....))))...--(((...((---(((((.(......))))))-)).))))).)))..-----))....)))))...(((....)))..... ( -36.30) >pel.398 5066313 117 - 5888780 UGGGGCGAAAGUAUAAGACUGAACCCUUUGCUGGGUCAA--UCCGACCU-CCACCUGCAAGAAAGCCCGCUCAAAGCGGGCUUUCAUGCUUCAAUUGGUACCGAGAAGGGGACUCGAACC .((..(((.(((.....)))...((((((.((.(((...--....(((.-......(((.((((((((((.....)))))))))).))).......)))))).))))))))..)))..)) ( -45.65) >ppk.2732 5360998 118 + 6181863 UGGGGCAAAAGUAUAAGACCGAACCCUGUUGAGGGUCAAAUCCAAACAU-UCACCUACAAGAAAGCCCGCU-AAAGCGGGCUCUCAAGCUUUAAUUGGUACCGAGAAGGGGACUCGAACC ((((((..............((.((((....))))))............-..........((.(((((((.-...))))))).))..))))))...(((..((((.......)))).))) ( -32.90) >pf5.501 6103756 114 - 7074893 UGGGGCGGAAGUAUACGACUGAUCCCU-UUUUGGGUCAA--UGUAACCUUCCACCUACAAGAAAGCCCGCA-ACAGCGGGCUCUCCUGA--UAAUUGGUACCGAGAAGGGGACUCGAACC ..(((.(((((..((((..((((((..-....)))))).--))))..))))).)))....((..((((((.-...))))))((((((..--...(((....)))..)))))).))..... ( -42.70) >pfo.433 5526903 115 - 6438405 UGGGGCGGAAGUAUACGACCGAUCCCU-UAUAGGGUCAA--UGUAACCUUCCACCUGCAAGAAAGCCCGCA-ACAGCGGGCUCUCCUGAC-UAAUUGGUACCGAGAAGGGGACUCGAACC ..(((.(((((..((((...(((((..-....)))))..--))))..))))).))).((.((.(((((((.-...))))))).)).))..-.....(((..((((.......)))).))) ( -41.10) >pst.2667 5061374 113 + 6397126 UGGGGCGGAAGUAUACGACCGAUCCAU-UGCUGGGUCAA--UCCAACAU-CCACCUACAAGAAAGCCCGCU-AAUGCGGGCUUCCUUGU--UAAUUGGUACCGAGAAGGGGACUCGAACC (((((((........)).))((((((.-...))))))..--.)))...(-(((((.(((((.((((((((.-...)))))))).)))))--.....))).((.....)))))........ ( -42.60) >consensus UGGGGCGAAAGUAUACGACCGAUCCCU_UGCUGGGUCAA__UCCAACCU_CCACCUACAAGAAAGCCCGCA_AAAGCGGGCUCUCAUGA__UAAUUGGUACCGAGAAGGGGACUCGAACC ((((((....))....((((.............))))................))))...((.(((((((.....))))))).))...........(((..((((.......)))).))) (-24.02 = -24.60 + 0.58)

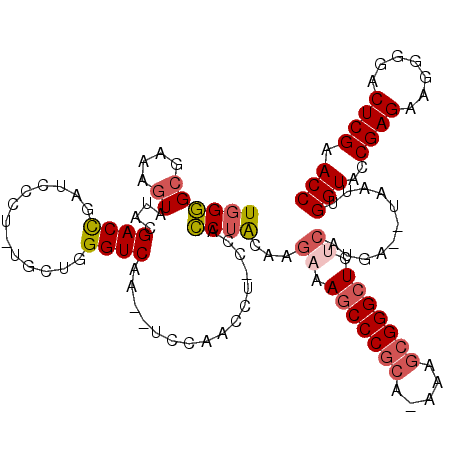
| Location | 5,332,171 – 5,332,279 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.62 |
| Mean single sequence MFE | -54.07 |
| Consensus MFE | -35.76 |
| Energy contribution | -38.18 |
| Covariance contribution | 2.42 |
| Combinations/Pair | 1.19 |
| Mean z-score | -5.44 |
| Structure conservation index | 0.66 |
| SVM decision value | 1.87 |
| SVM RNA-class probability | 0.980829 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2817 5332171 108 - 6264404 GGUUCGAGUCCCCUCCUCGGUACCAUAUA-----AGCAAGCCCCGUCC-UGCGGGGUUUGUCUCCGCA---AGGUUCGG--UUGACCCAUCA-GGGUGUCGGUCGUAUAAUCUUGCACCA ((((((((.......))))).))).....-----.((((((((((...-..))))))))))....(((---(((((((.--.((((((....-.)).))))..))...)))))))).... ( -45.30) >pel.398 5066313 117 + 5888780 GGUUCGAGUCCCCUUCUCGGUACCAAUUGAAGCAUGAAAGCCCGCUUUGAGCGGGCUUUCUUGCAGGUGG-AGGUCGGA--UUGACCCAGCAAAGGGUUCAGUCUUAUACUUUCGCCCCA ((((((((.......))))).))).......(((.((((((((((.....)))))))))).))).(((((-((((.(((--(((((((......))).)))))))...)))))))))... ( -57.10) >ppk.2732 5360998 118 - 6181863 GGUUCGAGUCCCCUUCUCGGUACCAAUUAAAGCUUGAGAGCCCGCUUU-AGCGGGCUUUCUUGUAGGUGA-AUGUUUGGAUUUGACCCUCAACAGGGUUCGGUCUUAUACUUUUGCCCCA ((((((((.......))))).))).......((..((((((((((...-.))))))))))..)).((..(-(.((..((((..((((((....))))))..))))...)).))..))... ( -50.20) >pf5.501 6103756 114 + 7074893 GGUUCGAGUCCCCUUCUCGGUACCAAUUA--UCAGGAGAGCCCGCUGU-UGCGGGCUUUCUUGUAGGUGGAAGGUUACA--UUGACCCAAAA-AGGGAUCAGUCGUAUACUUCCGCCCCA ((((((((.......))))).))).....--.(((((((((((((...-.)))))))))))))..((((((((..((((--(((((((....-.))).))))).)))..))))))))... ( -58.10) >pfo.433 5526903 115 + 6438405 GGUUCGAGUCCCCUUCUCGGUACCAAUUA-GUCAGGAGAGCCCGCUGU-UGCGGGCUUUCUUGCAGGUGGAAGGUUACA--UUGACCCUAUA-AGGGAUCGGUCGUAUACUUCCGCCCCA ((((((((.......))))).))).....-(.(((((((((((((...-.)))))))))))))).((((((((..((((--(((((((....-.))).))))).)))..))))))))... ( -58.30) >pst.2667 5061374 113 - 6397126 GGUUCGAGUCCCCUUCUCGGUACCAAUUA--ACAAGGAAGCCCGCAUU-AGCGGGCUUUCUUGUAGGUGG-AUGUUGGA--UUGACCCAGCA-AUGGAUCGGUCGUAUACUUCCGCCCCA ((((((((.......))))).))).....--((((((((((((((...-.)))))))))))))).(((((-(.((..((--((((.(((...-.))).))))))....)).))))))... ( -55.40) >consensus GGUUCGAGUCCCCUUCUCGGUACCAAUUA__GCAAGAAAGCCCGCUUU_AGCGGGCUUUCUUGCAGGUGG_AGGUUAGA__UUGACCCAACA_AGGGAUCGGUCGUAUACUUCCGCCCCA ((((((((.......))))).)))........(((((((((((((.....)))))))))))))..(((((.((((......(((((((......))).))))......)))))))))... (-35.76 = -38.18 + 2.42)
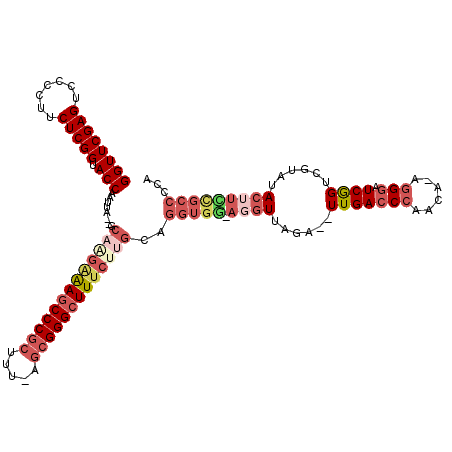
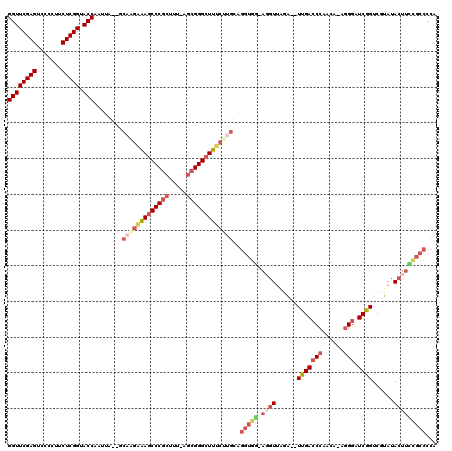
| Location | 5,332,209 – 5,332,319 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.53 |
| Mean single sequence MFE | -40.72 |
| Consensus MFE | -25.82 |
| Energy contribution | -26.10 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.53 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.80 |
| SVM RNA-class probability | 0.853162 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2817 5332209 110 + 6264404 -CCGAACCU---UGCGGAGACAAACCCCGCA-GGACGGGGCUUGCU-----UAUAUGGUACCGAGGAGGGGACUCGAACCCCUACAGCCUAUGGCCACUACCACCUCAAGGUAGCGUGUC -.((.((((---((.((((.(((.(((((..-...))))).)))))-----....((((((((((((((((.......)))))....))).)))....))))))).))))))..)).... ( -39.00) >pau.2955 5604138 110 + 6537648 -CCGAACCU---UGCGGAGACAAACCCCGCA-GGACGGGGCUUGCU-----UAUAUGGUACCGAGGAGGGGACUCGAACCCCUACAGCCUAUGGCCACUACCACCUCAAGGUAGCGUGUC -.((.((((---((.((((.(((.(((((..-...))))).)))))-----....((((((((((((((((.......)))))....))).)))....))))))).))))))..)).... ( -39.00) >pel.398 5066352 118 - 5888780 -UCCGACCU-CCACCUGCAAGAAAGCCCGCUCAAAGCGGGCUUUCAUGCUUCAAUUGGUACCGAGAAGGGGACUCGAACCCCUACACCCUAUGGGCACAACCACCUCAAGGUUGCGUGUC -.((((((.-......(((.((((((((((.....)))))))))).))).......))).....(.(((((.......))))).)......)))((.(((((.......))))).))... ( -44.94) >ppk.2732 5361038 118 + 6181863 UCCAAACAU-UCACCUACAAGAAAGCCCGCU-AAAGCGGGCUCUCAAGCUUUAAUUGGUACCGAGAAGGGGACUCGAACCCCUACACCCUAUGGGCACAACCACCUCAAGGUUGCGUGUC .....((((-.(((((....((.(((((((.-...))))))).))..(((......)))...(((.(((((.......)))))........(((......))).))).))).)).)))). ( -36.70) >pf5.501 6103794 116 - 7074893 -UGUAACCUUCCACCUACAAGAAAGCCCGCA-ACAGCGGGCUCUCCUGA--UAAUUGGUACCGAGAAGGGGACUCGAACCCCUACACCCUAUGGGCACAACCACCUCAAGGUUGCGUGUC -.(((((((((((..((((.((.(((((((.-...))))))).)).)).--))..)))....(((.(((((.......)))))........(((......))).)))))))))))..... ( -42.60) >pfo.433 5526941 117 - 6438405 -UGUAACCUUCCACCUGCAAGAAAGCCCGCA-ACAGCGGGCUCUCCUGAC-UAAUUGGUACCGAGAAGGGGACUCGAACCCCUACACCCUAUGGGCACAACCACCUCAAGGUUGCGUGUC -.(((((((((((...(((.((.(((((((.-...))))))).)).)).)-....)))....(((.(((((.......)))))........(((......))).)))))))))))..... ( -42.10) >consensus _CCGAACCU_CCACCUGCAAGAAAGCCCGCA_AAAGCGGGCUCUCAUG___UAAUUGGUACCGAGAAGGGGACUCGAACCCCUACACCCUAUGGGCACAACCACCUCAAGGUUGCGUGUC .....(((...............(((((((.....)))))))..............))).....(.(((((.......))))).).(((...)))(.(((((.......))))).).... (-25.82 = -26.10 + 0.28)

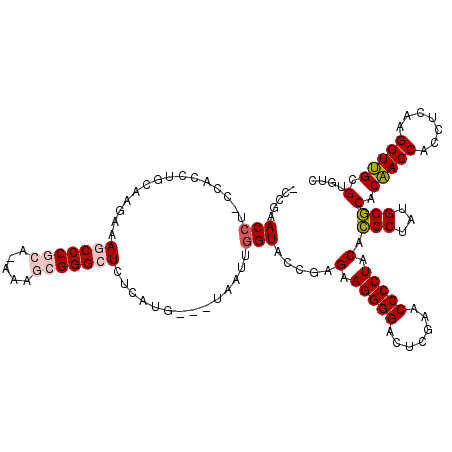
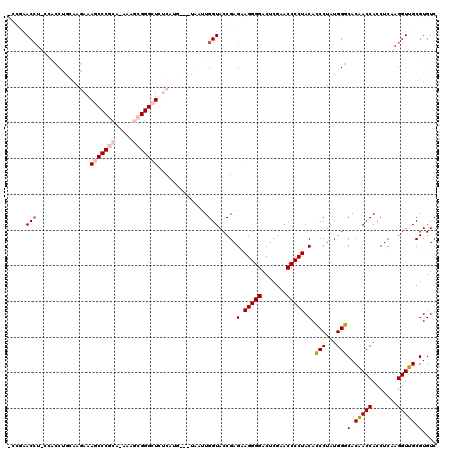
| Location | 5,332,209 – 5,332,319 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.53 |
| Mean single sequence MFE | -49.05 |
| Consensus MFE | -34.09 |
| Energy contribution | -34.82 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.54 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.97 |
| SVM RNA-class probability | 0.892147 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2817 5332209 110 - 6264404 GACACGCUACCUUGAGGUGGUAGUGGCCAUAGGCUGUAGGGGUUCGAGUCCCCUCCUCGGUACCAUAUA-----AGCAAGCCCCGUCC-UGCGGGGUUUGUCUCCGCA---AGGUUCGG- ....((..((((((.(((((((.(((((...))).(.(((((.......))))).).)).)))))....-----.((((((((((...-..))))))))))..)).))---)))).)).- ( -47.30) >pau.2955 5604138 110 - 6537648 GACACGCUACCUUGAGGUGGUAGUGGCCAUAGGCUGUAGGGGUUCGAGUCCCCUCCUCGGUACCAUAUA-----AGCAAGCCCCGUCC-UGCGGGGUUUGUCUCCGCA---AGGUUCGG- ....((..((((((.(((((((.(((((...))).(.(((((.......))))).).)).)))))....-----.((((((((((...-..))))))))))..)).))---)))).)).- ( -47.30) >pel.398 5066352 118 + 5888780 GACACGCAACCUUGAGGUGGUUGUGCCCAUAGGGUGUAGGGGUUCGAGUCCCCUUCUCGGUACCAAUUGAAGCAUGAAAGCCCGCUUUGAGCGGGCUUUCUUGCAGGUGG-AGGUCGGA- (((.(((((((.......))))))).((((.(((...(((((.......))))).))).............(((.((((((((((.....)))))))))).)))..))))-..)))...- ( -52.60) >ppk.2732 5361038 118 - 6181863 GACACGCAACCUUGAGGUGGUUGUGCCCAUAGGGUGUAGGGGUUCGAGUCCCCUUCUCGGUACCAAUUAAAGCUUGAGAGCCCGCUUU-AGCGGGCUUUCUUGUAGGUGA-AUGUUUGGA ....(((((((.......))))))).(((..(((...(((((.......))))).)))..((((.......((..((((((((((...-.))))))))))..)).)))).-.....))). ( -45.20) >pf5.501 6103794 116 + 7074893 GACACGCAACCUUGAGGUGGUUGUGCCCAUAGGGUGUAGGGGUUCGAGUCCCCUUCUCGGUACCAAUUA--UCAGGAGAGCCCGCUGU-UGCGGGCUUUCUUGUAGGUGGAAGGUUACA- .....(.((((((((((....((..(((...)))..))((((.......)))).))))....(((.(((--.(((((((((((((...-.)))))))))))))))).))))))))).).- ( -51.50) >pfo.433 5526941 117 + 6438405 GACACGCAACCUUGAGGUGGUUGUGCCCAUAGGGUGUAGGGGUUCGAGUCCCCUUCUCGGUACCAAUUA-GUCAGGAGAGCCCGCUGU-UGCGGGCUUUCUUGCAGGUGGAAGGUUACA- (((.(((((((.......))))))).((((.(((...(((((.......))))).)))...........-(.(((((((((((((...-.))))))))))))))..))))...)))...- ( -50.40) >consensus GACACGCAACCUUGAGGUGGUUGUGCCCAUAGGGUGUAGGGGUUCGAGUCCCCUUCUCGGUACCAAUUA___CAAGAAAGCCCGCUCU_UGCGGGCUUUCUUGCAGGUGG_AGGUUAGA_ (((.(((((((.......)))))))((....(((...(((((.......))))).))).................((((((((((.....)))))))))).....))......))).... (-34.09 = -34.82 + 0.72)

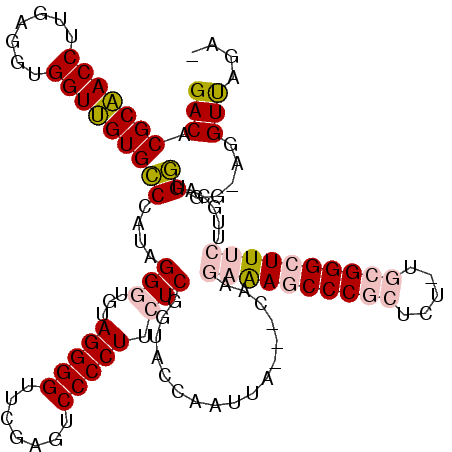
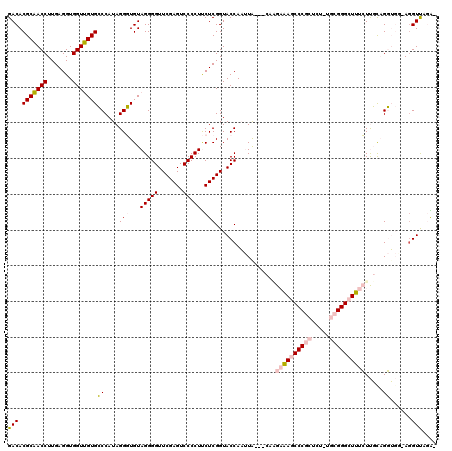
| Location | 5,332,227 – 5,332,341 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.00 |
| Mean single sequence MFE | -40.00 |
| Consensus MFE | -33.78 |
| Energy contribution | -35.00 |
| Covariance contribution | 1.22 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.59 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.28 |
| SVM RNA-class probability | 0.940151 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2817 5332227 114 + 6264404 AACCCCGCA-GGACGGGGCUUGCU-----UAUAUGGUACCGAGGAGGGGACUCGAACCCCUACAGCCUAUGGCCACUACCACCUCAAGGUAGCGUGUCUACCAAUUCCACCACCUCGGCA ..(((((..-...)))))..((((-----.....))))(((((((((((.......))))).........((((.(((((.......))))).).)))..............)))))).. ( -38.50) >pau.2955 5604156 114 + 6537648 AACCCCGCA-GGACGGGGCUUGCU-----UAUAUGGUACCGAGGAGGGGACUCGAACCCCUACAGCCUAUGGCCACUACCACCUCAAGGUAGCGUGUCUACCAAUUCCACCACCUCGGCA ..(((((..-...)))))..((((-----.....))))(((((((((((.......))))).........((((.(((((.......))))).).)))..............)))))).. ( -38.50) >pel.398 5066372 120 - 5888780 AAGCCCGCUCAAAGCGGGCUUUCAUGCUUCAAUUGGUACCGAGAAGGGGACUCGAACCCCUACACCCUAUGGGCACAACCACCUCAAGGUUGCGUGUCUACCAAUUCCACCACCUCGGCA ((((((((.....))))))))...((((......))))(((((.(((((.......)))))........(((((((((((.......))))..))))))).............))))).. ( -41.80) >ppk.2732 5361059 119 + 6181863 AAGCCCGCU-AAAGCGGGCUCUCAAGCUUUAAUUGGUACCGAGAAGGGGACUCGAACCCCUACACCCUAUGGGCACAACCACCUCAAGGUUGCGUGUCUACCAAUUCCACCACCUCGGCA .(((((((.-...))))))).....(((......))).(((((.(((((.......)))))........(((((((((((.......))))..))))))).............))))).. ( -40.40) >psp.2438 4780896 117 + 5928787 AAGCCCGCU-AAUGCGGGCUCCCUUGU--UAAUUGGUACCGAGAAGGGGACUCGAACCCCUACACCCAUUGGGCACAACCACCUCAAGGUUGCGUGUCUACCAAUUCCACCACCUCGGCA .(((((((.-...))))))).......--.........(((((.(((((.......)))))........(((((((((((.......))))..))))))).............))))).. ( -40.10) >pst.2667 5061432 117 + 6397126 AAGCCCGCU-AAUGCGGGCUUCCUUGU--UAAUUGGUACCGAGAAGGGGACUCGAACCCCUACACCCAUUGGGCACAACCACCUCAAGGUUGCGUGUCUACCAAUUCCACCACCUCGGCA ((((((((.-...))))))))......--.........(((((.(((((.......)))))........(((((((((((.......))))..))))))).............))))).. ( -40.70) >consensus AAGCCCGCU_AAAGCGGGCUUCCUUG___UAAUUGGUACCGAGAAGGGGACUCGAACCCCUACACCCUAUGGGCACAACCACCUCAAGGUUGCGUGUCUACCAAUUCCACCACCUCGGCA ((((((((.....)))))))).................(((((.(((((.......)))))........(((((((((((.......))))..))))))).............))))).. (-33.78 = -35.00 + 1.22)

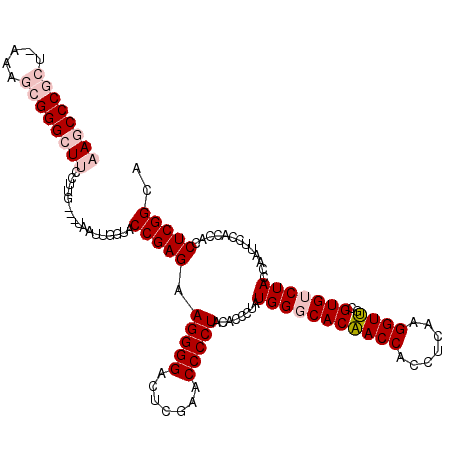
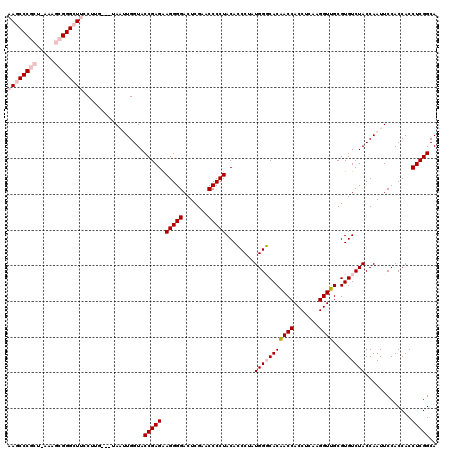
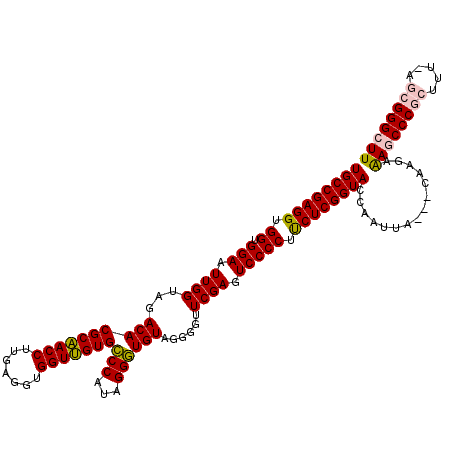
| Location | 5,332,227 – 5,332,341 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.00 |
| Mean single sequence MFE | -48.70 |
| Consensus MFE | -45.03 |
| Energy contribution | -44.70 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.22 |
| Structure conservation index | 0.92 |
| SVM decision value | 2.14 |
| SVM RNA-class probability | 0.988824 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2817 5332227 114 - 6264404 UGCCGAGGUGGUGGAAUUGGUAGACACGCUACCUUGAGGUGGUAGUGGCCAUAGGCUGUAGGGGUUCGAGUCCCCUCCUCGGUACCAUAUA-----AGCAAGCCCCGUCC-UGCGGGGUU ((((((((.((.(((.((((...((((((((((.......)))))))(((...))))))......)))).))))).)))))))).......-----....(((((((...-..))))))) ( -49.30) >pau.2955 5604156 114 - 6537648 UGCCGAGGUGGUGGAAUUGGUAGACACGCUACCUUGAGGUGGUAGUGGCCAUAGGCUGUAGGGGUUCGAGUCCCCUCCUCGGUACCAUAUA-----AGCAAGCCCCGUCC-UGCGGGGUU ((((((((.((.(((.((((...((((((((((.......)))))))(((...))))))......)))).))))).)))))))).......-----....(((((((...-..))))))) ( -49.30) >pel.398 5066372 120 + 5888780 UGCCGAGGUGGUGGAAUUGGUAGACACGCAACCUUGAGGUGGUUGUGCCCAUAGGGUGUAGGGGUUCGAGUCCCCUUCUCGGUACCAAUUGAAGCAUGAAAGCCCGCUUUGAGCGGGCUU ((((((((.((.(((.((((......(((((((.......)))))))(((..........)))..)))).))))).))))))))...............((((((((.....)))))))) ( -48.00) >ppk.2732 5361059 119 - 6181863 UGCCGAGGUGGUGGAAUUGGUAGACACGCAACCUUGAGGUGGUUGUGCCCAUAGGGUGUAGGGGUUCGAGUCCCCUUCUCGGUACCAAUUAAAGCUUGAGAGCCCGCUUU-AGCGGGCUU ((((((((.((.(((.((((......(((((((.......)))))))(((..........)))..)))).))))).))))))))...............((((((((...-.)))))))) ( -48.40) >psp.2438 4780896 117 - 5928787 UGCCGAGGUGGUGGAAUUGGUAGACACGCAACCUUGAGGUGGUUGUGCCCAAUGGGUGUAGGGGUUCGAGUCCCCUUCUCGGUACCAAUUA--ACAAGGGAGCCCGCAUU-AGCGGGCUU ((((((((.((.(((.((((......(((((((.......)))))))(((.((....)).)))..)))).))))).))))))))((.....--....))((((((((...-.)))))))) ( -48.90) >pst.2667 5061432 117 - 6397126 UGCCGAGGUGGUGGAAUUGGUAGACACGCAACCUUGAGGUGGUUGUGCCCAAUGGGUGUAGGGGUUCGAGUCCCCUUCUCGGUACCAAUUA--ACAAGGAAGCCCGCAUU-AGCGGGCUU ((((((((.((.(((.((((......(((((((.......)))))))(((.((....)).)))..)))).))))).)))))))).......--......((((((((...-.)))))))) ( -48.30) >consensus UGCCGAGGUGGUGGAAUUGGUAGACACGCAACCUUGAGGUGGUUGUGCCCAUAGGGUGUAGGGGUUCGAGUCCCCUUCUCGGUACCAAUUA___CAAGAAAGCCCGCUUU_AGCGGGCUU ((((((((.((.(((.((((...((((((((((.......)))))))(((...))))))......)))).))))).))))))))...............((((((((.....)))))))) (-45.03 = -44.70 + -0.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:11:42 2007