
| Sequence ID | pao.2532 |
|---|---|
| Location | 4,756,848 – 4,756,978 |
| Length | 130 |
| Max. P | 0.994688 |

| Location | 4,756,848 – 4,756,965 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.99 |
| Mean single sequence MFE | -40.43 |
| Consensus MFE | -33.84 |
| Energy contribution | -33.79 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.09 |
| Mean z-score | -4.32 |
| Structure conservation index | 0.84 |
| SVM decision value | 2.35 |
| SVM RNA-class probability | 0.992821 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
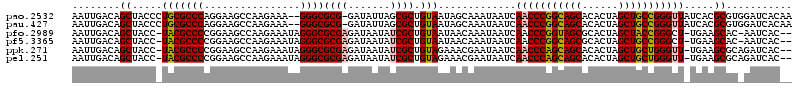
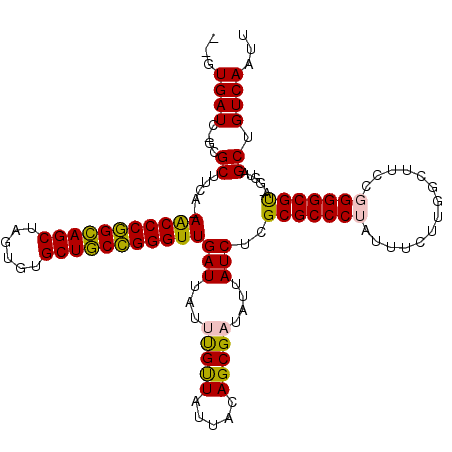
>pao.2532 4756848 117 + 6264404 UCAGAAAAACUCCAAUUGACAGCUACCCUGCGCCCAGGAAGCCAAGAAA--GGGCGCG-GAUAUUAGCGCUGUAAUAGCAAAUAAUCAACCCGGCAGCACACUAGCUGCCGGGUUAUCAC ...((................((((..((((((((..............--)))))))-)....))))((((...))))......))(((((((((((......)))))))))))..... ( -44.54) >pau.427 5767678 117 - 6537648 UCAGAAAAACUCCAAUUGACAGCUACCCUGCGCCCAGGAAGCCAAGAAA--GGGCGCG-GAUAUUAGCGCUGUAAUAGCAAAUAAUCAACCCGGCAGCACACUAGCUGCCGGGUUAUCAC ...((................((((..((((((((..............--)))))))-)....))))((((...))))......))(((((((((((......)))))))))))..... ( -44.54) >pfo.2989 5694818 110 + 6438405 --------ACUCCAAUUGACAGCUACC-UACGCCCCGGAAGCCAAGAAAUAGGGCGCGAGAUAAUAUCGCUGUAAUAACAAAUAAUCAACCCGGUAGCGCACUAGCUACCGGGCU-UGAA --------..........(((((....-..((((((...............))).))).(((...))))))))............(((((((((((((......))))))))).)-))). ( -37.76) >pf5.3365 6351746 110 + 7074893 --------ACUCCAAUUGACAGCUACC-UACGCCCCGGAAGCCAAGAAAUAGGGCGCGAGAUAAUAUCGCUGUAAUAACAAAUAAUCAACCCGGCAGCGCACUAGCUGCCGGGCU-UGAA --------..........(((((....-..((((((...............))).))).(((...))))))))............(((((((((((((......))))))))).)-))). ( -39.76) >ppk.271 5620379 110 - 6181863 --------ACUCCAAUUGACAGCUACC-UACGCCCCGGAAGCCAAGAAAUAGGGCGCGAGAUAAUAUCGCUGUAGAAACGAAUAAUCAACCCAGCAGCACACUAGCUGCUGGGUU-UGAA --------..................(-((((((((....)..........))))((((.......)))).))))..........(((((((((((((......))))))))).)-))). ( -38.00) >pel.251 5361940 110 - 5888780 --------ACUCCAAUUGACAGCUACC-UACGCCCCGGAAGCCAAGAAAUAGGGCGCGAGAUAAUAUCGCUGUAGAAACGAAUAAUCAACCCAGCAGCACACUAGCUGCUGGGUU-UGAA --------..................(-((((((((....)..........))))((((.......)))).))))..........(((((((((((((......))))))))).)-))). ( -38.00) >consensus ________ACUCCAAUUGACAGCUACC_UACGCCCCGGAAGCCAAGAAAUAGGGCGCGAGAUAAUAUCGCUGUAAUAACAAAUAAUCAACCCGGCAGCACACUAGCUGCCGGGUU_UGAA ..................(((((.......(((((................))))).((.......)))))))..............(((((((((((......)))))))))))..... (-33.84 = -33.79 + -0.05)

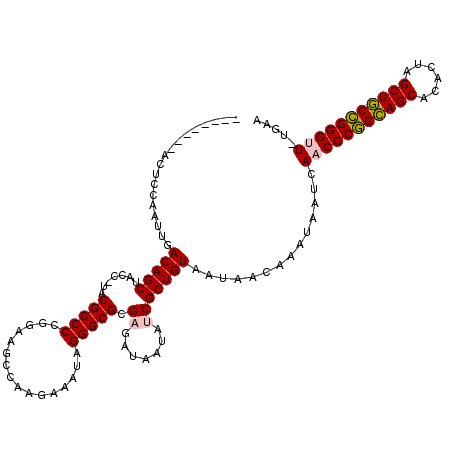
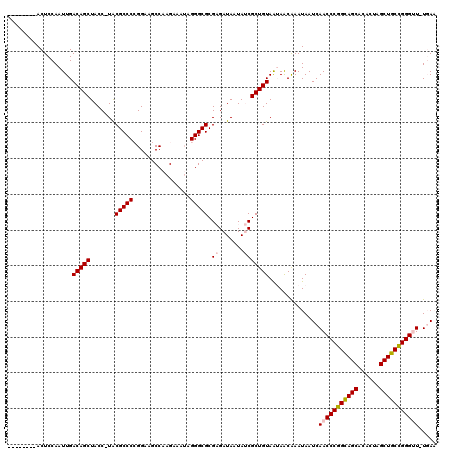
| Location | 4,756,848 – 4,756,965 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 86.99 |
| Mean single sequence MFE | -44.31 |
| Consensus MFE | -38.90 |
| Energy contribution | -38.07 |
| Covariance contribution | -0.83 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.52 |
| Structure conservation index | 0.88 |
| SVM decision value | 2.50 |
| SVM RNA-class probability | 0.994688 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2532 4756848 117 - 6264404 GUGAUAACCCGGCAGCUAGUGUGCUGCCGGGUUGAUUAUUUGCUAUUACAGCGCUAAUAUC-CGCGCCC--UUUCUUGGCUUCCUGGGCGCAGGGUAGCUGUCAAUUGGAGUUUUUCUGA ((((((((((((((((......))))))))))).)))))...(((..(((((.....((((-(((((((--..............)))))).))))))))))....)))........... ( -47.94) >pau.427 5767678 117 + 6537648 GUGAUAACCCGGCAGCUAGUGUGCUGCCGGGUUGAUUAUUUGCUAUUACAGCGCUAAUAUC-CGCGCCC--UUUCUUGGCUUCCUGGGCGCAGGGUAGCUGUCAAUUGGAGUUUUUCUGA ((((((((((((((((......))))))))))).)))))...(((..(((((.....((((-(((((((--..............)))))).))))))))))....)))........... ( -47.94) >pfo.2989 5694818 110 - 6438405 UUCA-AGCCCGGUAGCUAGUGCGCUACCGGGUUGAUUAUUUGUUAUUACAGCGAUAUUAUCUCGCGCCCUAUUUCUUGGCUUCCGGGGCGUA-GGUAGCUGUCAAUUGGAGU-------- ((((-(((((((((((......)))))))))))..............(((((.....(((((.(((((((..............))))))))-)))))))))....))))..-------- ( -41.04) >pf5.3365 6351746 110 - 7074893 UUCA-AGCCCGGCAGCUAGUGCGCUGCCGGGUUGAUUAUUUGUUAUUACAGCGAUAUUAUCUCGCGCCCUAUUUCUUGGCUUCCGGGGCGUA-GGUAGCUGUCAAUUGGAGU-------- ((((-(((((((((((......)))))))))))..............(((((.....(((((.(((((((..............))))))))-)))))))))....))))..-------- ( -43.04) >ppk.271 5620379 110 + 6181863 UUCA-AACCCAGCAGCUAGUGUGCUGCUGGGUUGAUUAUUCGUUUCUACAGCGAUAUUAUCUCGCGCCCUAUUUCUUGGCUUCCGGGGCGUA-GGUAGCUGUCAAUUGGAGU-------- .(((-(.(((((((((......)))))))))))))...((((.....(((((.....(((((.(((((((..............))))))))-)))))))))....))))..-------- ( -42.94) >pel.251 5361940 110 + 5888780 UUCA-AACCCAGCAGCUAGUGUGCUGCUGGGUUGAUUAUUCGUUUCUACAGCGAUAUUAUCUCGCGCCCUAUUUCUUGGCUUCCGGGGCGUA-GGUAGCUGUCAAUUGGAGU-------- .(((-(.(((((((((......)))))))))))))...((((.....(((((.....(((((.(((((((..............))))))))-)))))))))....))))..-------- ( -42.94) >consensus UUCA_AACCCGGCAGCUAGUGUGCUGCCGGGUUGAUUAUUUGUUAUUACAGCGAUAUUAUCUCGCGCCCUAUUUCUUGGCUUCCGGGGCGUA_GGUAGCUGUCAAUUGGAGU________ .....(((((((((((......)))))))))))..............(((((.....((((..(((((((..............)))))))..))))))))).................. (-38.90 = -38.07 + -0.83)

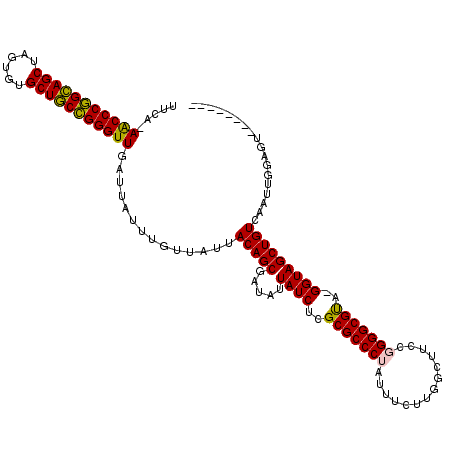
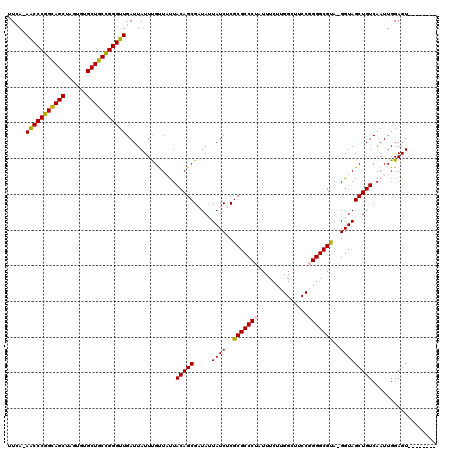
| Location | 4,756,861 – 4,756,978 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 87.92 |
| Mean single sequence MFE | -43.17 |
| Consensus MFE | -33.93 |
| Energy contribution | -33.66 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.12 |
| Mean z-score | -4.37 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.94 |
| SVM RNA-class probability | 0.983465 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
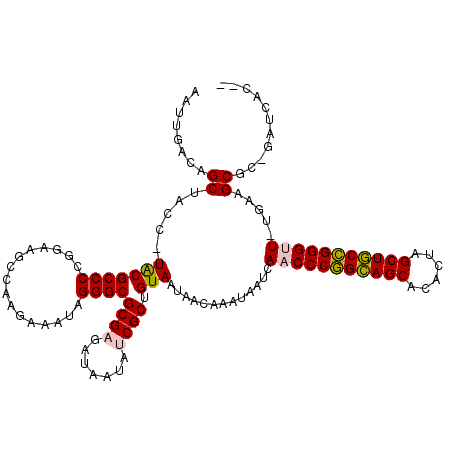
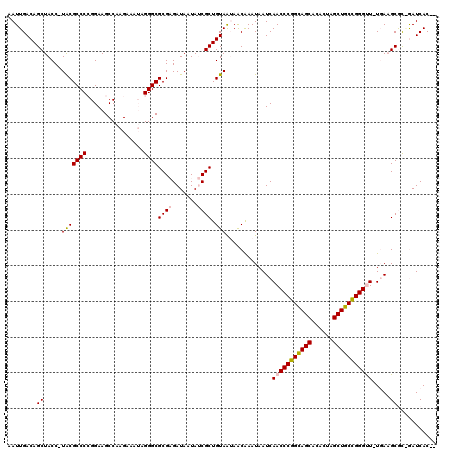
>pao.2532 4756861 117 + 6264404 AAUUGACAGCUACCCUGCGCCCAGGAAGCCAAGAAA--GGGCGCG-GAUAUUAGCGCUGUAAUAGCAAAUAAUCAACCCGGCAGCACACUAGCUGCCGGGUUAUCACGCGUGGAUCACAA ...(((...((((.((((((((..............--)))))))-)......((((((...))))........(((((((((((......))))))))))).....)))))).)))... ( -47.54) >pau.427 5767691 117 - 6537648 AAUUGACAGCUACCCUGCGCCCAGGAAGCCAAGAAA--GGGCGCG-GAUAUUAGCGCUGUAAUAGCAAAUAAUCAACCCGGCAGCACACUAGCUGCCGGGUUAUCACGCGUGGAUCACAA ...(((...((((.((((((((..............--)))))))-)......((((((...))))........(((((((((((......))))))))))).....)))))).)))... ( -47.54) >pfo.2989 5694823 115 + 6438405 AAUUGACAGCUACC-UACGCCCCGGAAGCCAAGAAAUAGGGCGCGAGAUAAUAUCGCUGUAAUAACAAAUAAUCAACCCGGUAGCGCACUAGCUACCGGGCU-UGAAGCAC-AAUCAC-- .(((((((((....-..((((((...............))).))).(((...))))))))............(((((((((((((......))))))))).)-)))....)-)))...-- ( -38.76) >pf5.3365 6351751 115 + 7074893 AAUUGACAGCUACC-UACGCCCCGGAAGCCAAGAAAUAGGGCGCGAGAUAAUAUCGCUGUAAUAACAAAUAAUCAACCCGGCAGCGCACUAGCUGCCGGGCU-UGAAGCAC-AAUCAC-- .(((((((((....-..((((((...............))).))).(((...))))))))............(((((((((((((......))))))))).)-)))....)-)))...-- ( -40.76) >ppk.271 5620384 116 - 6181863 AAUUGACAGCUACC-UACGCCCCGGAAGCCAAGAAAUAGGGCGCGAGAUAAUAUCGCUGUAGAAACGAAUAAUCAACCCAGCAGCACACUAGCUGCUGGGUU-UGAAGCGCAGAUCAC-- ..(((...(((..(-((((((((....)..........))))((((.......)))).))))..........(((((((((((((......))))))))).)-)))))).))).....-- ( -42.20) >pel.251 5361945 116 - 5888780 AAUUGACAGCUACC-UACGCCCCGGAAGCCAAGAAAUAGGGCGCGAGAUAAUAUCGCUGUAGAAACGAAUAAUCAACCCAGCAGCACACUAGCUGCUGGGUU-UGAAGCGCAGAUCAC-- ..(((...(((..(-((((((((....)..........))))((((.......)))).))))..........(((((((((((((......))))))))).)-)))))).))).....-- ( -42.20) >consensus AAUUGACAGCUACC_UACGCCCCGGAAGCCAAGAAAUAGGGCGCGAGAUAAUAUCGCUGUAAUAACAAAUAAUCAACCCGGCAGCACACUAGCUGCCGGGUU_UGAAGCGC_GAUCAC__ ........((.....(((((((................))))((((.......)))).))).............(((((((((((......))))))))))).....))........... (-33.93 = -33.66 + -0.28)
| Location | 4,756,861 – 4,756,978 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.92 |
| Mean single sequence MFE | -46.66 |
| Consensus MFE | -39.98 |
| Energy contribution | -39.04 |
| Covariance contribution | -0.94 |
| Combinations/Pair | 1.19 |
| Mean z-score | -3.78 |
| Structure conservation index | 0.86 |
| SVM decision value | 2.49 |
| SVM RNA-class probability | 0.994558 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
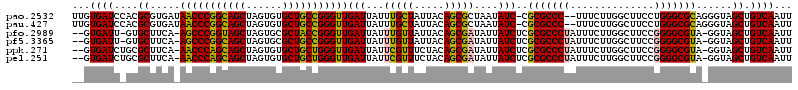
>pao.2532 4756861 117 - 6264404 UUGUGAUCCACGCGUGAUAACCCGGCAGCUAGUGUGCUGCCGGGUUGAUUAUUUGCUAUUACAGCGCUAAUAUC-CGCGCCC--UUUCUUGGCUUCCUGGGCGCAGGGUAGCUGUCAAUU ..(((...)))((((((((((((((((((......))))))))))).)))))..))....(((((.....((((-(((((((--..............)))))).))))))))))..... ( -49.34) >pau.427 5767691 117 + 6537648 UUGUGAUCCACGCGUGAUAACCCGGCAGCUAGUGUGCUGCCGGGUUGAUUAUUUGCUAUUACAGCGCUAAUAUC-CGCGCCC--UUUCUUGGCUUCCUGGGCGCAGGGUAGCUGUCAAUU ..(((...)))((((((((((((((((((......))))))))))).)))))..))....(((((.....((((-(((((((--..............)))))).))))))))))..... ( -49.34) >pfo.2989 5694823 115 - 6438405 --GUGAUU-GUGCUUCA-AGCCCGGUAGCUAGUGCGCUACCGGGUUGAUUAUUUGUUAUUACAGCGAUAUUAUCUCGCGCCCUAUUUCUUGGCUUCCGGGGCGUA-GGUAGCUGUCAAUU --(((((.-..((.(((-(.(((((((((......)))))))))))))......)).)))))...((((((((((.(((((((..............))))))))-))))).)))).... ( -43.14) >pf5.3365 6351751 115 - 7074893 --GUGAUU-GUGCUUCA-AGCCCGGCAGCUAGUGCGCUGCCGGGUUGAUUAUUUGUUAUUACAGCGAUAUUAUCUCGCGCCCUAUUUCUUGGCUUCCGGGGCGUA-GGUAGCUGUCAAUU --(((((.-..((.(((-(.(((((((((......)))))))))))))......)).)))))...((((((((((.(((((((..............))))))))-))))).)))).... ( -45.14) >ppk.271 5620384 116 + 6181863 --GUGAUCUGCGCUUCA-AACCCAGCAGCUAGUGUGCUGCUGGGUUGAUUAUUCGUUUCUACAGCGAUAUUAUCUCGCGCCCUAUUUCUUGGCUUCCGGGGCGUA-GGUAGCUGUCAAUU --((.(((((((((((.-(((((((((((......))))))))))).................((((.......))))(((.........)))....))))))))-))).))........ ( -46.50) >pel.251 5361945 116 + 5888780 --GUGAUCUGCGCUUCA-AACCCAGCAGCUAGUGUGCUGCUGGGUUGAUUAUUCGUUUCUACAGCGAUAUUAUCUCGCGCCCUAUUUCUUGGCUUCCGGGGCGUA-GGUAGCUGUCAAUU --((.(((((((((((.-(((((((((((......))))))))))).................((((.......))))(((.........)))....))))))))-))).))........ ( -46.50) >consensus __GUGAUC_GCGCUUCA_AACCCGGCAGCUAGUGUGCUGCCGGGUUGAUUAUUUGUUAUUACAGCGAUAUUAUCUCGCGCCCUAUUUCUUGGCUUCCGGGGCGUA_GGUAGCUGUCAAUU ...((((....((.....(((((((((((......)))))))))))(((...(((((.....)))))....)))..(((((((..............)))))))......)).))))... (-39.98 = -39.04 + -0.94)
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:11:06 2007