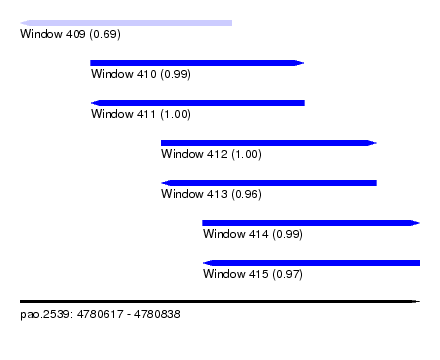
| Sequence ID | pao.2539 |
|---|---|
| Location | 4,780,617 – 4,780,838 |
| Length | 221 |
| Max. P | 0.996816 |

| Location | 4,780,617 – 4,780,734 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.90 |
| Mean single sequence MFE | -46.25 |
| Consensus MFE | -32.09 |
| Energy contribution | -31.82 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.32 |
| SVM RNA-class probability | 0.687568 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2539 4780617 117 - 6264404 CCACCCAAGACGUGAAAG-UGCCCCGAGUGGGCAAUGCAGGCCAAGGGGUCUGCAAGAUUUUCCGGCUGCUCCCGUC-GGAGAC-GCCAAACAAGCAGGUGACCAAGCUGGGGAACGCUG ...........(((....-(((((.....))))).((((((((....))))))))...((..((((((.((((....-)))).(-(((.........))))....))))))..))))).. ( -44.40) >pfo.2996 5718669 116 - 6438405 GUCGAUAACGAGCUGUAA-CCACCCGAUGCGGUGGCGCAAACCAUGGGGUUUGCACGAUUUUCUGGCUGCUCCCGUCGGGAGGG-GCCAAACAAGCAGGUGACCAAGCUGGGGAACGC-- ..((.(..(.((((....-.((((...((((((.(.(((((((....))))))).).)))...(((((.(((((...))))).)-)))).....)))))))....)))).)..).)).-- ( -45.00) >pf5.3372 6375607 117 - 7074893 GCCAGUAACGUGCUGUAGCCCACCCUGUGCGGUGGCGCAAACCAAAGGGUUUGCACGAUUUUCUGGCUGCUCCCGUCGGGAGGA-GCCGAAUAAGCAGGUGACCAAGCUGGGGAACGC-- .(((((.((.((((((.(((.(((......))))))(((((((....)))))))))....(((.((((.(((((...))))).)-))))))..)))).))......))))).......-- ( -48.90) >ppk.264 5644245 117 + 6181863 CCCGAUAACGAGCUGCAAGACACCCAUGUGGGUGGCGCAAACCAAGGGGUUUGCACGAUUUUCUGGCUGCUCCCGCCGGGAGAA-GCCAAACAAGCAGGUGACCAAGCUGGGGAACGC-- ((((........((((.((((((((....)))))(.(((((((....))))))).).....)))((((.(((((...))))).)-)))......))))((......))))))......-- ( -45.60) >pel.244 5385809 115 + 5888780 CCCGAUAACGAGCGGCA--ACCACCAAGACGGUGGCGCAAACCAAGGGGUUUGCACGAUUUUCUGGCUGCUCCCGCCGGGAGAU-GCCAAACAAGCAGGUGACCAAGCUGGGGAAUGC-- (((..........(((.--.(((((.....))))).(((((((....)))))))...((((((((((.......))))))))))-))).....(((.(....)...))))))......-- ( -43.90) >pss.2756 5416504 117 - 6093698 GUCUGUAACGUGUGGUAU-CCACCGAUGGCGGUGGCGCAAACCAAGGGGUUUGCAAGAUUUUCUGGCUGCUUCCGUCGGGAGGGAGCCAAACAAGCAGGUGACCAAGCUGGGGAACGC-- ..((((...((.((((.(-((.(((((((.((..(((((((((....))))))).((.....)).))..)).)))))))..))).)))).))..))))((..((......))..))..-- ( -49.70) >consensus CCCGAUAACGAGCGGUAA_CCACCCAAGGCGGUGGCGCAAACCAAGGGGUUUGCACGAUUUUCUGGCUGCUCCCGUCGGGAGAA_GCCAAACAAGCAGGUGACCAAGCUGGGGAACGC__ ......................((((....(((.(((((((((....))))))).........((((..((((.....))))...)))).........)).)))....))))........ (-32.09 = -31.82 + -0.27)
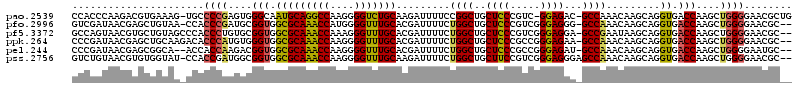
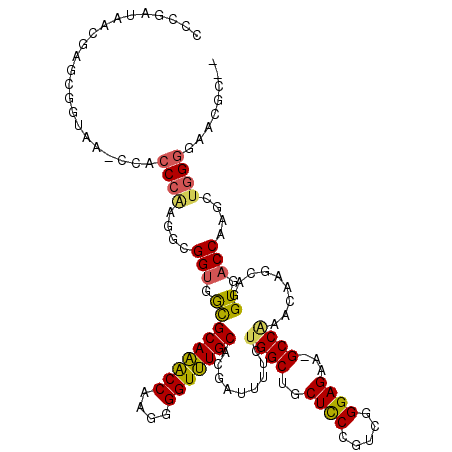
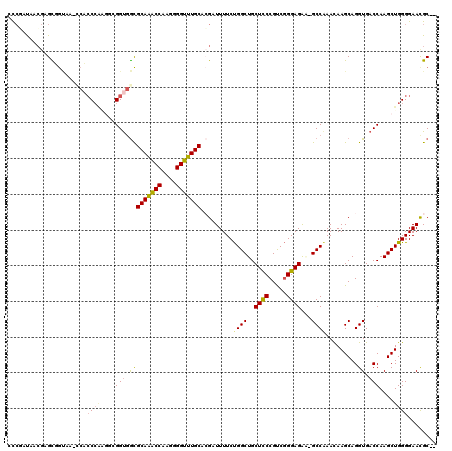
| Location | 4,780,656 – 4,780,774 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.93 |
| Mean single sequence MFE | -50.35 |
| Consensus MFE | -27.15 |
| Energy contribution | -28.15 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.90 |
| Structure conservation index | 0.54 |
| SVM decision value | 2.47 |
| SVM RNA-class probability | 0.994345 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2539 4780656 118 + 6264404 CC-GACGGGAGCAGCCGGAAAAUCUUGCAGACCCCUUGGCCUGCAUUGCCCACUCGGGGCA-CUUUCACGUCUUGGGUGGAACGCCCGAGGCGAGUCCACCCAGAACGGAAAAAGGCCGG ((-...(((..(.((.((.....)).)).)..)))..(((((....(((((.....)))))-.((((.(((..((((((((.((((...))))..))))))))..))))))).))))))) ( -54.10) >pfo.2996 5718706 117 + 6438405 CCCGACGGGAGCAGCCAGAAAAUCGUGCAAACCCCAUGGUUUGCGCCACCGCAUCGGGUGG-UUACAGCUCGUUAUCGACACCGACCCAGUCGG--CUGCCAAUAACGGAAAAAGGCCGG .....(((((((..............(((((((....)))))))((((((......)))))-)....))))(((((.(.(((((((...)))))--.)).).))))).........))). ( -41.60) >pf5.3372 6375644 118 + 7074893 CCCGACGGGAGCAGCCAGAAAAUCGUGCAAACCCUUUGGUUUGCGCCACCGCACAGGGUGGGCUACAGCACGUUACUGGCACCGACCCAGUCGG--UCGCCAAUAACGGAAAAAGGCCGG .....(((..((((((........(((((((((....)))))))))((((......))))))))...)).(((((.((((((((((...)))))--).)))).)))))........))). ( -51.10) >ppk.264 5644282 118 - 6181863 CCCGGCGGGAGCAGCCAGAAAAUCGUGCAAACCCCUUGGUUUGCGCCACCCACAUGGGUGUCUUGCAGCUCGUUAUCGGGACCGGCCUGACCGG--CCACCAAUAACGGAAAAAGGCCGG (((((..(((((.((.(((.....(((((((((....)))))))))(((((....)))))))).)).)))).)..))))).(((((((..(((.--..........)))....))))))) ( -57.90) >pel.244 5385846 116 - 5888780 CCCGGCGGGAGCAGCCAGAAAAUCGUGCAAACCCCUUGGUUUGCGCCACCGUCUUGGUGGU--UGCCGCUCGUUAUCGGGGCCGACCUGAUCGG--CCACCAAUAACGGAAAAAGGCCGG .(((((((.(((.((((((.....(((((((((....))))))))).....)).)))).))--).))..(((((((.((((((((.....))))--)).)).)))))))......))))) ( -57.30) >pss.2756 5416542 117 + 6093698 CCCGACGGAAGCAGCCAGAAAAUCUUGCAAACCCCUUGGUUUGCGCCACCGCCAUCGGUGG-AUACCACACGUUACAGACGUUGACAGCGCCAA--UCGUCUAUAACGGAAAAAGGCCGG .(((.(....)..(((..........(((((((....))))))).((((((....))))))-........(((((.((((((((.......)))--.))))).)))))......)))))) ( -40.10) >consensus CCCGACGGGAGCAGCCAGAAAAUCGUGCAAACCCCUUGGUUUGCGCCACCGCCUUGGGUGG_CUACAGCUCGUUAUCGGCACCGACCCAGUCGG__CCACCAAUAACGGAAAAAGGCCGG .(((..(....).(((........(((((((((....)))))))))((((......))))..........(((((.(((..((((.....)))).....))).)))))......)))))) (-27.15 = -28.15 + 1.00)

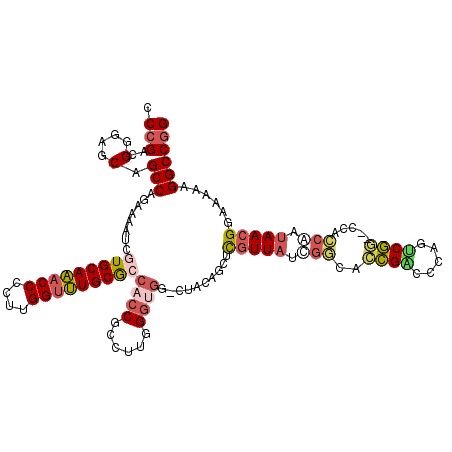
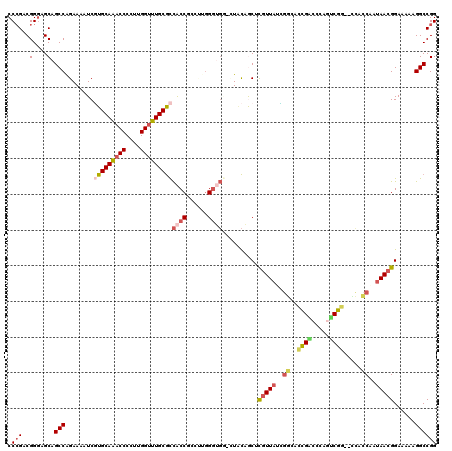
| Location | 4,780,656 – 4,780,774 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 75.93 |
| Mean single sequence MFE | -57.80 |
| Consensus MFE | -37.46 |
| Energy contribution | -36.38 |
| Covariance contribution | -1.08 |
| Combinations/Pair | 1.51 |
| Mean z-score | -3.70 |
| Structure conservation index | 0.65 |
| SVM decision value | 2.58 |
| SVM RNA-class probability | 0.995430 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2539 4780656 118 - 6264404 CCGGCCUUUUUCCGUUCUGGGUGGACUCGCCUCGGGCGUUCCACCCAAGACGUGAAAG-UGCCCCGAGUGGGCAAUGCAGGCCAAGGGGUCUGCAAGAUUUUCCGGCUGCUCCCGUC-GG .(((((.((((((((..((((((((..((((...)))).))))))))..))).)))))-(((((.....))))).((((((((....)))))))).........)))))........-.. ( -58.00) >pfo.2996 5718706 117 - 6438405 CCGGCCUUUUUCCGUUAUUGGCAG--CCGACUGGGUCGGUGUCGAUAACGAGCUGUAA-CCACCCGAUGCGGUGGCGCAAACCAUGGGGUUUGCACGAUUUUCUGGCUGCUCCCGUCGGG (((((........((((((((((.--(((((...)))))))))))))))((((.((..-(((((......))))).(((((((....)))))))...........)).))))..))))). ( -51.80) >pf5.3372 6375644 118 - 7074893 CCGGCCUUUUUCCGUUAUUGGCGA--CCGACUGGGUCGGUGCCAGUAACGUGCUGUAGCCCACCCUGUGCGGUGGCGCAAACCAAAGGGUUUGCACGAUUUUCUGGCUGCUCCCGUCGGG (((((.......((((((((((.(--(((((...))))))))))))))))....((((((((((......))))(.(((((((....))))))).)........))))))....))))). ( -56.20) >ppk.264 5644282 118 + 6181863 CCGGCCUUUUUCCGUUAUUGGUGG--CCGGUCAGGCCGGUCCCGAUAACGAGCUGCAAGACACCCAUGUGGGUGGCGCAAACCAAGGGGUUUGCACGAUUUUCUGGCUGCUCCCGCCGGG (((((........((((((((.((--((((.....))))))))))))))((((.((.((((((((....)))))(.(((((((....))))))).).....))).)).))))..))))). ( -63.40) >pel.244 5385846 116 + 5888780 CCGGCCUUUUUCCGUUAUUGGUGG--CCGAUCAGGUCGGCCCCGAUAACGAGCGGCA--ACCACCAAGACGGUGGCGCAAACCAAGGGGUUUGCACGAUUUUCUGGCUGCUCCCGCCGGG (((((........((((((((.((--(((((...)))))))))))))))(((((((.--.(((((.....))))).(((((((....)))))))...........)))))))..))))). ( -64.50) >pss.2756 5416542 117 - 6093698 CCGGCCUUUUUCCGUUAUAGACGA--UUGGCGCUGUCAACGUCUGUAACGUGUGGUAU-CCACCGAUGGCGGUGGCGCAAACCAAGGGGUUUGCAAGAUUUUCUGGCUGCUUCCGUCGGG ...(((.....((((((((((((.--(((((...)))))))))))))))).).)))..-...(((((((.((..(((((((((....))))))).((.....)).))..)).))))))). ( -52.90) >consensus CCGGCCUUUUUCCGUUAUUGGCGG__CCGACUAGGUCGGUCCCGAUAACGAGCGGUAA_CCACCCAAGGCGGUGGCGCAAACCAAGGGGUUUGCACGAUUUUCUGGCUGCUCCCGUCGGG (((((.......(((((((((.....(((((...)))))..))))))))).((.((...(((((......))))).(((((((....)))))))...........)).))....))))). (-37.46 = -36.38 + -1.08)

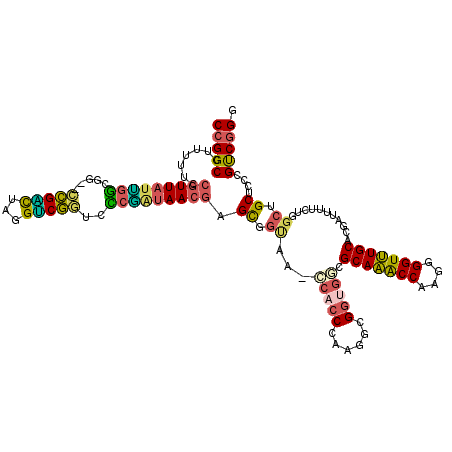
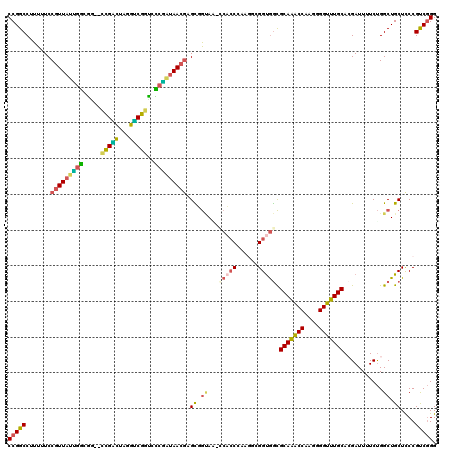
| Location | 4,780,695 – 4,780,814 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.59 |
| Mean single sequence MFE | -51.35 |
| Consensus MFE | -31.71 |
| Energy contribution | -32.80 |
| Covariance contribution | 1.09 |
| Combinations/Pair | 1.28 |
| Mean z-score | -3.87 |
| Structure conservation index | 0.62 |
| SVM decision value | 2.75 |
| SVM RNA-class probability | 0.996816 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
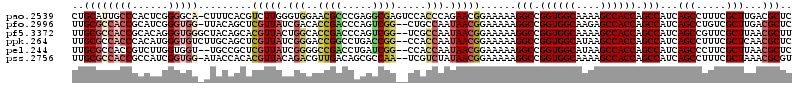
>pao.2539 4780695 119 + 6264404 CUGCAUUGCCCACUCGGGGCA-CUUUCACGUCUUGGGUGGAACGCCCGAGGCGAGUCCACCCAGAACGGAAAAAGGCCGGUGGCAAAAGCCACCAGCCAUCAGCCUUUCGCUUGACGCUC ..((..(((((.....)))))-.((((.(((..((((((((.((((...))))..))))))))..)))))))..(((.((((((....)))))).))).(((((.....)).))).)).. ( -57.00) >pfo.2996 5718746 117 + 6438405 UUGCGCCACCGCAUCGGGUGG-UUACAGCUCGUUAUCGACACCGACCCAGUCGG--CUGCCAAUAACGGAAAAAGGCCGGUGGCAAGAGCCACCAGCCAUCAGCCUGUCGCUUGACGCUC ..((((((((......)))))-)........((((.(((((..(((...)))((--(((((......)).....(((.((((((....)))))).)))..))))))))))..)))))).. ( -48.80) >pf5.3372 6375684 118 + 7074893 UUGCGCCACCGCACAGGGUGGGCUACAGCACGUUACUGGCACCGACCCAGUCGG--UCGCCAAUAACGGAAAAAGGCCGGUGGCAAAAGCCACCAGCCAUCAGCCGUUCGCUUAACGCUU ..((((((((......)))))((.((.((.(((((.((((((((((...)))))--).)))).)))))......(((.((((((....)))))).)))....)).))..))....))).. ( -55.70) >ppk.264 5644322 118 - 6181863 UUGCGCCACCCACAUGGGUGUCUUGCAGCUCGUUAUCGGGACCGGCCUGACCGG--CCACCAAUAACGGAAAAAGGCCGGUGGCAUAAGCCACCAGCCAUCAGCCUUUCGCUCAACGCUC ..(((.(((((....)))))..(((.((((((((((.(((.((((.....))))--.).)).))))))).....(((.((((((....)))))).)))...........))))))))).. ( -49.40) >pel.244 5385886 116 - 5888780 UUGCGCCACCGUCUUGGUGGU--UGCCGCUCGUUAUCGGGGCCGACCUGAUCGG--CCACCAAUAACGGAAAAAGGCCGGUGGCAUAAGCCACCAGCCAUCAGCCCUUCGCUUAACGCUC ..((((((((.....))))).--))).(((((((((.((((((((.....))))--)).)).))))))......(((.((((((....)))))).)))...))).....((.....)).. ( -52.00) >pss.2756 5416582 117 + 6093698 UUGCGCCACCGCCAUCGGUGG-AUACCACACGUUACAGACGUUGACAGCGCCAA--UCGUCUAUAACGGAAAAAGGCCGGUGGCAAAAGCCACCAGCCAUCAGCCUUUCGCUAAACGCGU ..(((((((((....))))))-........(((((.((((((((.......)))--.))))).)))))......(((.((((((....)))))).)))...(((.....)))...))).. ( -45.20) >consensus UUGCGCCACCGCCUUGGGUGG_CUACAGCUCGUUAUCGGCACCGACCCAGUCGG__CCACCAAUAACGGAAAAAGGCCGGUGGCAAAAGCCACCAGCCAUCAGCCUUUCGCUUAACGCUC ..((((((((......))))).........(((((.(((..((((.....)))).....))).)))))......(((.((((((....)))))).)))...(((.....)))...))).. (-31.71 = -32.80 + 1.09)
| Location | 4,780,695 – 4,780,814 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 75.59 |
| Mean single sequence MFE | -63.23 |
| Consensus MFE | -47.33 |
| Energy contribution | -45.95 |
| Covariance contribution | -1.38 |
| Combinations/Pair | 1.41 |
| Mean z-score | -5.13 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.50 |
| SVM RNA-class probability | 0.959717 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
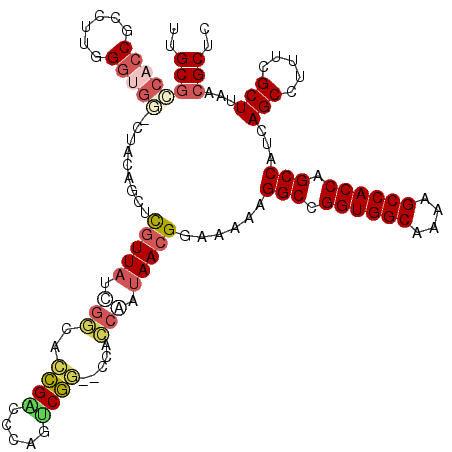
>pao.2539 4780695 119 - 6264404 GAGCGUCAAGCGAAAGGCUGAUGGCUGGUGGCUUUUGCCACCGGCCUUUUUCCGUUCUGGGUGGACUCGCCUCGGGCGUUCCACCCAAGACGUGAAAG-UGCCCCGAGUGGGCAAUGCAG ..((((((..(....)..))))((((((((((....)))))))))).((((((((..((((((((..((((...)))).))))))))..))).)))))-(((((.....)))))..)).. ( -65.30) >pfo.2996 5718746 117 - 6438405 GAGCGUCAAGCGACAGGCUGAUGGCUGGUGGCUCUUGCCACCGGCCUUUUUCCGUUAUUGGCAG--CCGACUGGGUCGGUGUCGAUAACGAGCUGUAA-CCACCCGAUGCGGUGGCGCAA ..((((((.(((...((.((..((((((((((....))))))))))......(((((((((((.--(((((...))))))))))))))))........-.)).))..)))..)))))).. ( -59.80) >pf5.3372 6375684 118 - 7074893 AAGCGUUAAGCGAACGGCUGAUGGCUGGUGGCUUUUGCCACCGGCCUUUUUCCGUUAUUGGCGA--CCGACUGGGUCGGUGCCAGUAACGUGCUGUAGCCCACCCUGUGCGGUGGCGCAA ..((.....))..(((((....((((((((((....))))))))))......((((((((((.(--(((((...)))))))))))))))).))))).(((((((......))))).)).. ( -65.00) >ppk.264 5644322 118 + 6181863 GAGCGUUGAGCGAAAGGCUGAUGGCUGGUGGCUUAUGCCACCGGCCUUUUUCCGUUAUUGGUGG--CCGGUCAGGCCGGUCCCGAUAACGAGCUGCAAGACACCCAUGUGGGUGGCGCAA ..(((((((((....((..((.((((((((((....)))))))))).))..))((((((((.((--((((.....))))))))))))))..))).))...(((((....))))))))).. ( -66.30) >pel.244 5385886 116 + 5888780 GAGCGUUAAGCGAAGGGCUGAUGGCUGGUGGCUUAUGCCACCGGCCUUUUUCCGUUAUUGGUGG--CCGAUCAGGUCGGCCCCGAUAACGAGCGGCA--ACCACCAAGACGGUGGCGCAA .........(((....((((..((((((((((....))))))))))......(((((((((.((--(((((...))))))))))))))))..)))).--.(((((.....)))))))).. ( -64.30) >pss.2756 5416582 117 - 6093698 ACGCGUUUAGCGAAAGGCUGAUGGCUGGUGGCUUUUGCCACCGGCCUUUUUCCGUUAUAGACGA--UUGGCGCUGUCAACGUCUGUAACGUGUGGUAU-CCACCGAUGGCGGUGGCGCAA .(((.....)))...((..((.((((((((((....)))))))))).))..))((((((((((.--(((((...))))))))))))))).((((....-((((((....)))))))))). ( -58.70) >consensus GAGCGUUAAGCGAAAGGCUGAUGGCUGGUGGCUUUUGCCACCGGCCUUUUUCCGUUAUUGGCGG__CCGACUAGGUCGGUCCCGAUAACGAGCGGUAA_CCACCCAAGGCGGUGGCGCAA ..((((...((....((..((.((((((((((....)))))))))).))..))((((((((.....(((((...)))))..))))))))..)).......((((......)))))))).. (-47.33 = -45.95 + -1.38)

| Location | 4,780,718 – 4,780,838 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.64 |
| Mean single sequence MFE | -50.53 |
| Consensus MFE | -35.87 |
| Energy contribution | -36.02 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.31 |
| Mean z-score | -3.75 |
| Structure conservation index | 0.71 |
| SVM decision value | 2.07 |
| SVM RNA-class probability | 0.987091 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
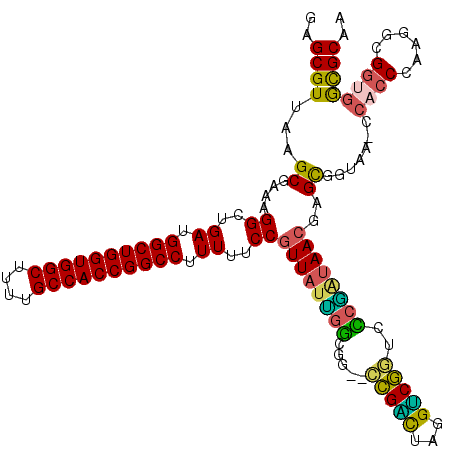
>pao.2539 4780718 120 + 6264404 UUCACGUCUUGGGUGGAACGCCCGAGGCGAGUCCACCCAGAACGGAAAAAGGCCGGUGGCAAAAGCCACCAGCCAUCAGCCUUUCGCUUGACGCUCGGGCUGUAGACGCAAGGUCGUCGC (((.(((..((((((((.((((...))))..))))))))..))))))...(((.((((((....)))))).)))..((((((...((.....))..))))))..((((......)))).. ( -58.80) >pst.391 5718741 114 - 6397126 ACAGCACGUUACAAACGUUGGUCGCGCCAA--CCGUCUAUAACGGAAAAAGGCCGGUGGCAAAAGCCACCAGCCAUCAGCCUUUCGCUAAACGCGUGGGCUGGACACUCAAAAUCG---- ......(((((.(.(((((((.....))))--.))).).)))))((....(((.((((((....)))))).))).(((((((..(((.....))).)))))))..........)).---- ( -44.30) >pfo.2996 5718769 114 + 6438405 ACAGCUCGUUAUCGACACCGACCCAGUCGG--CUGCCAAUAACGGAAAAAGGCCGGUGGCAAGAGCCACCAGCCAUCAGCCUGUCGCUUGACGCUCGGGCUGGAGGAGCAAAGUCG---- ...(((((((((.(.(((((((...)))))--.)).).))))).......(((.((((((....)))))).))).((((((((..((.....)).))))))))..)))).......---- ( -50.00) >pf5.3372 6375708 114 + 7074893 ACAGCACGUUACUGGCACCGACCCAGUCGG--UCGCCAAUAACGGAAAAAGGCCGGUGGCAAAAGCCACCAGCCAUCAGCCGUUCGCUUAACGCUUGGGCUGGAGACACAAAGUCG---- .((((.(((((.((((((((((...)))))--).)))).)))))......(((.((((((....)))))).))).((((.((((.....)))).))))))))..(((.....))).---- ( -51.30) >ppk.264 5644346 114 - 6181863 GCAGCUCGUUAUCGGGACCGGCCUGACCGG--CCACCAAUAACGGAAAAAGGCCGGUGGCAUAAGCCACCAGCCAUCAGCCUUUCGCUCAACGCUCAGGCUGGCAUCGCAAGGUCG---- ((...(((((((.(((.((((.....))))--.).)).))))))).....(((.((((((....)))))).)))..((((((...((.....))..))))))))(((....)))..---- ( -48.80) >pel.244 5385908 114 - 5888780 GCCGCUCGUUAUCGGGGCCGACCUGAUCGG--CCACCAAUAACGGAAAAAGGCCGGUGGCAUAAGCCACCAGCCAUCAGCCCUUCGCUUAACGCUCGGACUGGAGUCGCAAAGUCG---- ((.(((((((((.((((((((.....))))--)).)).))))).......(((.((((((....)))))).)))..(((.((...((.....))..)).))))))).)).......---- ( -50.00) >consensus ACAGCUCGUUAUCGGCACCGACCCAGUCGG__CCACCAAUAACGGAAAAAGGCCGGUGGCAAAAGCCACCAGCCAUCAGCCUUUCGCUUAACGCUCGGGCUGGAGACGCAAAGUCG____ ......(((((.(((..((((.....)))).....))).)))))((....(((.((((((....)))))).))).(((((((...((.....))..)))))))..........))..... (-35.87 = -36.02 + 0.14)

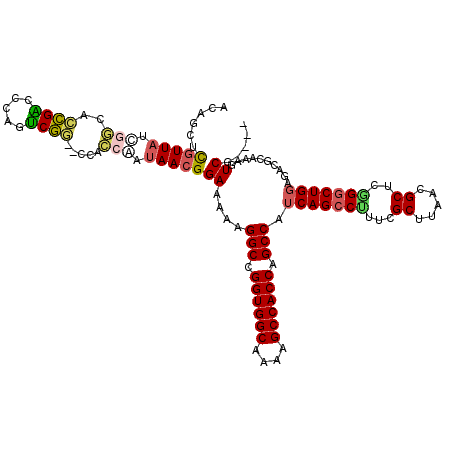
| Location | 4,780,718 – 4,780,838 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.64 |
| Mean single sequence MFE | -59.42 |
| Consensus MFE | -45.12 |
| Energy contribution | -43.65 |
| Covariance contribution | -1.47 |
| Combinations/Pair | 1.40 |
| Mean z-score | -4.75 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.966692 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2539 4780718 120 - 6264404 GCGACGACCUUGCGUCUACAGCCCGAGCGUCAAGCGAAAGGCUGAUGGCUGGUGGCUUUUGCCACCGGCCUUUUUCCGUUCUGGGUGGACUCGCCUCGGGCGUUCCACCCAAGACGUGAA ..((((......))))..(((((...((.....))....)))))..((((((((((....))))))))))...((((((..((((((((..((((...)))).))))))))..))).))) ( -64.70) >pst.391 5718741 114 + 6397126 ----CGAUUUUGAGUGUCCAGCCCACGCGUUUAGCGAAAGGCUGAUGGCUGGUGGCUUUUGCCACCGGCCUUUUUCCGUUAUAGACGG--UUGGCGCGACCAACGUUUGUAACGUGCUGU ----........(((...(((((..(((.....)))...)))))..((((((((((....))))))))))......(((((((((((.--((((.....))))))))))))))).))).. ( -51.90) >pfo.2996 5718769 114 - 6438405 ----CGACUUUGCUCCUCCAGCCCGAGCGUCAAGCGACAGGCUGAUGGCUGGUGGCUCUUGCCACCGGCCUUUUUCCGUUAUUGGCAG--CCGACUGGGUCGGUGUCGAUAACGAGCUGU ----.......((((...(((((.(.((.....))..).)))))..((((((((((....)))))))))).......((((((((((.--(((((...)))))))))))))))))))... ( -57.30) >pf5.3372 6375708 114 - 7074893 ----CGACUUUGUGUCUCCAGCCCAAGCGUUAAGCGAACGGCUGAUGGCUGGUGGCUUUUGCCACCGGCCUUUUUCCGUUAUUGGCGA--CCGACUGGGUCGGUGCCAGUAACGUGCUGU ----.(((.....)))..(((((...((.....))....)))))..((((((((((....))))))))))......((((((((((.(--(((((...))))))))))))))))...... ( -58.90) >ppk.264 5644346 114 + 6181863 ----CGACCUUGCGAUGCCAGCCUGAGCGUUGAGCGAAAGGCUGAUGGCUGGUGGCUUAUGCCACCGGCCUUUUUCCGUUAUUGGUGG--CCGGUCAGGCCGGUCCCGAUAACGAGCUGC ----.......((.....((((((..((.....))...))))))..((((((((((....))))))))))......(((((((((.((--((((.....))))))))))))))).))... ( -61.60) >pel.244 5385908 114 + 5888780 ----CGACUUUGCGACUCCAGUCCGAGCGUUAAGCGAAGGGCUGAUGGCUGGUGGCUUAUGCCACCGGCCUUUUUCCGUUAUUGGUGG--CCGAUCAGGUCGGCCCCGAUAACGAGCGGC ----.....((((.....((((((..((.....))...))))))..((((((((((....))))))))))......(((((((((.((--(((((...)))))))))))))))).)))). ( -62.10) >consensus ____CGACUUUGCGUCUCCAGCCCGAGCGUUAAGCGAAAGGCUGAUGGCUGGUGGCUUUUGCCACCGGCCUUUUUCCGUUAUUGGCGG__CCGACUAGGUCGGUCCCGAUAACGAGCUGU ..................(((((...((.....))....)))))..((((((((((....))))))))))......(((((((((.....((((.....))))..)))))))))...... (-45.12 = -43.65 + -1.47)



Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:10:53 2007