
| Sequence ID | pao.2541 |
|---|---|
| Location | 4,781,786 – 4,781,947 |
| Length | 161 |
| Max. P | 0.999748 |

| Location | 4,781,786 – 4,781,906 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 87.48 |
| Mean single sequence MFE | -45.52 |
| Consensus MFE | -38.17 |
| Energy contribution | -38.37 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.831812 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2541 4781786 120 + 6264404 GGGUUUACUCCUGGAUGUUACCGUUUCGUCCAGCCGGGGCCGGACGUCGUUUUGGUGUCUGAUUCGGUAACCGAAUCGGGAGCACCAUCUGCGUAGGCUUGUGGUUUAAGGCUUGCGCCA (((......)))((((.....((...(((((.((....)).))))).))....((((((((((((((...)))))))))..)))))))))((((((((((........)))))))))).. ( -49.70) >pau.418 5792615 120 - 6537648 GGGUUUACUCCUGGAUGUUACCGUUUCGUCCAGCCGGGGCCGGACGUCGUUUUGGUGUCUGAUUCGGUAACCGAAUCGGGAGCACCAUCUGCGUAGGCUUGUGGUUUAAGGCUUGCGCCA (((......)))((((.....((...(((((.((....)).))))).))....((((((((((((((...)))))))))..)))))))))((((((((((........)))))))))).. ( -49.70) >ppk.262 5645409 117 - 6181863 --GUUUACUCCUGGUUUUUACCGUUUCAUCUGGCCGAGGCCGGAUGUCGUUUUGGUGUCUGAUUCGGUAAC-GAAUCGGGAGCACCAUCUGCGUGGGCUGAUGUUUUAAGGCUUGCGCCG --..........(((....)))....((((((((....))))))))......((((((((((((((....)-)))))))..))))))...(((..((((..........))))..))).. ( -47.30) >pel.242 5386975 117 - 5888780 --GUUUACUCCUGGUUUCUACCGUUUCAUCUGGCCGGAGCCGGAUGUCGUUUUGGUGUCUGAUUCGCGAAC-GAAUCGGGAGCACCAUCUGCGUAGGCUUGAGGUUUAAGGCUUGCGCCG --..........(((....)))....((((((((....))))))))......((((((((((((((....)-)))))))..))))))...((((((((((........)))))))))).. ( -46.40) >pfo.2998 5719837 116 + 6438405 ---UUAACUCCUGCUUGUUACCGUUUCAUCCGAUCGAAACCGAAUGUCGUUUUGGUGUCUGAUUCGGUAAG-GAACCGGGAGCACCAUCUGCGUAGGCUUGAGGUUUAAGACUUGCGCCG ---....((((((.((.((((((..(((..(.((((((((........)))))))))..)))..)))))).-))..))))))........((((((((((((...))))).))))))).. ( -39.60) >pf5.3374 6376777 116 + 7074893 ---UUAACUCCUGCUUGUUACCGUUUCAUCCAACCGGAGCUGGAUGUCGUUUUGGUGUCUGAUUCGGUAAG-GAACCGGGAGCACCAUCUGCGUAGGCUUGAGGUUUAAGACUUGCGUCG ---....((((((.((.((((((..(((..((.(((((((........)))))))))..)))..)))))).-))..))))))........((((((((((((...))))).))))))).. ( -40.40) >consensus __GUUUACUCCUGGUUGUUACCGUUUCAUCCAGCCGGAGCCGGAUGUCGUUUUGGUGUCUGAUUCGGUAAC_GAAUCGGGAGCACCAUCUGCGUAGGCUUGAGGUUUAAGGCUUGCGCCG .......(((((((((.((((((..(((....((((((((........))))))))...)))..))))))...)))))))))........((((((((((........)))))))))).. (-38.17 = -38.37 + 0.20)

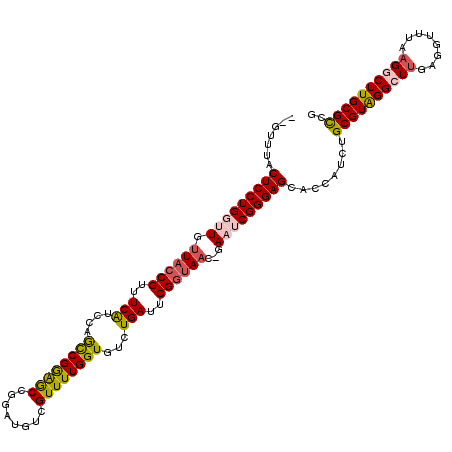
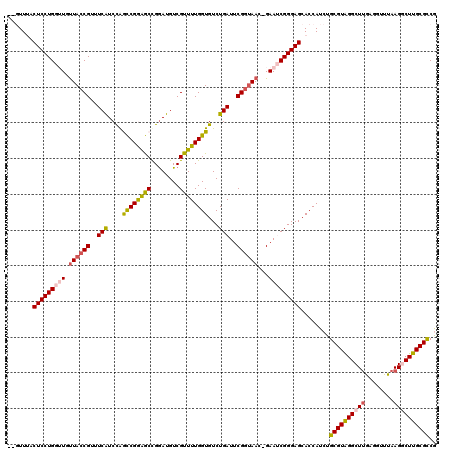
| Location | 4,781,786 – 4,781,906 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.48 |
| Mean single sequence MFE | -39.10 |
| Consensus MFE | -34.72 |
| Energy contribution | -33.00 |
| Covariance contribution | -1.72 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.79 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.18 |
| SVM RNA-class probability | 0.989709 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2541 4781786 120 - 6264404 UGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUCGGUUACCGAAUCAGACACCAAAACGACGUCCGGCCCCGGCUGGACGAAACGGUAACAUCCAGGAGUAAACCC .(((....)))....((((...((....))...))))(((((.(((((((...)))))))..............((((((((....))))))))....((......)).)))))...... ( -44.60) >pau.418 5792615 120 + 6537648 UGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUCGGUUACCGAAUCAGACACCAAAACGACGUCCGGCCCCGGCUGGACGAAACGGUAACAUCCAGGAGUAAACCC .(((....)))....((((...((....))...))))(((((.(((((((...)))))))..............((((((((....))))))))....((......)).)))))...... ( -44.60) >ppk.262 5645409 117 + 6181863 CGGCGCAAGCCUUAAAACAUCAGCCCACGCAGAUGGUGCUCCCGAUUC-GUUACCGAAUCAGACACCAAAACGACAUCCGGCCUCGGCCAGAUGAAACGGUAAAAACCAGGAGUAAAC-- .(((....)))......((((.((....)).)))).((((((.(((((-(....))))))..............((((.(((....))).))))....(((....))).))))))...-- ( -40.40) >pel.242 5386975 117 + 5888780 CGGCGCAAGCCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGAUUC-GUUCGCGAAUCAGACACCAAAACGACAUCCGGCUCCGGCCAGAUGAAACGGUAGAAACCAGGAGUAAAC-- .(((....)))(((..(((.....((((.....(((((.((..(((((-(....)))))).)))))))......((((.(((....))).)))).....)))).....)))..)))..-- ( -39.30) >pfo.2998 5719837 116 - 6438405 CGGCGCAAGUCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGGUUC-CUUACCGAAUCAGACACCAAAACGACAUUCGGUUUCGAUCGGAUGAAACGGUAACAAGCAGGAGUUAA--- .(((....)))....(((....((....))....)))(((((..(((.-.((((((.....(....).......((((((((....))))))))...))))))..))).)))))...--- ( -30.50) >pf5.3374 6376777 116 - 7074893 CGACGCAAGUCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGGUUC-CUUACCGAAUCAGACACCAAAACGACAUCCAGCUCCGGUUGGAUGAAACGGUAACAAGCAGGAGUUAA--- .(((....)))....(((....((....))....)))(((((..(((.-.((((((.....(....).......((((((((....))))))))...))))))..))).)))))...--- ( -35.20) >consensus CGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUC_GUUACCGAAUCAGACACCAAAACGACAUCCGGCCCCGGCCGGAUGAAACGGUAACAACCAGGAGUAAAC__ .(((....)))...........(((........(((((.((..(((((.......))))).)))))))......((((((((....))))))))....)))................... (-34.72 = -33.00 + -1.72)

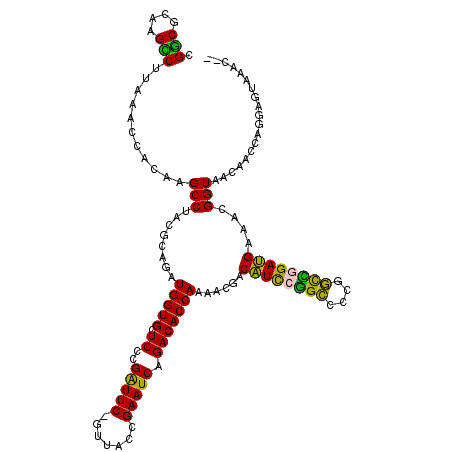
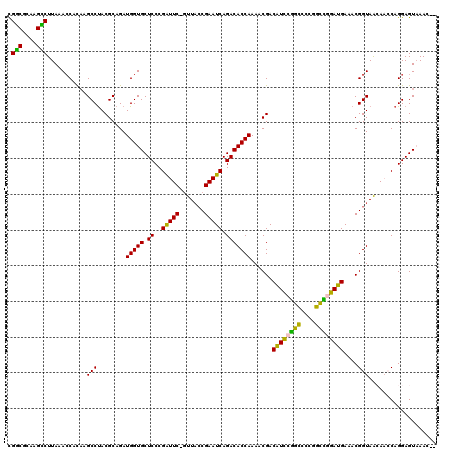
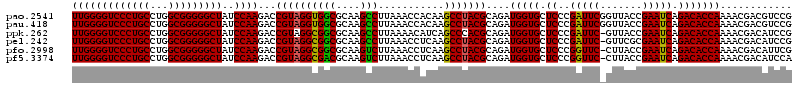
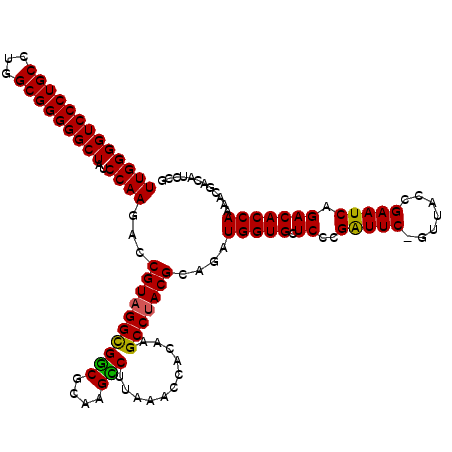
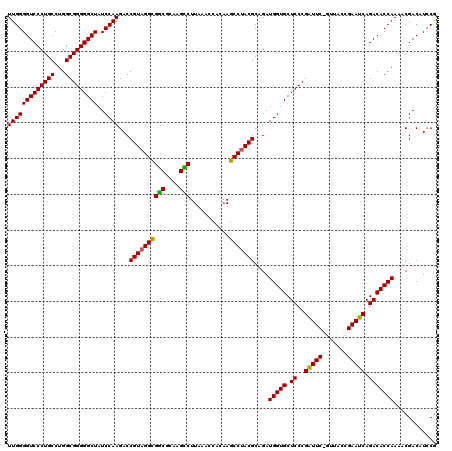
| Location | 4,781,826 – 4,781,946 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.31 |
| Mean single sequence MFE | -46.65 |
| Consensus MFE | -43.47 |
| Energy contribution | -42.83 |
| Covariance contribution | -0.64 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.93 |
| SVM decision value | 3.34 |
| SVM RNA-class probability | 0.999043 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2541 4781826 120 - 6264404 UUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGUGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUCGGUUACCGAAUCAGACACCAAAACGACGUCCG (((((((((((((...)))))))))..))))(((((((((((((....)))...........)))))))....(((((.((..(((((((...))))))).))))))).......))).. ( -50.80) >pau.418 5792655 120 + 6537648 UUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGUGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUCGGUUACCGAAUCAGACACCAAAACGACGUCCG (((((((((((((...)))))))))..))))(((((((((((((....)))...........)))))))....(((((.((..(((((((...))))))).))))))).......))).. ( -50.80) >ppk.262 5645447 119 + 6181863 UUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGCCUUAAAACAUCAGCCCACGCAGAUGGUGCUCCCGAUUC-GUUACCGAAUCAGACACCAAAACGACAUCCG (((((((((((((...)))))))))..))))...(((.((((((....)))...........))).)))....(((((.((..(((((-(....)))))).)))))))............ ( -43.40) >pel.242 5387013 119 + 5888780 UUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGCCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGAUUC-GUUCGCGAAUCAGACACCAAAACGACAUCCG (((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((.((..(((((-(....)))))).)))))))............ ( -49.10) >pfo.2998 5719874 119 - 6438405 UUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGUCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGGUUC-CUUACCGAAUCAGACACCAAAACGACAUUCG (((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((((..((((..-...))))....)).)))))............ ( -42.60) >pf5.3374 6376814 119 - 7074893 UUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGACGCAAGUCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGGUUC-CUUACCGAAUCAGACACCAAAACGACAUCCA (((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((((..((((..-...))))....)).)))))............ ( -43.20) >consensus UUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUC_GUUACCGAAUCAGACACCAAAACGACAUCCG (((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((.((..(((((.......))))).)))))))............ (-43.47 = -42.83 + -0.64)
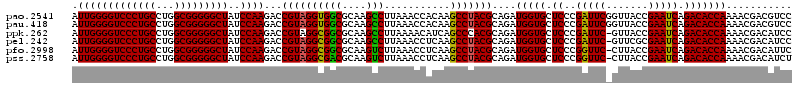
| Location | 4,781,827 – 4,781,947 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.31 |
| Mean single sequence MFE | -46.95 |
| Consensus MFE | -43.77 |
| Energy contribution | -43.13 |
| Covariance contribution | -0.64 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.68 |
| Structure conservation index | 0.93 |
| SVM decision value | 4.00 |
| SVM RNA-class probability | 0.999748 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2541 4781827 120 - 6264404 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGUGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUCGGUUACCGAAUCAGACACCAAAACGACGUCC .(((((((((((((...)))))))))..))))(((((((((((((....)))...........)))))))....(((((.((..(((((((...))))))).))))))).......))). ( -51.10) >pau.418 5792656 120 + 6537648 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGUGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUCGGUUACCGAAUCAGACACCAAAACGACGUCC .(((((((((((((...)))))))))..))))(((((((((((((....)))...........)))))))....(((((.((..(((((((...))))))).))))))).......))). ( -51.10) >ppk.262 5645448 119 + 6181863 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGCCUUAAAACAUCAGCCCACGCAGAUGGUGCUCCCGAUUC-GUUACCGAAUCAGACACCAAAACGACAUCC .(((((((((((((...)))))))))..))))...(((.((((((....)))...........))).)))....(((((.((..(((((-(....)))))).)))))))........... ( -43.70) >pel.242 5387014 119 + 5888780 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGCCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGAUUC-GUUCGCGAAUCAGACACCAAAACGACAUCC .(((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((.((..(((((-(....)))))).)))))))........... ( -49.40) >pfo.2998 5719875 119 - 6438405 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGUCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGGUUC-CUUACCGAAUCAGACACCAAAACGACAUUC .(((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((((..((((..-...))))....)).)))))........... ( -42.90) >pss.2758 5417711 119 - 6093698 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGACGCAAGUCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGGUUC-CUUACCGAAUCAGACACCAAAACGACAUCU .(((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((((..((((..-...))))....)).)))))........... ( -43.50) >consensus AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUC_GUUACCGAAUCAGACACCAAAACGACAUCC .(((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((.((..(((((.......))))).)))))))........... (-43.77 = -43.13 + -0.64)
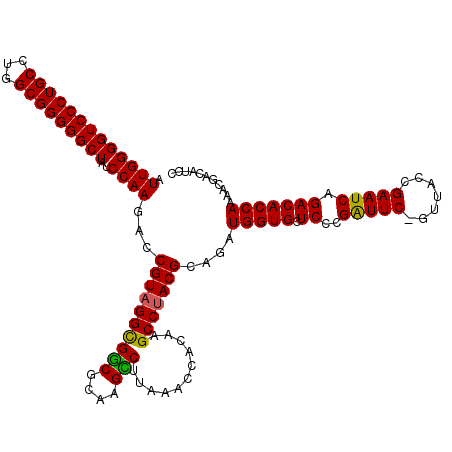
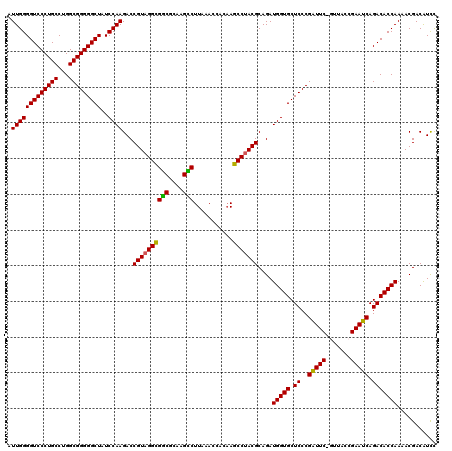
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:10:45 2007