
| Sequence ID | pao.2545 |
|---|---|
| Location | 4,792,382 – 4,792,741 |
| Length | 359 |
| Max. P | 0.999943 |

| Location | 4,792,382 – 4,792,502 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.94 |
| Mean single sequence MFE | -45.50 |
| Consensus MFE | -44.81 |
| Energy contribution | -44.53 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.98 |
| SVM decision value | 4.73 |
| SVM RNA-class probability | 0.999943 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792382 120 - 6264404 CAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGUGA ..(((((((((.....(((..((...(((....)))...))....))).....)))))))))..(((((..(((((((........))))))).....))))).((((.(....).)))) ( -44.90) >pau.414 5803213 120 + 6537648 CAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGUGA ..(((((((((.....(((..((...(((....)))...))....))).....)))))))))..(((((..(((((((........))))))).....))))).((((.(....).)))) ( -44.90) >pel.2704 5002339 120 - 5888780 CAAUGGUCGGUACAGAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCUCACAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCAA ..(((((((((...((((...((...(((....)))...))...)))).....)))))))))..(((((..(((((((........))))))).....)))))...((.(....).)).. ( -43.50) >pf5.74 6950730 120 + 7074893 CAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGA ..(((((((((.....(((..((...(((....)))...))....))).....)))))))))..(((((..(((((((........))))))).....))))).((((.(....).)))) ( -46.80) >pfo.66 6315588 120 + 6438405 CAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGA ..(((((((((.....(((..((...(((....)))...))....))).....)))))))))..(((((..(((((((........))))))).....))))).((((.(....).)))) ( -46.80) >psp.2973 5783011 120 - 5928787 CAAUGGUCGGUACAGAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCUCACAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGA ..(((((((((...((((...((...(((....)))...))...)))).....)))))))))..(((((..(((((((........))))))).....))))).((((.(....).)))) ( -46.10) >consensus CAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGA ..(((((((((.....(((((((...(((....)))...)).)))))......)))))))))..(((((..(((((((........))))))).....))))).((((.(....).)))) (-44.81 = -44.53 + -0.28)

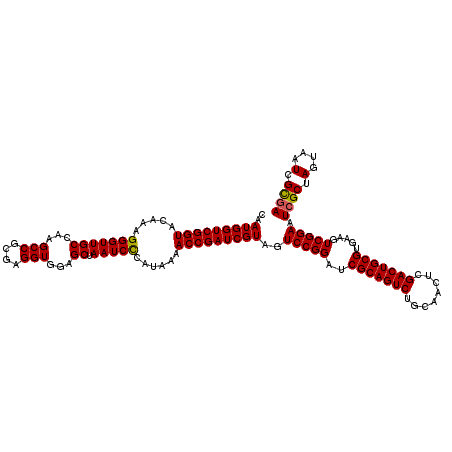
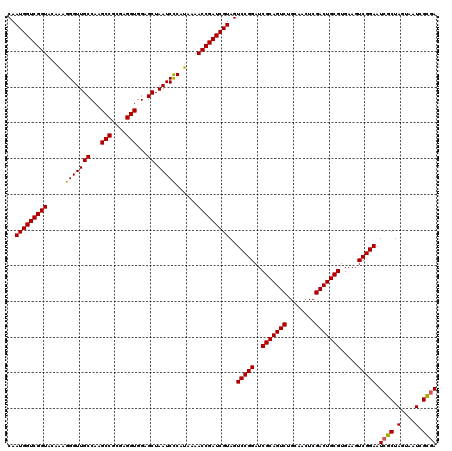
| Location | 4,792,422 – 4,792,542 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.22 |
| Mean single sequence MFE | -43.90 |
| Consensus MFE | -43.12 |
| Energy contribution | -42.90 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.51 |
| Structure conservation index | 0.98 |
| SVM decision value | 2.60 |
| SVM RNA-class probability | 0.995613 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792422 120 - 6264404 AGUCAUCAUGGCCCUUACGGCCAGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUG .((.(((.(((((.....)))))(((((((....(.....)...(((((((.....(((..((...(((....)))...))....))).....)))))))))))))).))).))...... ( -45.20) >pau.414 5803253 120 + 6537648 AGUCAUCAUGGCCCUUACGGCCAGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUG .((.(((.(((((.....)))))(((((((....(.....)...(((((((.....(((..((...(((....)))...))....))).....)))))))))))))).))).))...... ( -45.20) >ppk.338 5482746 120 + 6181863 AGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAGAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCUCACAAAACCGAUCGUAGUCCGGAUCGCAGUCUG .((.(((..((((.....))))((((((((....(.....)...(((((((...((((...((...(((....)))...))...)))).....)))))))))))))))))).))...... ( -42.90) >pf5.74 6950770 120 + 7074893 AGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUG .((.(((..((((.....))))((((((((....(.....)...(((((((.....(((..((...(((....)))...))....))).....)))))))))))))))))).))...... ( -43.60) >pfo.66 6315628 120 + 6438405 AGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUG .((.(((..((((.....))))((((((((....(.....)...(((((((.....(((..((...(((....)))...))....))).....)))))))))))))))))).))...... ( -43.60) >psp.2973 5783051 120 - 5928787 AGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAGAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCUCACAAAACCGAUCGUAGUCCGGAUCGCAGUCUG .((.(((..((((.....))))((((((((....(.....)...(((((((...((((...((...(((....)))...))...)))).....)))))))))))))))))).))...... ( -42.90) >consensus AGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUG .((.(((..((((.....)))).(((((((....(.....)...(((((((.....(((((((...(((....)))...)).)))))......)))))))))))))).))).))...... (-43.12 = -42.90 + -0.22)

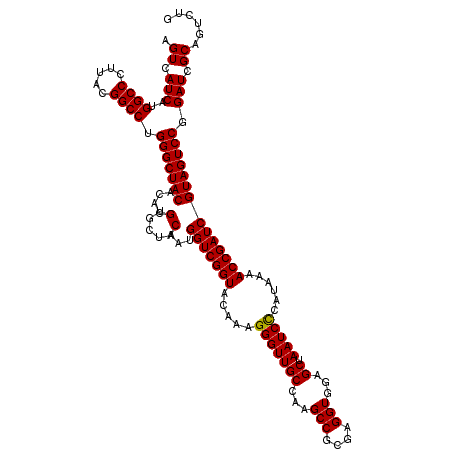
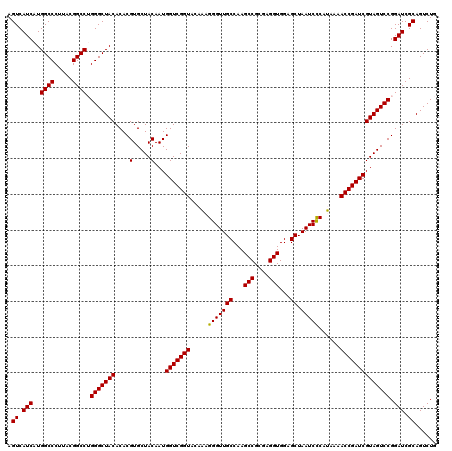
| Location | 4,792,462 – 4,792,582 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 99.11 |
| Mean single sequence MFE | -37.97 |
| Consensus MFE | -36.77 |
| Energy contribution | -37.10 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -0.82 |
| Structure conservation index | 0.97 |
| SVM decision value | 0.31 |
| SVM RNA-class probability | 0.682587 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792462 120 + 6264404 CUCCACCUCGCGGCUUGGCAACCCUUUGUACCGACCAUUGUAGCACGUGUGUAGCCCUGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGU .....(((..(((((.(((.((.(..(((..(((...)))..)))...).)).)))..)))))..)))(((..((((((.....)))))).............(((.....))))))... ( -37.00) >pau.414 5803293 120 - 6537648 CUCCACCUCGCGGCUUGGCAACCCUUUGUACCGACCAUUGUAGCACGUGUGUAGCCCUGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGU .....(((..(((((.(((.((.(..(((..(((...)))..)))...).)).)))..)))))..)))(((..((((((.....)))))).............(((.....))))))... ( -37.00) >ppk.338 5482786 120 - 6181863 CUCCACCUCGCGGCUUGGCAACCCUCUGUACCGACCAUUGUAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGU .....(((..(((((((((.((.(.((((..(((...)))..))).).).)).).))))))))..)))(((..((((((.....)))))).............(((.....))))))... ( -38.90) >pf5.74 6950810 120 - 7074893 CUCCACCUCGCGGCUUGGCAACCCUUUGUACCGACCAUUGUAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGU .....(((..(((((((((.((.(..(((..(((...)))..)))...).)).).))))))))..)))(((..((((((.....)))))).............(((.....))))))... ( -38.00) >pfo.66 6315668 120 - 6438405 CUCCACCUCGCGGCUUGGCAACCCUUUGUACCGACCAUUGUAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGU .....(((..(((((((((.((.(..(((..(((...)))..)))...).)).).))))))))..)))(((..((((((.....)))))).............(((.....))))))... ( -38.00) >psp.2973 5783091 120 + 5928787 CUCCACCUCGCGGCUUGGCAACCCUCUGUACCGACCAUUGUAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGU .....(((..(((((((((.((.(.((((..(((...)))..))).).).)).).))))))))..)))(((..((((((.....)))))).............(((.....))))))... ( -38.90) >consensus CUCCACCUCGCGGCUUGGCAACCCUUUGUACCGACCAUUGUAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGU .....(((..(((((((((.((.(..(((..(((...)))..)))...).)).).))))))))..)))(((..((((((.....)))))).............(((.....))))))... (-36.77 = -37.10 + 0.33)

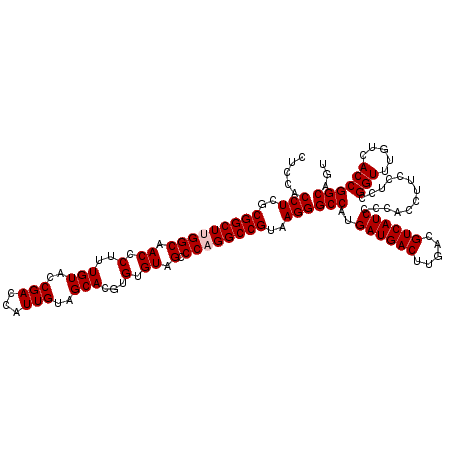
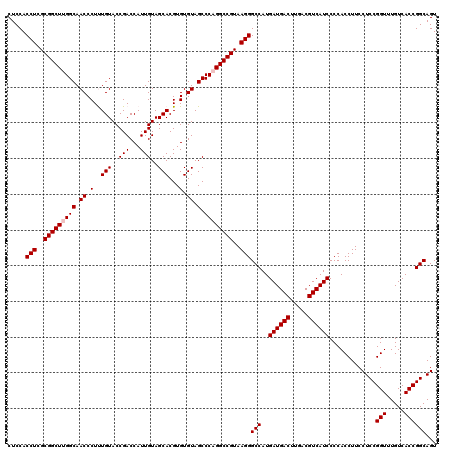
| Location | 4,792,462 – 4,792,582 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 99.11 |
| Mean single sequence MFE | -46.27 |
| Consensus MFE | -45.52 |
| Energy contribution | -45.30 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.35 |
| Structure conservation index | 0.98 |
| SVM decision value | 2.06 |
| SVM RNA-class probability | 0.987054 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792462 120 - 6264404 ACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCAGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAG .((.(((((.....))))).))..(((...((((((.....))))))..(((((((.((((((.(((.((....)))))....)))))).....))))))))))..(((....))).... ( -47.60) >pau.414 5803293 120 + 6537648 ACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCAGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAG .((.(((((.....))))).))..(((...((((((.....))))))..(((((((.((((((.(((.((....)))))....)))))).....))))))))))..(((....))).... ( -47.60) >ppk.338 5482786 120 + 6181863 ACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAGAGGGUUGCCAAGCCGCGAGGUGGAG .((.(((((.....))))).))..(((...((((((.....))))))..(((((((.(((((..(((.((....))))).....))))).....))))))))))..(((....))).... ( -45.80) >pf5.74 6950810 120 + 7074893 ACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAG .((.(((((.....))))).))..(((...((((((.....))))))..(((((((.(((((..(((.((....))))).....))))).....))))))))))..(((....))).... ( -45.40) >pfo.66 6315668 120 + 6438405 ACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAG .((.(((((.....))))).))..(((...((((((.....))))))..(((((((.(((((..(((.((....))))).....))))).....))))))))))..(((....))).... ( -45.40) >psp.2973 5783091 120 - 5928787 ACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAGAGGGUUGCCAAGCCGCGAGGUGGAG .((.(((((.....))))).))..(((...((((((.....))))))..(((((((.(((((..(((.((....))))).....))))).....))))))))))..(((....))).... ( -45.80) >consensus ACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAG .((.(((((.....))))).))..(((...((((((.....))))))..(((((((.(((((..(((.((....))))).....))))).....))))))))))..(((....))).... (-45.52 = -45.30 + -0.22)

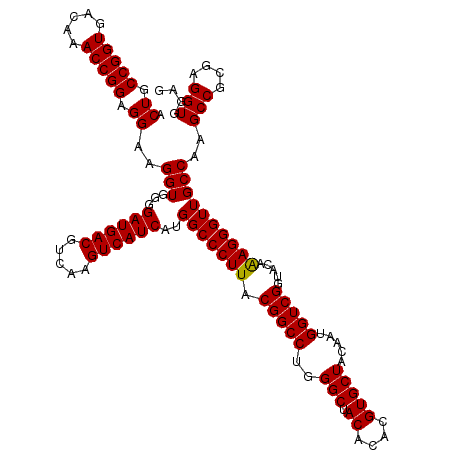
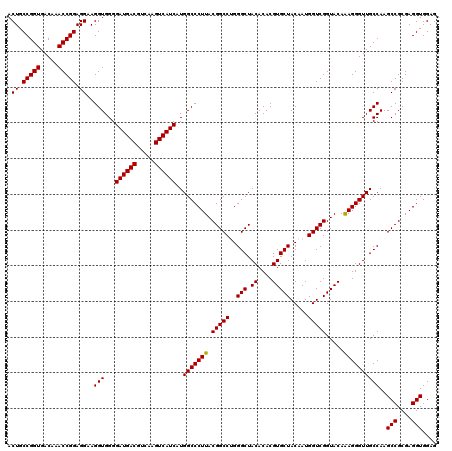
| Location | 4,792,502 – 4,792,621 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.61 |
| Mean single sequence MFE | -41.72 |
| Consensus MFE | -40.30 |
| Energy contribution | -40.30 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.40 |
| Structure conservation index | 0.97 |
| SVM decision value | 2.05 |
| SVM RNA-class probability | 0.986603 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792502 119 + 6264404 UAGCACGUGUGUAGCCCUGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACC-CGAGGUGCUGGUAACUAAGGA ..((.((.(((.((((.(((((.....))))).((((((.....)))))).............))))...)))))))....(((((((..((((((((-...))))..)))).))))))) ( -42.20) >pau.414 5803333 119 - 6537648 UAGCACGUGUGUAGCCCUGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACC-CGAGGUGCUGGUAACUAAGGA ..((.((.(((.((((.(((((.....))))).((((((.....)))))).............))))...)))))))....(((((((..((((((((-...))))..)))).))))))) ( -42.20) >ppk.338 5482826 120 - 6181863 UAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUUACGUGCUGGUAACUAAGGA (((((((((((.((((..((((.....))))..((((((.....)))))).............))))...))).((((.(((....))).)))).......))))))))........... ( -41.40) >pf5.74 6950850 120 - 7074893 UAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGA (((((((((((.((((..((((.....))))..((((((.....)))))).............))))...))).((((.(((....))).)))).......))))))))........... ( -41.50) >pfo.66 6315708 120 - 6438405 UAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGA (((((((((((.((((..((((.....))))..((((((.....)))))).............))))...))).((((.(((....))).)))).......))))))))........... ( -41.50) >psp.2973 5783131 120 + 5928787 UAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGA (((((((((((.((((..((((.....))))..((((((.....)))))).............))))...))).((((.(((....))).)))).......))))))))........... ( -41.50) >consensus UAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGA ..((.((.(((.((((..((((.....))))..((((((.....)))))).............))))...)))))))....(((((((..(((((((......)))..)))).))))))) (-40.30 = -40.30 + 0.00)

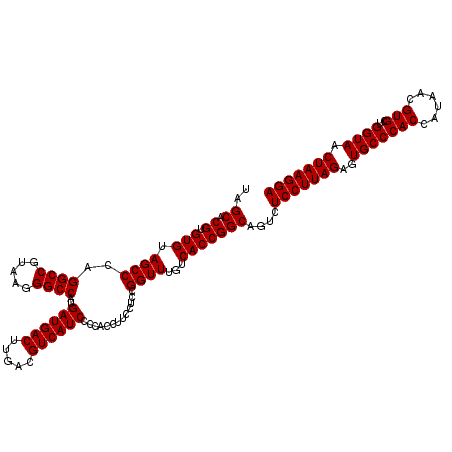
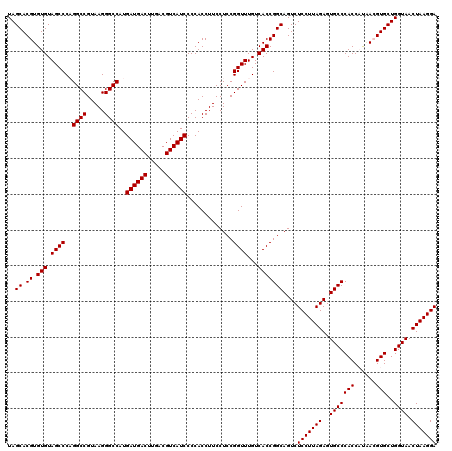
| Location | 4,792,502 – 4,792,621 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.61 |
| Mean single sequence MFE | -45.50 |
| Consensus MFE | -43.83 |
| Energy contribution | -43.83 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.50 |
| SVM RNA-class probability | 0.994634 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792502 119 - 6264404 UCCUUAGUUACCAGCACCUCG-GGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCAGGGCUACACACGUGCUA ............((((((((.-(((.((((.(((....))).))))(....)..))).))))...(((..((((((.....)))))).(((((((.......))))))))))...)))). ( -47.40) >pau.414 5803333 119 + 6537648 UCCUUAGUUACCAGCACCUCG-GGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCAGGGCUACACACGUGCUA ............((((((((.-(((.((((.(((....))).))))(....)..))).))))...(((..((((((.....)))))).(((((((.......))))))))))...)))). ( -47.40) >ppk.338 5482826 120 + 6181863 UCCUUAGUUACCAGCACGUAAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUA ............(((((((..((((.((((.(((....))).))))(....)..)))).......(((..((((((.....)))))).((((((.........)))))))))))))))). ( -45.00) >pf5.74 6950850 120 + 7074893 UCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUA ............(((((((..((((.((((.(((....))).))))(....)..)))).......(((..((((((.....)))))).((((((.........)))))))))))))))). ( -44.40) >pfo.66 6315708 120 + 6438405 UCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUA ............(((((((..((((.((((.(((....))).))))(....)..)))).......(((..((((((.....)))))).((((((.........)))))))))))))))). ( -44.40) >psp.2973 5783131 120 - 5928787 UCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUA ............(((((((..((((.((((.(((....))).))))(....)..)))).......(((..((((((.....)))))).((((((.........)))))))))))))))). ( -44.40) >consensus UCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUA (((((.((((((..(((......)))((((.(((....))).))))))))))......)))))..(((..((((((.....)))))).((((((.........)))))))))........ (-43.83 = -43.83 + 0.00)

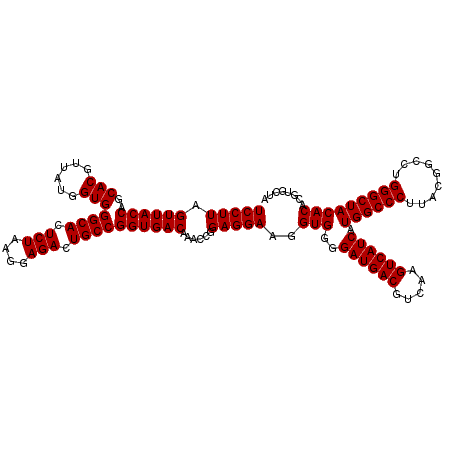
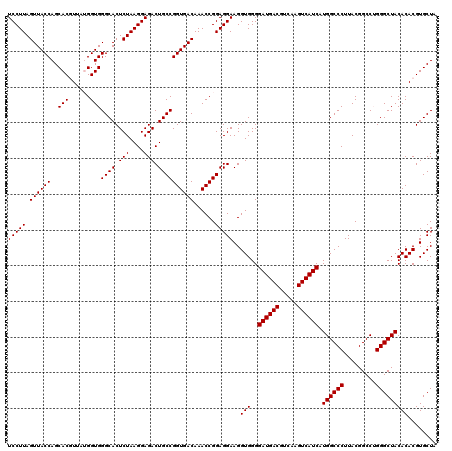
| Location | 4,792,542 – 4,792,661 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.05 |
| Mean single sequence MFE | -34.50 |
| Consensus MFE | -34.47 |
| Energy contribution | -34.47 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -0.97 |
| Structure conservation index | 1.00 |
| SVM decision value | 1.27 |
| SVM RNA-class probability | 0.937975 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792542 119 + 6264404 UGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACC-CGAGGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCA .((((.((.(((.......(((((((.....))))).))..(((((((..((((((((-...))))..)))).)))))))...))).)))).)).....(((......)))......... ( -35.90) >pau.414 5803373 119 - 6537648 UGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACC-CGAGGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCA .((((.((.(((.......(((((((.....))))).))..(((((((..((((((((-...))))..)))).)))))))...))).)))).)).....(((......)))......... ( -35.90) >ppk.338 5482866 120 - 6181863 UGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUUACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCA .((((.((.(((.......(((((((.....))))).))..(((((((..(((((((......)))..)))).)))))))...))).)))).)).....(((......)))......... ( -33.80) >pf5.74 6950890 120 - 7074893 UGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCA .((((.((.(((.......(((((((.....))))).))..(((((((..(((((((......)))..)))).)))))))...))).)))).)).....(((......)))......... ( -33.80) >pfo.66 6315748 120 - 6438405 UGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCA .((((.((.(((.......(((((((.....))))).))..(((((((..(((((((......)))..)))).)))))))...))).)))).)).....(((......)))......... ( -33.80) >psp.2973 5783171 120 + 5928787 UGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCA .((((.((.(((.......(((((((.....))))).))..(((((((..(((((((......)))..)))).)))))))...))).)))).)).....(((......)))......... ( -33.80) >consensus UGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCA .((((.((.(((.......(((((((.....))))).))..(((((((..(((((((......)))..)))).)))))))...))).)))).)).....(((......)))......... (-34.47 = -34.47 + -0.00)

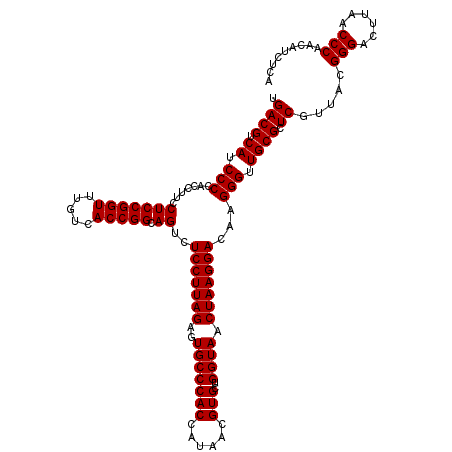
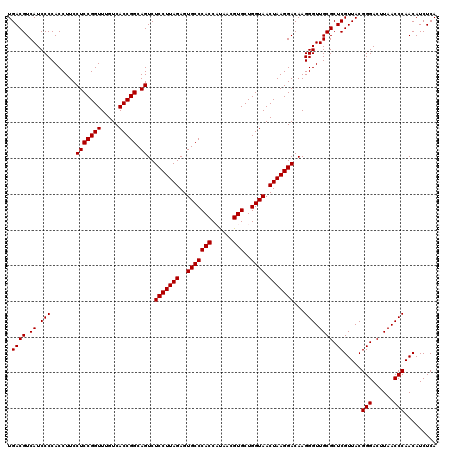
| Location | 4,792,542 – 4,792,661 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.05 |
| Mean single sequence MFE | -42.10 |
| Consensus MFE | -42.03 |
| Energy contribution | -42.03 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.06 |
| Structure conservation index | 1.00 |
| SVM decision value | 1.55 |
| SVM RNA-class probability | 0.963466 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792542 119 - 6264404 UGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACCUCG-GGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCA ...(((((((((......))).............((((...((((.((.....)).((((.-(((.((((.(((....))).))))(....)..))).)))))))).))))..)))))). ( -43.10) >pau.414 5803373 119 + 6537648 UGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACCUCG-GGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCA ...(((((((((......))).............((((...((((.((.....)).((((.-(((.((((.(((....))).))))(....)..))).)))))))).))))..)))))). ( -43.10) >ppk.338 5482866 120 + 6181863 UGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUAAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCA ...(((((((((((..(..(......)..)..)))))..((((((.((((((..(((......)))((((.(((....))).))))))))))..(((.......))))))))))))))). ( -41.60) >pf5.74 6950890 120 + 7074893 UGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCA ...(((((((((((..(..(......)..)..)))))..((((((.((((((..(((......)))((((.(((....))).))))))))))..(((.......))))))))))))))). ( -41.60) >pfo.66 6315748 120 + 6438405 UGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCA ...(((((((((((..(..(......)..)..)))))..((((((.((((((..(((......)))((((.(((....))).))))))))))..(((.......))))))))))))))). ( -41.60) >psp.2973 5783171 120 - 5928787 UGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCA ...(((((((((((..(..(......)..)..)))))..((((((.((((((..(((......)))((((.(((....))).))))))))))..(((.......))))))))))))))). ( -41.60) >consensus UGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCA ...(((((((((((..(..(......)..)..)))))..((((((.((((((..(((......)))((((.(((....))).))))))))))..(((.......))))))))))))))). (-42.03 = -42.03 + 0.00)

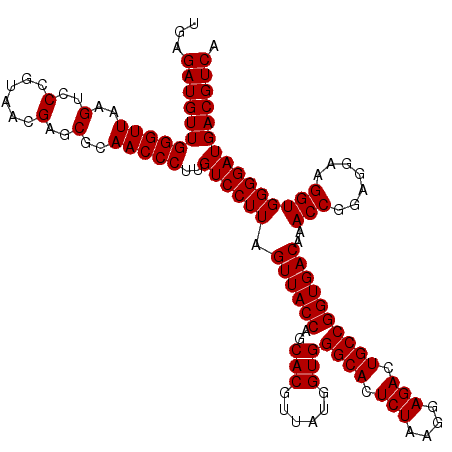
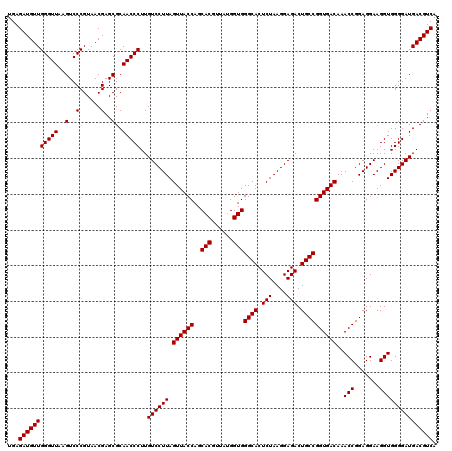
| Location | 4,792,582 – 4,792,701 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.28 |
| Mean single sequence MFE | -36.87 |
| Consensus MFE | -35.97 |
| Energy contribution | -35.97 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.06 |
| Structure conservation index | 0.98 |
| SVM decision value | 1.24 |
| SVM RNA-class probability | 0.934381 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792582 119 + 6264404 CUCCUUAGAGUGCCCACC-CGAGGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCUGUGUCUGA .(((((((..((((((((-...))))..)))).)))))))...((((((.(((((...)))).).))))))......(((.((((((.((((.((.......))))))...))))))))) ( -38.90) >pau.414 5803413 119 - 6537648 CUCCUUAGAGUGCCCACC-CGAGGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCUGUGUCUGA .(((((((..((((((((-...))))..)))).)))))))...((((((.(((((...)))).).))))))......(((.((((((.((((.((.......))))))...))))))))) ( -38.90) >pss.3053 5952034 120 + 6093698 CUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCUGUCUCAAU .(((((((..(((((((......)))..)))).)))))))..((((((((((((((...(((......)))....((....)).))))))((.(....).)).))))).)))........ ( -35.30) >pel.2704 5002539 120 + 5888780 CUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCUGUGUCAGA .(((((((..(((((((......)))..)))).)))))))...((((((.(((((...)))).).))))))..........((((((.((((.((.......))))))...))))))... ( -36.40) >ppk.338 5482906 120 - 6181863 CUCCUUAGAGUGCCCACCAUUACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCUGUGUCAGA .(((((((..(((((((......)))..)))).)))))))...((((((.(((((...)))).).))))))..........((((((.((((.((.......))))))...))))))... ( -36.40) >psp.2973 5783211 120 + 5928787 CUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCUGUCUCAAU .(((((((..(((((((......)))..)))).)))))))..((((((((((((((...(((......)))....((....)).))))))((.(....).)).))))).)))........ ( -35.30) >consensus CUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCUGUGUCAGA .(((((((..(((((((......)))..)))).)))))))..((((((((((((((...(((......)))....((....)).))))))((.(....).)).))))).)))........ (-35.97 = -35.97 + -0.00)
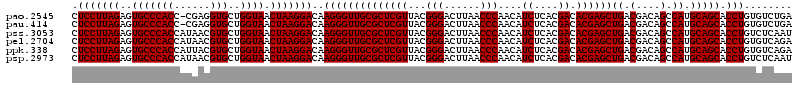
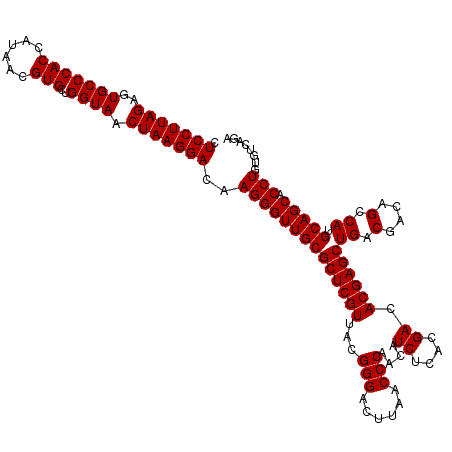
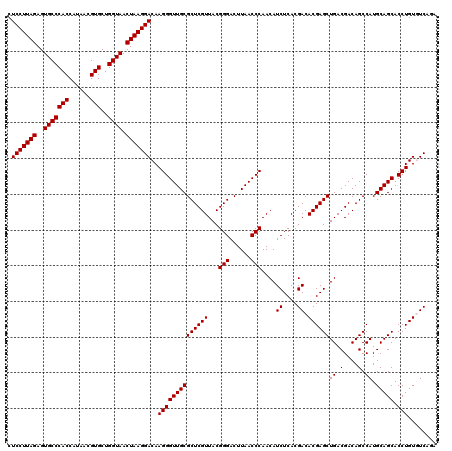
| Location | 4,792,582 – 4,792,701 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.28 |
| Mean single sequence MFE | -42.10 |
| Consensus MFE | -41.30 |
| Energy contribution | -41.30 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -0.74 |
| Structure conservation index | 0.98 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.677761 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792582 119 - 6264404 UCAGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACCUCG-GGUGGGCACUCUAAGGAG .......((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).))))(((((((...(((.(......-).))).....))))))). ( -41.70) >pau.414 5803413 119 + 6537648 UCAGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACCUCG-GGUGGGCACUCUAAGGAG .......((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).))))(((((((...(((.(......-).))).....))))))). ( -41.70) >pss.3053 5952034 120 - 6093698 AUUGAGACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAG .......((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).))))(((((((...(((.((.....)).))).....))))))). ( -42.30) >pel.2704 5002539 120 - 5888780 UCUGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAG .......((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).))))(((((((...(((.((.....)).))).....))))))). ( -42.30) >ppk.338 5482906 120 + 6181863 UCUGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUAAUGGUGGGCACUCUAAGGAG .......((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).))))(((((((...(((.((.....)).))).....))))))). ( -42.30) >psp.2973 5783211 120 - 5928787 AUUGAGACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAG .......((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).))))(((((((...(((.((.....)).))).....))))))). ( -42.30) >consensus UCUGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAG .......((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).))))(((((((...(((.(.......).))).....))))))). (-41.30 = -41.30 + 0.00)

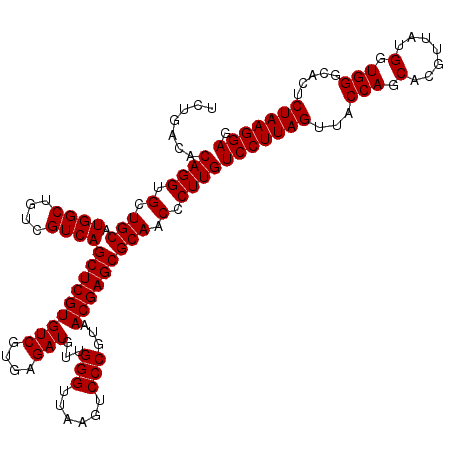
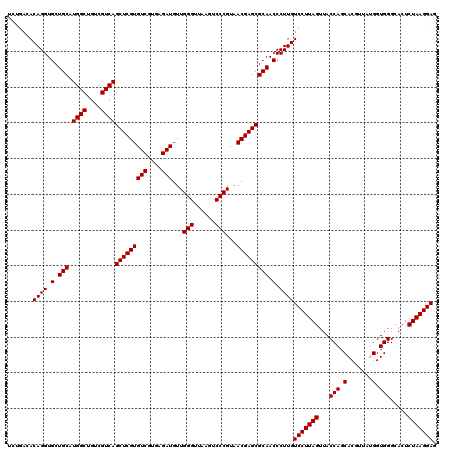
| Location | 4,792,621 – 4,792,741 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.11 |
| Mean single sequence MFE | -42.17 |
| Consensus MFE | -43.99 |
| Energy contribution | -41.77 |
| Covariance contribution | -2.22 |
| Combinations/Pair | 1.13 |
| Mean z-score | -0.77 |
| Structure conservation index | 1.04 |
| SVM decision value | 1.16 |
| SVM RNA-class probability | 0.923739 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2545 4792621 120 - 6264404 GCUGAGAACUUUCCAGAGAUGGAUUGGUGCCUUCGGGAACUCAGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUG .(((((.(((.((((....))))..))).((....))..)))))....(((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).)))).. ( -43.80) >pau.414 5803452 120 + 6537648 GCUGAGAACUUUCCAGAGAUGGAUUGGUGCCUUCGGGAACUCAGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUG .(((((.(((.((((....))))..))).((....))..)))))....(((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).)))).. ( -43.80) >pss.3053 5952074 120 - 6093698 CCAAUGAACUUUCCAGAGAUGGAUUGGUGCCUUCGGGAGCAUUGAGACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUG .(((((.(((.((((....))))..))).((....))..)))))....(((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).)))).. ( -41.10) >ppk.338 5482946 120 + 6181863 GCAGAGAACUUUCCAGAGAUGGAUUGGUGCCUUCGGGAACUCUGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUG .(((((.(((.((((....))))..))).((....))..)))))....(((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).)))).. ( -43.80) >pf5.74 6950970 120 + 7074893 CCAAUGAACUUUCUAGAGAUAGAUUGGUGCCUUCGGGAACAUUGAGACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUG .(((((.(((.((((....))))..))).((....))..)))))....(((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).)))).. ( -39.40) >pfo.66 6315828 120 + 6438405 CCAAUGAACUUUCCAGAGAUGGAUUGGUGCCUUCGGGAGCAUUGAGACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUG .(((((.(((.((((....))))..))).((....))..)))))....(((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).)))).. ( -41.10) >consensus CCAAAGAACUUUCCAGAGAUGGAUUGGUGCCUUCGGGAACACUGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUG ((((((.(((.((((....))))..))).((....))..))))).)..(((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).)))).. (-43.99 = -41.77 + -2.22)

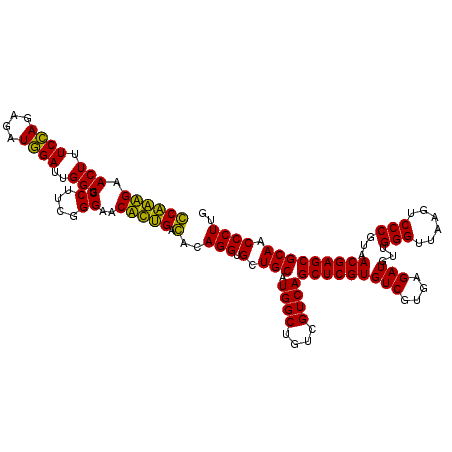
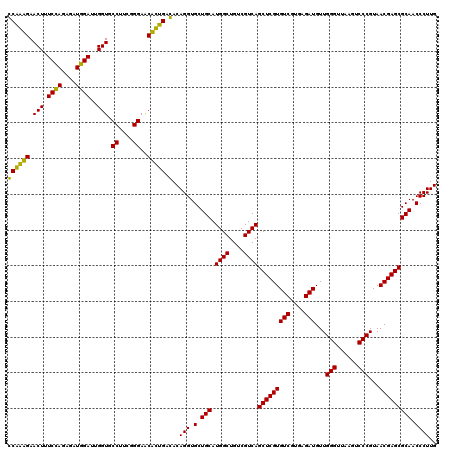
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:09:53 2007