
| Sequence ID | pao.3269 |
|---|---|
| Location | 6,172,084 – 6,172,202 |
| Length | 118 |
| Max. P | 0.988025 |

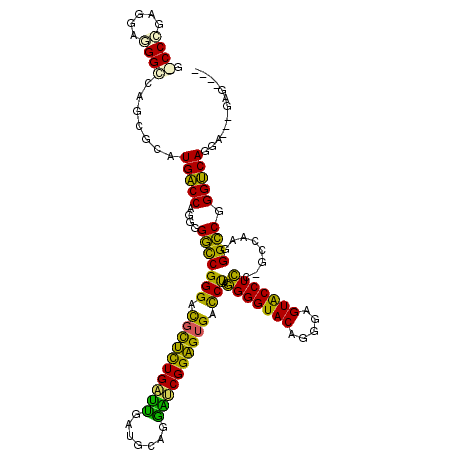
| Location | 6,172,084 – 6,172,193 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 72.23 |
| Mean single sequence MFE | -49.03 |
| Consensus MFE | -43.60 |
| Energy contribution | -40.60 |
| Covariance contribution | -3.00 |
| Combinations/Pair | 1.55 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.91 |
| SVM RNA-class probability | 0.982410 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
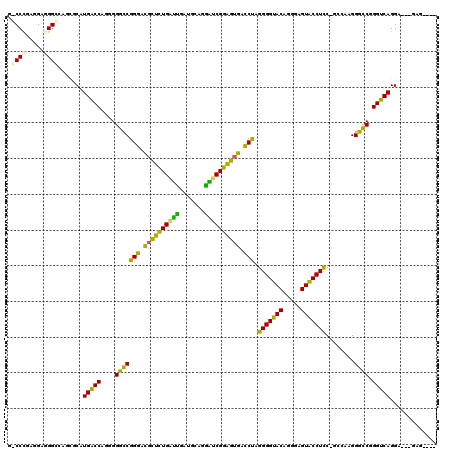
>pao.3269 6172084 109 - 6264404 G-CCCGCAGCGGG-----GCAUGGCCAGGGGACCGGGAUGCGCUGGGUGAGGUCGAUCCGGCGGGUUCCAGGGGUACAGGGAGUACCUCC-UUCG-GGGUCGGGCCAGGC---GAAAACG .-((((...))))-----((.(((((....(((((((((.((((((((.......)))))))).))))).(((((((.....)))))))(-....-))))).))))).))---....... ( -54.40) >pf5.58 6979890 112 + 7074893 A-CCCGAGGAGGGCGAGUGCAUGACCAAGGGGCCGGGAUGUUCUGAUCCACGGGUGAUCGGAGUGACCUAGGGGUACAGGGAGUGCCUCC-G-CAAGGGCCGGGUCAGGA-AAGAG---- .-(((.....)))........(((((....(((((((.(..((((((((....).)))))))..).))).(((((((.....)))))))(-.-...))))).)))))...-.....---- ( -42.00) >pss.169 276442 109 - 6093698 G-CCUGAGGAAGGUACGCGCAUGACCAGGGGGCCGGGACGCGUUGAUCAA--GUGGAUCGGCGUGACCUAGGGGUACAGGGAGUACCUCC-GCCAAGGGCCGGGUCAGGC---AAG---- (-(((.....))))....((.(((((....(((((((.((((((((((..--...)))))))))).))).(((((((.....))))).))-......)))).))))).))---...---- ( -50.20) >pel.55 5800994 112 + 5888780 GCCCCGAGGACGGGCUGCGCAUGACCAGGGGGCCGGGCCGUUCUGACUGUUGCAGGGUCGGAGUGGCCUAGGGGUACAAGGAGUACCUCC-GCGAUGGGCCGGGUCAGGA---GAG---- ((((((....))))..))...(((((....((((((((((((((((((.......)))))))))))))).(((((((.....))))).))-......)))).)))))...---...---- ( -52.20) >ppk.81 6037643 112 + 6181863 GCCCCGAGGAGGGGUAGCACAUGACCAAGGGGUCGGGCUGCUCUGAUUGUUACAGGACCGGAGUGGCCUAGGGGUACAGGGAGUGCCUCCAGUAAUGGGCCGGGUCAGGA----AA---- (((((.....)))))......(((((....((((((((..(((((...(((....))))))))..)))).(((((((.....)))))))((....)))))).)))))...----..---- ( -48.80) >pfo.23 47877 115 - 6438405 GGCCCGAGGAAGGCCGGUGCAUGACCAGGGGGCCGGGACGUUCUGACUGUUGCAGGGUCGGAGUGACCUACGGGUACAGGGAGUGCCUGC-GAUGAGGGCCGGGUCAGGGUAAAAG---- ((((.......))))..(((.(((((....(((((((.((((((((((.......)))))))))).))).(((((((.....))))))).-......)))).)))))..)))....---- ( -46.60) >consensus G_CCCGAGGAGGGCCAGCGCAUGACCAGGGGGCCGGGACGCUCUGAUUGAUGCAGGAUCGGAGUGACCUAGGGGUACAGGGAGUACCUCC_GCCAAGGGCCGGGUCAGGA___GAG____ .((((.....)))).......(((((....(((((((.((((((((((.......)))))))))).))).(((((((.....)))))))........)))).)))))............. (-43.60 = -40.60 + -3.00)

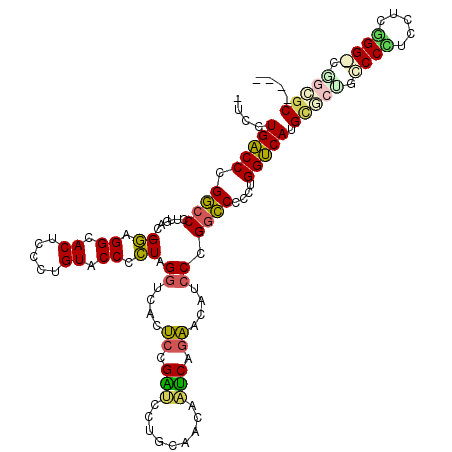
| Location | 6,172,091 – 6,172,202 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 72.88 |
| Mean single sequence MFE | -42.92 |
| Consensus MFE | -25.21 |
| Energy contribution | -24.73 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.45 |
| Mean z-score | -1.31 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.543364 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3269 6172091 111 + 6264404 -GCCUGGCCCGACCC-CGAA-GGAGGUACUCCCUGUACCCCUGGAACCCGCCGGAUCGACCUCACCCAGCGCAUCCCGGUCCCCUGGCCAUGC-----CCCGCUGCGGG-CGGGGCGGUG -((((((((.((((.-...(-((.(((((.....))))))))(((...(((.((...........)).)))..))).))))....))))).((-----(((((.....)-))))))))). ( -51.20) >pf5.58 6979895 112 - 7074893 -UCCUGACCCGGCCCUUG-C-GGAGGCACUCCCUGUACCCCUAGGUCACUCCGAUCACCCGUGGAUCAGAACAUCCCGGCCCCUUGGUCAUGCACUCGCCCUCCUCGGG-UGGUGC---- -...(((((.((((....-(-((((......((((......))))...))))).......(.((((......)))))))))....))))).((((.(((((.....)))-))))))---- ( -39.90) >pss.169 276445 111 + 6093698 -GCCUGACCCGGCCCUUGGC-GGAGGUACUCCCUGUACCCCUAGGUCACGCCGAUCCAC--UUGAUCAACGCGUCCCGGCCCCCUGGUCAUGCGCGUACCUUCCUCAGG-CCGCAC---- -((((((....(((...)))-((((((((.....)))))...((((.((((.((((...--..))))...((((.((((....))))..))))))))))))))))))))-).....---- ( -41.70) >pel.55 5800997 114 - 5888780 -UCCUGACCCGGCCCAUCGC-GGAGGUACUCCUUGUACCCCUAGGCCACUCCGACCCUGCAACAGUCAGAACGGCCCGGCCCCCUGGUCAUGCGCAGCCCGUCCUCGGGGCCGCGC---- -...(((((.((((......-((.(((((.....)))))))..((((..((.(((.........))).))..)))).))))....))))).((((.((((.......)))).))))---- ( -47.60) >ppk.81 6037645 115 - 6181863 -UCCUGACCCGGCCCAUUACUGGAGGCACUCCCUGUACCCCUAGGCCACUCCGGUCCUGUAACAAUCAGAGCAGCCCGACCCCUUGGUCAUGUGCUACCCCUCCUCGGGGCGGCGC---- -...(((((.((((...(((.((((...))))..)))......)))).....((((((((..........))))...))))....))))).((((..((((.....))))..))))---- ( -39.20) >pfo.23 47882 115 + 6438405 ACCCUGACCCGGCCCUCAUC-GCAGGCACUCCCUGUACCCGUAGGUCACUCCGACCCUGCAACAGUCAGAACGUCCCGGCCCCCUGGUCAUGCACCGGCCUUCCUCGGGCCGACGC---- ...(((((............-(((((.....)))))....(((((((.....)).)))))....)))))..((((..(((((...((((.......))))......))))))))).---- ( -37.90) >consensus _UCCUGACCCGGCCCUUGAC_GGAGGCACUCCCUGUACCCCUAGGUCACUCCGAUCCUGCAACAAUCAGAACAUCCCGGCCCCCUGGUCAUGCGCUGCCCCUCCUCGGG_CGGCGC____ ....(((((.((((.......((.((.((.....)).)).)).((....((.(((.........))).))....)).))))....))))).(((((.((((.....)))).))))).... (-25.21 = -24.73 + -0.48)


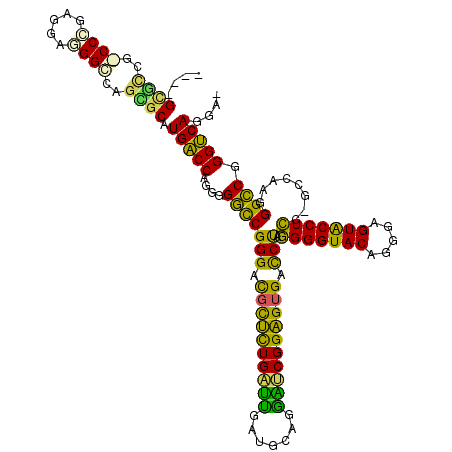
| Location | 6,172,091 – 6,172,202 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 72.88 |
| Mean single sequence MFE | -56.82 |
| Consensus MFE | -48.93 |
| Energy contribution | -45.93 |
| Covariance contribution | -3.00 |
| Combinations/Pair | 1.59 |
| Mean z-score | -2.26 |
| Structure conservation index | 0.86 |
| SVM decision value | 2.10 |
| SVM RNA-class probability | 0.988025 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3269 6172091 111 - 6264404 CACCGCCCCG-CCCGCAGCGGG-----GCAUGGCCAGGGGACCGGGAUGCGCUGGGUGAGGUCGAUCCGGCGGGUUCCAGGGGUACAGGGAGUACCUCC-UUCG-GGGUCGGGCCAGGC- ..((((((((-(.....)))))-----)).(((((....(((((((((.((((((((.......)))))))).))))).(((((((.....)))))))(-....-))))).))))))).- ( -64.20) >pf5.58 6979895 112 + 7074893 ----GCACCA-CCCGAGGAGGGCGAGUGCAUGACCAAGGGGCCGGGAUGUUCUGAUCCACGGGUGAUCGGAGUGACCUAGGGGUACAGGGAGUGCCUCC-G-CAAGGGCCGGGUCAGGA- ----(((((.-(((.....))).).)))).(((((....(((((((.(..((((((((....).)))))))..).))).(((((((.....)))))))(-.-...))))).)))))...- ( -50.40) >pss.169 276445 111 - 6093698 ----GUGCGG-CCUGAGGAAGGUACGCGCAUGACCAGGGGGCCGGGACGCGUUGAUCAA--GUGGAUCGGCGUGACCUAGGGGUACAGGGAGUACCUCC-GCCAAGGGCCGGGUCAGGC- ----((((((-(((.....)))).))))).(((((....(((((((.((((((((((..--...)))))))))).))).(((((((.....))))).))-......)))).)))))...- ( -55.40) >pel.55 5800997 114 + 5888780 ----GCGCGGCCCCGAGGACGGGCUGCGCAUGACCAGGGGGCCGGGCCGUUCUGACUGUUGCAGGGUCGGAGUGGCCUAGGGGUACAAGGAGUACCUCC-GCGAUGGGCCGGGUCAGGA- ----(((((((((.......))))))))).(((((....((((((((((((((((((.......)))))))))))))).(((((((.....))))).))-......)))).)))))...- ( -64.80) >ppk.81 6037645 115 + 6181863 ----GCGCCGCCCCGAGGAGGGGUAGCACAUGACCAAGGGGUCGGGCUGCUCUGAUUGUUACAGGACCGGAGUGGCCUAGGGGUACAGGGAGUGCCUCCAGUAAUGGGCCGGGUCAGGA- ----(.((..((((.....))))..)).).(((((....((((((((..(((((...(((....))))))))..)))).(((((((.....)))))))((....)))))).)))))...- ( -53.50) >pfo.23 47882 115 - 6438405 ----GCGUCGGCCCGAGGAAGGCCGGUGCAUGACCAGGGGGCCGGGACGUUCUGACUGUUGCAGGGUCGGAGUGACCUACGGGUACAGGGAGUGCCUGC-GAUGAGGGCCGGGUCAGGGU ----((..(((((.......)))))..)).(((((....(((((((.((((((((((.......)))))))))).))).(((((((.....))))))).-......)))).))))).... ( -52.60) >consensus ____GCGCCG_CCCGAGGAGGGCCAGCGCAUGACCAGGGGGCCGGGACGCUCUGAUUGAUGCAGGAUCGGAGUGACCUAGGGGUACAGGGAGUACCUCC_GCCAAGGGCCGGGUCAGGA_ ....((((..((((.....))))..)))).(((((....(((((((.((((((((((.......)))))))))).))).(((((((.....)))))))........)))).))))).... (-48.93 = -45.93 + -3.00)

Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:06:37 2007