
| Sequence ID | pao.2746 |
|---|---|
| Location | 5,196,939 – 5,197,185 |
| Length | 246 |
| Max. P | 0.994882 |

| Location | 5,196,939 – 5,197,059 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.11 |
| Mean single sequence MFE | -50.45 |
| Consensus MFE | -33.21 |
| Energy contribution | -32.46 |
| Covariance contribution | -0.75 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.593445 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2746 5196939 120 + 6264404 GCUUGCCCAAGCCGCCCGGCUUGGGCAGAAUGCGCCCCGUCCUAGGGGAGUAGUCUCCCGCGAGCGCCGCACCGGCCUCGCCCGGCACGCGUCAACACACUUGGUCCGCAGACCAUGGCG (((((((((((((....))))))))))((.((((..(((......(((((....)))))(((((.((((...))))))))).)))..))))))........(((((....))))).))). ( -61.60) >pau.2882 5468918 120 + 6537648 GCUUGCCCAAGCCGCCCGGCUUGGGCAGAAUGCGCCCCGUCCUAGGGGAGUAGUCUCCCGCGAGCGCCGCACCGGCCUCGCCCGGCACGCGUCAACACACUUGGUCCGCAGACCAUGGCG (((((((((((((....))))))))))((.((((..(((......(((((....)))))(((((.((((...))))))))).)))..))))))........(((((....))))).))). ( -61.60) >ppk.434 5305714 115 - 6181863 GCUUGCCGAAAACGUUUUGCCUAGGCAGAAUCCGCACCGUCCUAGGGGAGUAGUCUCCCGCGAGCACCCAG-----CUCGCCCGGUACGCGUCAACAUACUUGGUCCACAGACCAUGGCG ....(((((....(((((((....))))))).(((((((......(((((....)))))((((((.....)-----))))).))))..)))))........(((((....))))).))). ( -45.40) >pel.475 4951136 114 - 5888780 GCUUGCCGAAACCGCCCAGCGUAGGCAGAAUCCGCUCCGUCCUAGGGGAGUAGUCUCCCGCGAGCCCCC-G-----CUCGCCCGGUACGCGUCAACACACUUGGCCCACAGGCCAUGGCG ....((((..........((((((((.......)))(((......(((((....)))))((((((....-)-----))))).))))))))...........(((((....))))))))). ( -43.20) >pfo.2801 5321639 115 + 6438405 ACUUGCCGAAUGGAUUACGCCUAGGCAGAAUCCGCUUCGUCCUAGGGGAGUAGUCUCCCACGAGCGCCAAG-----CUCGUCCGGCAUACGUCAACAUACUUGAUCCUCAGAUCAUGGCG ...(((((((((((((..((....))..)))))).))).......(((((....)))))((((((.....)-----)))))..))))..(((((.......(((((....)))))))))) ( -42.01) >pf5.3120 5888230 114 + 7074893 -CUUGCCGAAAGGGUUUUGCCUAGGCAGAAUCCGCUCCGUCCUAGGGGAGUAGUCUCCCACGAGCGCCACG-----CUCGUCCGGCAUGCGUCAACAUACUUGGUCAACAGACCAUGGCG -...(((((..(((((((((....)))))))))((.(((......(((((....)))))(((((((...))-----))))).)))...)).))........(((((....))))).))). ( -48.90) >consensus GCUUGCCGAAACCGCCCGGCCUAGGCAGAAUCCGCUCCGUCCUAGGGGAGUAGUCUCCCGCGAGCGCCACG_____CUCGCCCGGCACGCGUCAACACACUUGGUCCACAGACCAUGGCG ((.(((((............((((((.(........).).)))))(((((....)))))(((((............))))).))))).))((((.......(((((....))))))))). (-33.21 = -32.46 + -0.75)

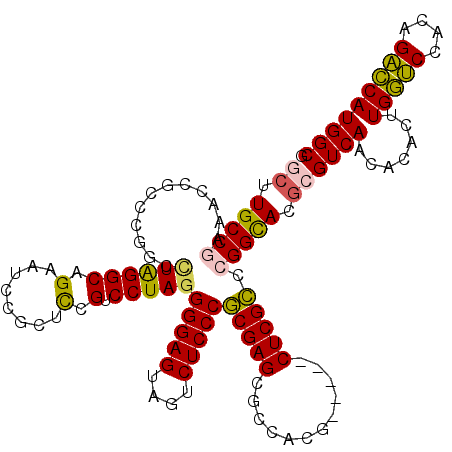
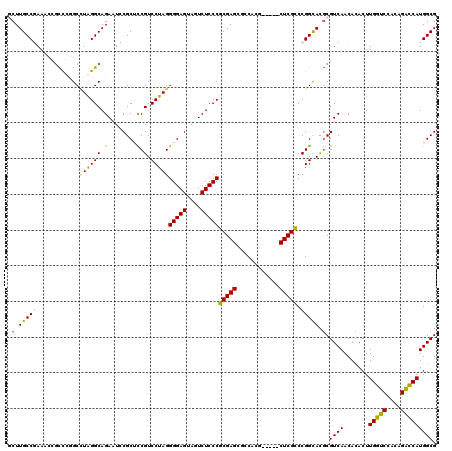
| Location | 5,196,979 – 5,197,077 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.31 |
| Mean single sequence MFE | -42.98 |
| Consensus MFE | -35.62 |
| Energy contribution | -34.01 |
| Covariance contribution | -1.61 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.26 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.966790 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2746 5196979 98 + 6264404 CCUAGGGGAGUAGUCUCCCGCGAGCGCCGCACCGGCCUCGCCCGGCACGCGUCAACACACUUGGUCCGCAGACCAUGGCGCGCGCGACCC-A---------------------AGCCUGC .....(((((....)))))(((((.((((...)))))))))...((.(((((((.......(((((....)))))))))))).)).....-.---------------------....... ( -42.61) >pau.2882 5468958 98 + 6537648 CCUAGGGGAGUAGUCUCCCGCGAGCGCCGCACCGGCCUCGCCCGGCACGCGUCAACACACUUGGUCCGCAGACCAUGGCGCGCGCGACCC-A---------------------AGCCUGC .....(((((....)))))(((((.((((...)))))))))...((.(((((((.......(((((....)))))))))))).)).....-.---------------------....... ( -42.61) >ppk.434 5305754 114 - 6181863 CCUAGGGGAGUAGUCUCCCGCGAGCACCCAG-----CUCGCCCGGUACGCGUCAACAUACUUGGUCCACAGACCAUGGCGCGUGCGACCC-ACCAUGCACGCCUGCAUGGAGAGAUCCGC .....(((((....)))))((((((.....)-----))))).((((((((((((.......(((((....))))))))))))))).....-.(((((((....))))))).......)). ( -51.31) >pel.475 4951176 93 - 5888780 CCUAGGGGAGUAGUCUCCCGCGAGCCCCC-G-----CUCGCCCGGUACGCGUCAACACACUUGGCCCACAGGCCAUGGCGCGUGCGACCC-AC--------------------GAUCUGC .....(((((....)))))((((((....-)-----)))))...((((((((((.......(((((....))))))))))))))).....-..--------------------....... ( -42.41) >pfo.2801 5321679 95 + 6438405 CCUAGGGGAGUAGUCUCCCACGAGCGCCAAG-----CUCGUCCGGCAUACGUCAACAUACUUGAUCCUCAGAUCAUGGCGUAUGCGACCCAAG--------------------CGUCCGC .....(((((....)))))((((((.....)-----))))).((((((((((((.......(((((....)))))))))))))))(((.....--------------------.))))). ( -37.11) >pf5.3120 5888269 95 + 7074893 CCUAGGGGAGUAGUCUCCCACGAGCGCCACG-----CUCGUCCGGCAUGCGUCAACAUACUUGGUCAACAGACCAUGGCGCAUGCGACCCAAG--------------------AGUCCGC .....(((((....)))))(((((((...))-----))))).((((((((((((.......(((((....)))))))))))))))(((.....--------------------.))))). ( -41.81) >consensus CCUAGGGGAGUAGUCUCCCGCGAGCGCCACG_____CUCGCCCGGCACGCGUCAACACACUUGGUCCACAGACCAUGGCGCGUGCGACCC_A_____________________AGUCCGC .....(((((....)))))(((((............)))))...((((((((((.......(((((....)))))))))))))))................................... (-35.62 = -34.01 + -1.61)

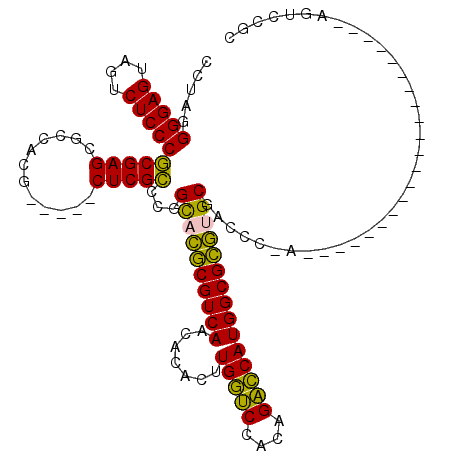
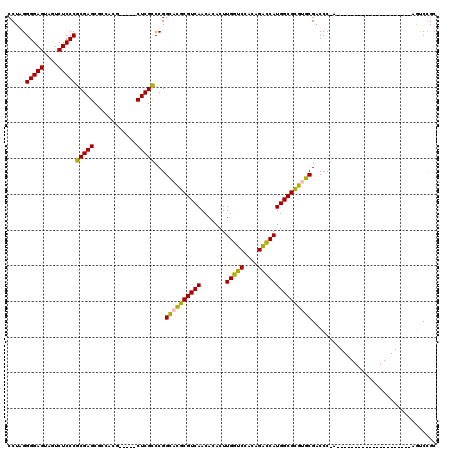
| Location | 5,196,979 – 5,197,077 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.31 |
| Mean single sequence MFE | -45.35 |
| Consensus MFE | -33.24 |
| Energy contribution | -31.58 |
| Covariance contribution | -1.66 |
| Combinations/Pair | 1.26 |
| Mean z-score | -0.91 |
| Structure conservation index | 0.73 |
| SVM decision value | -0.06 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>pao.2746 5196979 98 - 6264404 GCAGGCU---------------------U-GGGUCGCGCGCGCCAUGGUCUGCGGACCAAGUGUGUUGACGCGUGCCGGGCGAGGCCGGUGCGGCGCUCGCGGGAGACUACUCCCCUAGG .....((---------------------(-(((.((((.(((((.(((((....))))).((((....))))(..((((......))))..)))))).))))((((....)))))))))) ( -50.60) >pau.2882 5468958 98 - 6537648 GCAGGCU---------------------U-GGGUCGCGCGCGCCAUGGUCUGCGGACCAAGUGUGUUGACGCGUGCCGGGCGAGGCCGGUGCGGCGCUCGCGGGAGACUACUCCCCUAGG .....((---------------------(-(((.((((.(((((.(((((....))))).((((....))))(..((((......))))..)))))).))))((((....)))))))))) ( -50.60) >ppk.434 5305754 114 + 6181863 GCGGAUCUCUCCAUGCAGGCGUGCAUGGU-GGGUCGCACGCGCCAUGGUCUGUGGACCAAGUAUGUUGACGCGUACCGGGCGAG-----CUGGGUGCUCGCGGGAGACUACUCCCCUAGG ((((..(((.(((((((....))))))).-)))))))(((((.(((((((....))))).......)).))))).((..(((((-----(.....))))))(((((....)))))...)) ( -52.11) >pel.475 4951176 93 + 5888780 GCAGAUC--------------------GU-GGGUCGCACGCGCCAUGGCCUGUGGGCCAAGUGUGUUGACGCGUACCGGGCGAG-----C-GGGGGCUCGCGGGAGACUACUCCCCUAGG .......--------------------..-(.((((.((((((..(((((....))))).)))))))))).)...((..(((((-----(-....))))))(((((....)))))...)) ( -42.50) >pfo.2801 5321679 95 - 6438405 GCGGACG--------------------CUUGGGUCGCAUACGCCAUGAUCUGAGGAUCAAGUAUGUUGACGUAUGCCGGACGAG-----CUUGGCGCUCGUGGGAGACUACUCCCCUAGG .(((...--------------------..((.(((((((((....(((((....))))).))))).)))).))..))).(((((-----(.....))))))(((((....)))))..... ( -35.60) >pf5.3120 5888269 95 - 7074893 GCGGACU--------------------CUUGGGUCGCAUGCGCCAUGGUCUGUUGACCAAGUAUGUUGACGCAUGCCGGACGAG-----CGUGGCGCUCGUGGGAGACUACUCCCCUAGG .((((((--------------------....))))(((((((.(((((((....))))).......)).))))))))).(((((-----((...)))))))(((((....)))))..... ( -40.71) >consensus GCAGACU_____________________U_GGGUCGCACGCGCCAUGGUCUGUGGACCAAGUAUGUUGACGCGUGCCGGGCGAG_____CGCGGCGCUCGCGGGAGACUACUCCCCUAGG ..............................(.(((((((((....(((((....))))).))))).)))).)...((..(((((............)))))(((((....)))))...)) (-33.24 = -31.58 + -1.66)

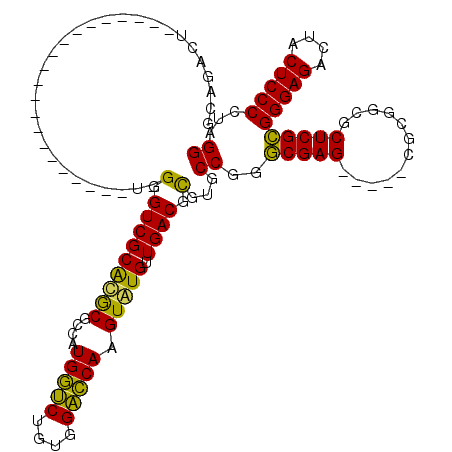
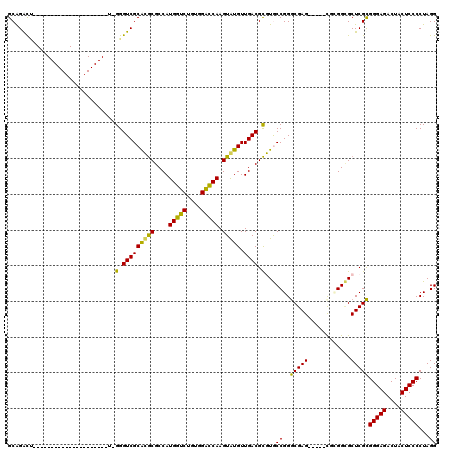
| Location | 5,197,019 – 5,197,115 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.84 |
| Mean single sequence MFE | -37.54 |
| Consensus MFE | -34.70 |
| Energy contribution | -32.09 |
| Covariance contribution | -2.61 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.42 |
| SVM RNA-class probability | 0.951994 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2746 5197019 96 + 6264404 CCCGGCACGCGUCAACACACUUGGUCCGCAGACCAUGGCGCGCGCGACCC-A---------------------AGCCUGCC-CAGACAGGCCA-AGGGUUUGACAAGACCUAUGACACGC ....((.(((((((.......(((((....)))))))))))).)).....-.---------------------.(((((..-....)))))((-.((((((....)))))).))...... ( -34.31) >pau.2882 5468998 96 + 6537648 CCCGGCACGCGUCAACACACUUGGUCCGCAGACCAUGGCGCGCGCGACCC-A---------------------AGCCUGCC-CAGACAGGCCA-AGGGUUUGACAAGACCUAUGACACGC ....((.(((((((.......(((((....)))))))))))).)).....-.---------------------.(((((..-....)))))((-.((((((....)))))).))...... ( -34.31) >ppk.434 5305789 117 - 6181863 CCCGGUACGCGUCAACAUACUUGGUCCACAGACCAUGGCGCGUGCGACCC-ACCAUGCACGCCUGCAUGGAGAGAUCCGCA-UAGACGGAUCG-AGGGUUUGACAAGACCUAUGACACGC ..((((((((((((.......(((((....)))))))))))))))(((((-.(((((((....)))))))...((((((..-....)))))).-.))))).................)). ( -48.61) >pel.475 4951210 97 - 5888780 CCCGGUACGCGUCAACACACUUGGCCCACAGGCCAUGGCGCGUGCGACCC-AC--------------------GAUCUGCG-CAGACAGGUCC-AGGGUUUGACAAGACCUAUGACACGC ..((((((((((((.......(((((....)))))))))))))))(((((-..--------------------((((((..-....)))))).-.))))).................)). ( -37.01) >pfo.2801 5321714 100 + 6438405 UCCGGCAUACGUCAACAUACUUGAUCCUCAGAUCAUGGCGUAUGCGACCCAAG--------------------CGUCCGCACCAGACGGAUCGCAGGGUUUGACAAGACCUAUGACACGC ..((((((((((((.......(((((....)))))))))))))))(((((..(--------------------(((((((....).)))).))).))))).................)). ( -33.81) >pf5.3120 5888304 99 + 7074893 UCCGGCAUGCGUCAACAUACUUGGUCAACAGACCAUGGCGCAUGCGACCCAAG--------------------AGUCCGCA-CAGACGGACCGCAGGGUUUGACAAGACCUAUGACACGC ..((((((((((((.......(((((....)))))))))))))))(((((..(--------------------.(((((..-....))))))...))))).................)). ( -37.21) >consensus CCCGGCACGCGUCAACACACUUGGUCCACAGACCAUGGCGCGUGCGACCC_A_____________________AGUCCGCA_CAGACAGACCG_AGGGUUUGACAAGACCUAUGACACGC ....((((((((((.......(((((....))))))))))))))).............................((((((....).)))))....((((((....))))))......... (-34.70 = -32.09 + -2.61)

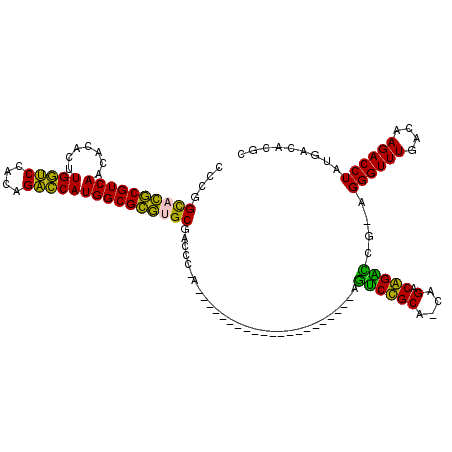
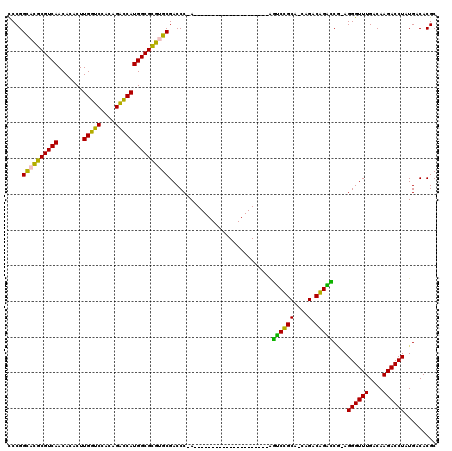
| Location | 5,197,019 – 5,197,115 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.84 |
| Mean single sequence MFE | -41.17 |
| Consensus MFE | -34.09 |
| Energy contribution | -32.37 |
| Covariance contribution | -1.72 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.14 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.631117 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2746 5197019 96 - 6264404 GCGUGUCAUAGGUCUUGUCAAACCCU-UGGCCUGUCUG-GGCAGGCU---------------------U-GGGUCGCGCGCGCCAUGGUCUGCGGACCAAGUGUGUUGACGCGUGCCGGG ((((((((........((...((((.-.(((((((...-.)))))))---------------------.-)))).))((((((..(((((....))))).))))))))))))))...... ( -43.40) >pau.2882 5468998 96 - 6537648 GCGUGUCAUAGGUCUUGUCAAACCCU-UGGCCUGUCUG-GGCAGGCU---------------------U-GGGUCGCGCGCGCCAUGGUCUGCGGACCAAGUGUGUUGACGCGUGCCGGG ((((((((........((...((((.-.(((((((...-.)))))))---------------------.-)))).))((((((..(((((....))))).))))))))))))))...... ( -43.40) >ppk.434 5305789 117 + 6181863 GCGUGUCAUAGGUCUUGUCAAACCCU-CGAUCCGUCUA-UGCGGAUCUCUCCAUGCAGGCGUGCAUGGU-GGGUCGCACGCGCCAUGGUCUGUGGACCAAGUAUGUUGACGCGUACCGGG ((((((((..((((.(((...((((.-.(((((((...-.)))))))...(((((((....))))))).-)))).))).).))).(((((....))))).......))))))))...... ( -47.80) >pel.475 4951210 97 + 5888780 GCGUGUCAUAGGUCUUGUCAAACCCU-GGACCUGUCUG-CGCAGAUC--------------------GU-GGGUCGCACGCGCCAUGGCCUGUGGGCCAAGUGUGUUGACGCGUACCGGG (((((((((((((((...........-)))))))((..-((.....)--------------------).-.))..((((((....(((((....))))).))))))))))))))...... ( -38.20) >pfo.2801 5321714 100 - 6438405 GCGUGUCAUAGGUCUUGUCAAACCCUGCGAUCCGUCUGGUGCGGACG--------------------CUUGGGUCGCAUACGCCAUGAUCUGAGGAUCAAGUAUGUUGACGUAUGCCGGA .((.(.((((.(((.......((((.(((.(((((.....)))))))--------------------)..)))).((((((....(((((....))))).)))))).))).))))))).. ( -36.80) >pf5.3120 5888304 99 - 7074893 GCGUGUCAUAGGUCUUGUCAAACCCUGCGGUCCGUCUG-UGCGGACU--------------------CUUGGGUCGCAUGCGCCAUGGUCUGUUGACCAAGUAUGUUGACGCAUGCCGGA ((((((..........((((.((((...(((((((...-.)))))))--------------------...)))).((((((....(((((....))))).)))))))))))))))).... ( -37.40) >consensus GCGUGUCAUAGGUCUUGUCAAACCCU_CGACCCGUCUG_UGCAGACU_____________________U_GGGUCGCACGCGCCAUGGUCUGUGGACCAAGUAUGUUGACGCGUGCCGGG ((((((((.............((((...(((((((.....))))))).......................)))).((((((....(((((....))))).))))))))))))))...... (-34.09 = -32.37 + -1.72)

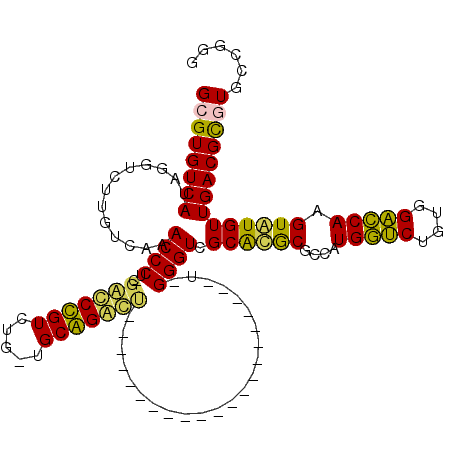
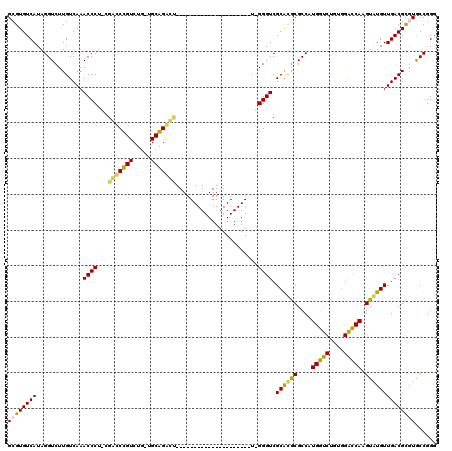
| Location | 5,197,059 – 5,197,155 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.36 |
| Mean single sequence MFE | -46.97 |
| Consensus MFE | -35.10 |
| Energy contribution | -32.44 |
| Covariance contribution | -2.66 |
| Combinations/Pair | 1.31 |
| Mean z-score | -3.41 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.87 |
| SVM RNA-class probability | 0.980956 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2746 5197059 96 + 6264404 CGCGCGACCC-A---------------------AGCCUGCC-CAGACAGGCCA-AGGGUUUGACAAGACCUAUGACACGCACGAUCUGACCCGGGGCGGGCAGAUGGCGCGUGUCAUGGU .....(((((-.---------------------.(((((..-....)))))..-.))))).........(((((((((((.(.(((((.((((...))))))))).).))))))))))). ( -48.20) >pau.2882 5469038 96 + 6537648 CGCGCGACCC-A---------------------AGCCUGCC-CAGACAGGCCA-AGGGUUUGACAAGACCUAUGACACGCACGAUCUGACCCGGGGCGGGCAGAUGGCGCGUGUCAUGGU .....(((((-.---------------------.(((((..-....)))))..-.))))).........(((((((((((.(.(((((.((((...))))))))).).))))))))))). ( -48.20) >ppk.434 5305829 116 - 6181863 CGUGCGACCC-ACCAUGCACGCCUGCAUGGAGAGAUCCGCA-UAGACGGAUCG-AGGGUUUGACAAGACCUAUGACACGCAC-ACCUUACCCGGGGCGGGGAGGCUGUACGUGUCAUAGC .((..(((((-.(((((((....)))))))...((((((..-....)))))).-.)))))..)).....((((((((((.((-(((((.((((...)))))))).))).)))))))))). ( -56.30) >pel.475 4951250 96 - 5888780 CGUGCGACCC-AC--------------------GAUCUGCG-CAGACAGGUCC-AGGGUUUGACAAGACCUAUGACACGCAC-ACCUUACCCGGGGCGGGGAGGUCGUACGUGUCAUAGC .((..(((((-..--------------------((((((..-....)))))).-.)))))..)).....((((((((((.((-(((((.((((...))))))))).)).)))))))))). ( -42.60) >pfo.2801 5321754 99 + 6438405 UAUGCGACCCAAG--------------------CGUCCGCACCAGACGGAUCGCAGGGUUUGACAAGACCUAUGACACGCAC-ACCUUACCCGGGGCGGGAAGGCUGUACGUGUCAUAGC .....(((((..(--------------------(((((((....).)))).))).))))).........((((((((((.((-(((((.((((...)))))))).))).)))))))))). ( -43.80) >pf5.3120 5888344 98 + 7074893 CAUGCGACCCAAG--------------------AGUCCGCA-CAGACGGACCGCAGGGUUUGACAAGACCUAUGACACGCAC-ACCUUACCCGGGGCGGGAAGGCUGUACGUGUCAUAGC .....(((((..(--------------------.(((((..-....))))))...))))).........((((((((((.((-(((((.((((...)))))))).))).)))))))))). ( -42.70) >consensus CGUGCGACCC_A_____________________AGUCCGCA_CAGACAGACCG_AGGGUUUGACAAGACCUAUGACACGCAC_ACCUUACCCGGGGCGGGAAGGCUGUACGUGUCAUAGC .....(((((........................((((((....).)))))....))))).........((((((((((.....((((.((((...)))))))).....)))))))))). (-35.10 = -32.44 + -2.66)

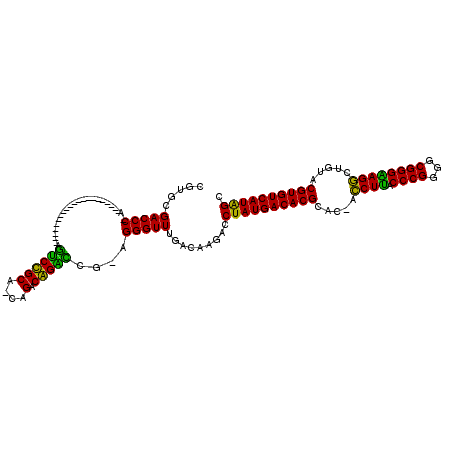
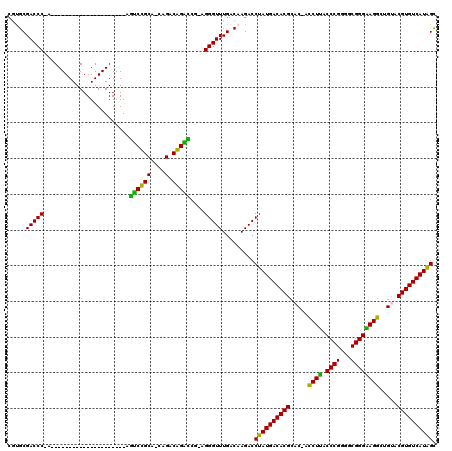
| Location | 5,197,059 – 5,197,155 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.36 |
| Mean single sequence MFE | -51.37 |
| Consensus MFE | -45.88 |
| Energy contribution | -44.14 |
| Covariance contribution | -1.75 |
| Combinations/Pair | 1.28 |
| Mean z-score | -4.26 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.43 |
| SVM RNA-class probability | 0.953161 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2746 5197059 96 - 6264404 ACCAUGACACGCGCCAUCUGCCCGCCCCGGGUCAGAUCGUGCGUGUCAUAGGUCUUGUCAAACCCU-UGGCCUGUCUG-GGCAGGCU---------------------U-GGGUCGCGCG ((((((((((((((.(((((((((...)))).))))).))))))))))).)))...((...((((.-.(((((((...-.)))))))---------------------.-))))...)). ( -53.90) >pau.2882 5469038 96 - 6537648 ACCAUGACACGCGCCAUCUGCCCGCCCCGGGUCAGAUCGUGCGUGUCAUAGGUCUUGUCAAACCCU-UGGCCUGUCUG-GGCAGGCU---------------------U-GGGUCGCGCG ((((((((((((((.(((((((((...)))).))))).))))))))))).)))...((...((((.-.(((((((...-.)))))))---------------------.-))))...)). ( -53.90) >ppk.434 5305829 116 + 6181863 GCUAUGACACGUACAGCCUCCCCGCCCCGGGUAAGGU-GUGCGUGUCAUAGGUCUUGUCAAACCCU-CGAUCCGUCUA-UGCGGAUCUCUCCAUGCAGGCGUGCAUGGU-GGGUCGCACG .((((((((((((((.(((.((((...))))..))))-)))))))))))))....(((...((((.-.(((((((...-.)))))))...(((((((....))))))).-)))).))).. ( -56.50) >pel.475 4951250 96 + 5888780 GCUAUGACACGUACGACCUCCCCGCCCCGGGUAAGGU-GUGCGUGUCAUAGGUCUUGUCAAACCCU-GGACCUGUCUG-CGCAGAUC--------------------GU-GGGUCGCACG .(((((((((((((.((((.((((...))))..))))-)))))))))))))....(((...((((.-.((.((((...-.)))).))--------------------..-)))).))).. ( -41.80) >pfo.2801 5321754 99 - 6438405 GCUAUGACACGUACAGCCUUCCCGCCCCGGGUAAGGU-GUGCGUGUCAUAGGUCUUGUCAAACCCUGCGAUCCGUCUGGUGCGGACG--------------------CUUGGGUCGCAUA .((((((((((((((.((((((((...)))).)))))-)))))))))))))....(((...((((.(((.(((((.....)))))))--------------------)..)))).))).. ( -51.40) >pf5.3120 5888344 98 - 7074893 GCUAUGACACGUACAGCCUUCCCGCCCCGGGUAAGGU-GUGCGUGUCAUAGGUCUUGUCAAACCCUGCGGUCCGUCUG-UGCGGACU--------------------CUUGGGUCGCAUG .((((((((((((((.((((((((...)))).)))))-)))))))))))))....(((...((((...(((((((...-.)))))))--------------------...)))).))).. ( -50.70) >consensus GCUAUGACACGUACAACCUCCCCGCCCCGGGUAAGGU_GUGCGUGUCAUAGGUCUUGUCAAACCCU_CGACCCGUCUG_UGCAGACU_____________________U_GGGUCGCACG .(((((((((((((.(((((((((...)))).))))).)))))))))))))....(((...((((...(((((((.....))))))).......................)))).))).. (-45.88 = -44.14 + -1.75)

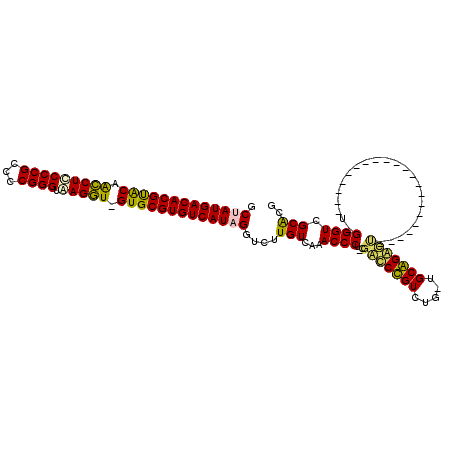
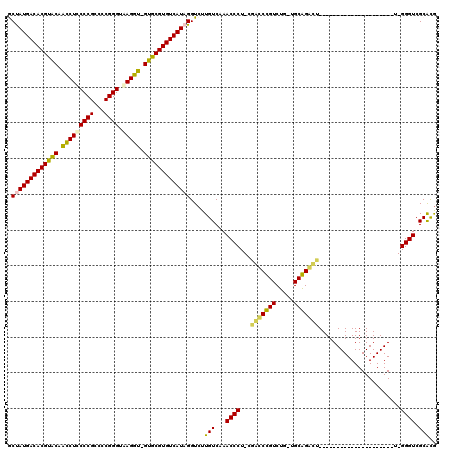
| Location | 5,197,070 – 5,197,185 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.26 |
| Mean single sequence MFE | -50.48 |
| Consensus MFE | -43.81 |
| Energy contribution | -41.15 |
| Covariance contribution | -2.66 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.81 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.61 |
| SVM RNA-class probability | 0.967086 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2746 5197070 115 + 6264404 --AGCCUGCC-CAGACAGGCCA-AGGGUUUGACAAGACCUAUGACACGCACGAUCUGACCCGGGGCGGGCAGAUGGCGCGUGUCAUGGUGAUUGGUCGACCCGCCCCU-UAGGAAGCCUG --.(((((..-....)))))..-.(((((.(((....((((((((((((.(.(((((.((((...))))))))).).))))))))))).)....))))))))((((..-..))..))... ( -53.50) >pau.2882 5469049 113 + 6537648 --AGCCUGCC-CAGACAGGCCA-AGGGUUUGACAAGACCUAUGACACGCACGAUCUGACCCGGGGCGGGCAGAUGGCGCGUGUCAUGGUGAUUGGUCGACCCGCCCCU-UAGGAAGCC-- --.(((((..-....)))))..-.(((((.(((....((((((((((((.(.(((((.((((...))))))))).).))))))))))).)....))))))))((((..-..))..)).-- ( -53.50) >ppk.434 5305859 113 - 6181863 GAGAUCCGCA-UAGACGGAUCG-AGGGUUUGACAAGACCUAUGACACGCAC-ACCUUACCCGGGGCGGGGAGGCUGUACGUGUCAUAGCCGU--GUCGACCCGCCCCCGUAGGAAUCC-- ..((((((..-....)))))).-.(((((.((((....((((((((((.((-(((((.((((...)))))))).))).))))))))))...)--)))))))).((......)).....-- ( -50.60) >pel.475 4951262 111 - 5888780 --GAUCUGCG-CAGACAGGUCC-AGGGUUUGACAAGACCUAUGACACGCAC-ACCUUACCCGGGGCGGGGAGGUCGUACGUGUCAUAGCCGU--GUCGACCCGCCCCCGUAGGAAUCC-- --((((((..-....)))))).-.(((((.((((....((((((((((.((-(((((.((((...))))))))).)).))))))))))...)--)))))))).((......)).....-- ( -47.40) >pfo.2801 5321767 112 + 6438405 --CGUCCGCACCAGACGGAUCGCAGGGUUUGACAAGACCUAUGACACGCAC-ACCUUACCCGGGGCGGGAAGGCUGUACGUGUCAUAGCCGU--GUCGACCCGCCCCU-UAGGAAAUC-- --.((((((....).))))).((.(((((.((((....((((((((((.((-(((((.((((...)))))))).))).))))))))))...)--))))))))))((..-..)).....-- ( -47.90) >pf5.3120 5888357 111 + 7074893 --AGUCCGCA-CAGACGGACCGCAGGGUUUGACAAGACCUAUGACACGCAC-ACCUUACCCGGGGCGGGAAGGCUGUACGUGUCAUAGCCGU--GUCGACCCGCCCCU-UAGGAAAAC-- --.(((((..-....))))).((.(((((.((((....((((((((((.((-(((((.((((...)))))))).))).))))))))))...)--))))))))))((..-..)).....-- ( -50.00) >consensus __AGUCCGCA_CAGACAGACCG_AGGGUUUGACAAGACCUAUGACACGCAC_ACCUUACCCGGGGCGGGAAGGCUGUACGUGUCAUAGCCGU__GUCGACCCGCCCCU_UAGGAAACC__ ...((((((....).)))))....(((((.(((.....((((((((((.....((((.((((...)))))))).....))))))))))......))))))))...((....))....... (-43.81 = -41.15 + -2.66)

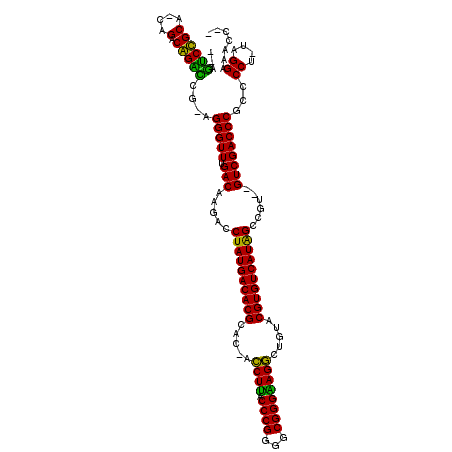
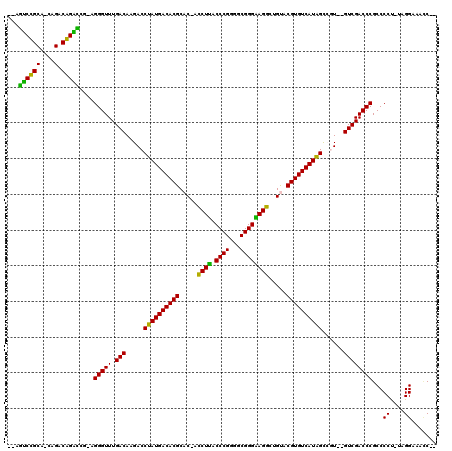
| Location | 5,197,070 – 5,197,185 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.26 |
| Mean single sequence MFE | -56.92 |
| Consensus MFE | -54.08 |
| Energy contribution | -52.12 |
| Covariance contribution | -1.97 |
| Combinations/Pair | 1.26 |
| Mean z-score | -4.06 |
| Structure conservation index | 0.95 |
| SVM decision value | 2.52 |
| SVM RNA-class probability | 0.994882 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2746 5197070 115 - 6264404 CAGGCUUCCUA-AGGGGCGGGUCGACCAAUCACCAUGACACGCGCCAUCUGCCCGCCCCGGGUCAGAUCGUGCGUGUCAUAGGUCUUGUCAAACCCU-UGGCCUGUCUG-GGCAGGCU-- ...(((((...-.)))))((((.(((.....((((((((((((((.(((((((((...)))).))))).))))))))))).)))...)))..)))).-.(((((((...-.)))))))-- ( -58.80) >pau.2882 5469049 113 - 6537648 --GGCUUCCUA-AGGGGCGGGUCGACCAAUCACCAUGACACGCGCCAUCUGCCCGCCCCGGGUCAGAUCGUGCGUGUCAUAGGUCUUGUCAAACCCU-UGGCCUGUCUG-GGCAGGCU-- --.(((((...-.)))))((((.(((.....((((((((((((((.(((((((((...)))).))))).))))))))))).)))...)))..)))).-.(((((((...-.)))))))-- ( -58.80) >ppk.434 5305859 113 + 6181863 --GGAUUCCUACGGGGGCGGGUCGAC--ACGGCUAUGACACGUACAGCCUCCCCGCCCCGGGUAAGGU-GUGCGUGUCAUAGGUCUUGUCAAACCCU-CGAUCCGUCUA-UGCGGAUCUC --.(.((((....)))))((((.(((--(.((((((((((((((((.(((.((((...))))..))))-)))))))))))).))).))))..)))).-.(((((((...-.))))))).. ( -56.90) >pel.475 4951262 111 + 5888780 --GGAUUCCUACGGGGGCGGGUCGAC--ACGGCUAUGACACGUACGACCUCCCCGCCCCGGGUAAGGU-GUGCGUGUCAUAGGUCUUGUCAAACCCU-GGACCUGUCUG-CGCAGAUC-- --......((.((.((((((((((((--(.(((((((((((((((.((((.((((...))))..))))-)))))))))))).))).)))).......-.))))))))).-)).))...-- ( -53.90) >pfo.2801 5321767 112 - 6438405 --GAUUUCCUA-AGGGGCGGGUCGAC--ACGGCUAUGACACGUACAGCCUUCCCGCCCCGGGUAAGGU-GUGCGUGUCAUAGGUCUUGUCAAACCCUGCGAUCCGUCUGGUGCGGACG-- --....(((((-..((((((((((.(--(.((((((((((((((((.((((((((...)))).)))))-)))))))))))))(......)...)).))))))))))))..)).)))..-- ( -55.60) >pf5.3120 5888357 111 - 7074893 --GUUUUCCUA-AGGGGCGGGUCGAC--ACGGCUAUGACACGUACAGCCUUCCCGCCCCGGGUAAGGU-GUGCGUGUCAUAGGUCUUGUCAAACCCUGCGGUCCGUCUG-UGCGGACU-- --.....((..-..))((((((.(((--(.((((((((((((((((.((((((((...)))).)))))-)))))))))))).))).))))..)))).))(((((((...-.)))))))-- ( -57.50) >consensus __GGAUUCCUA_AGGGGCGGGUCGAC__ACGGCUAUGACACGUACAACCUCCCCGCCCCGGGUAAGGU_GUGCGUGUCAUAGGUCUUGUCAAACCCU_CGACCCGUCUG_UGCAGACU__ .....((((....)))).((((.(((....(((((((((((((((.(((((((((...)))).))))).))))))))))).))))..)))..))))...(((((((.....))))))).. (-54.08 = -52.12 + -1.97)
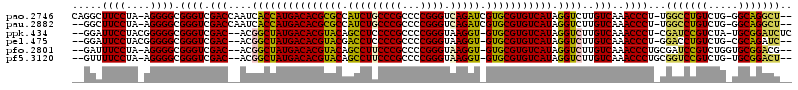

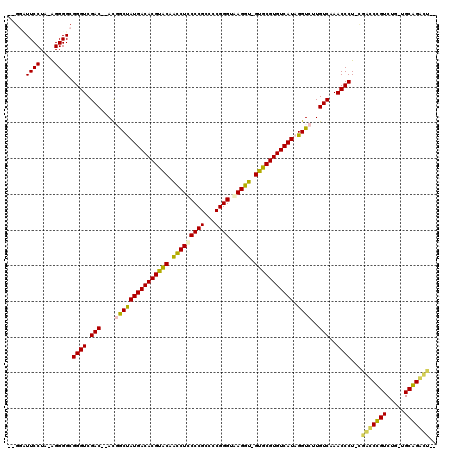
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:06:33 2007