
| Sequence ID | pao.2802 |
|---|---|
| Location | 5,308,684 – 5,308,971 |
| Length | 287 |
| Max. P | 0.997251 |

| Location | 5,308,684 – 5,308,802 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.24 |
| Mean single sequence MFE | -38.44 |
| Consensus MFE | -32.67 |
| Energy contribution | -30.05 |
| Covariance contribution | -2.62 |
| Combinations/Pair | 1.55 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.15 |
| SVM RNA-class probability | 0.922737 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
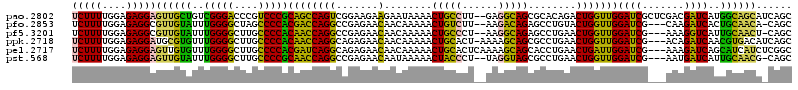
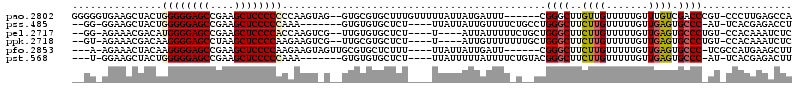
>pao.2802 5308684 118 + 6264404 UCUUUUGGAGAGGAGUUGCUGUCGGGACCCGUCCCGCAGCCAGUCGGAAGAAGAAUAAAACUGCCUU--GAGGCAGCGCACAGACUGGUUGGAUCGCUCGACGAUCAUGGCAGCAUCAGC (((((....)))))(.(((((((((((....)))).(((((((((....)).........(((((..--..)))))........)))))))(((((.....)))))..))))))).)... ( -46.50) >pfo.2853 5425324 114 + 6438405 UCUUUUGGAGAGGCGUUGUAUUUGGGGCUAGCCCCACGACCAGGCCGAGAACAACAAAAACUGUCUU--AAGACAGAGCCUGUACUGGUUGGAUCG---CAAGAUCACUGCAACA-CAGC ........(.(((((((((...(((((....)))))((.......))...))))).....(((((..--..))))).)))).).(((((((((((.---...))))....)))).-))). ( -39.20) >pf5.3201 6041057 114 + 7074893 UCUUUUGGAGAGGCGUUGUAUUUGGGGCUUGCCCCACAACCAGGCCGAGAACAACAAAAACUGCCCU--AAGGCAGAGCCUGAACUGGUUGGAUCG---AAAGGUCAUUGCAACU-CAGC .......(((..(((.......(((((....)))))(((((((..((.(.....).....(((((..--..)))))....))..)))))))((((.---...))))..)))..))-)... ( -39.70) >ppk.2718 5338372 116 + 6181863 UCUUUUGGAGAGGAUGCGUGUUUGGGGCUUGCCCCACAACCAGGCAGAGAACAACAAAAACUGCACU-AAAAGCAGCGCCUGAACUGGUUGGAUCG---ACAGAUCAACGUGACAUCAGC ............(((((((((((((((....)))))(((((((.(((.(.....).....((((...-....))))...)))..)))))))((((.---...)))))))))).))))... ( -34.90) >pel.2717 5029948 117 + 5888780 UCUUUUGGAGAGGAGUUGUGUUUGGGGCUUGCCCCACGAUCAGGCAGAGAACAACAAAAACUGCACUCAAAAGCAGCACCUGAACUGAUUGGAUCG---AAAGAUCAGCAUCAUCUCGGC .......((((.(((((.....(((((....)))))(((((((.(((.(.....).....((((........))))...)))..)))))))((((.---...))))))).)).))))... ( -36.30) >pst.568 5350273 114 - 6397126 UCUUUUGGAGAGGAGUUGUAUUUGGGGCUUGCCCCGCAACCAGGCCGAGAACAAUAAAAACUACCCU--UAGGUAGCGCCUGAACUGGUUGGAUCG---AAUGAUCAUUGCAACG-CAGC (((((....)))))((((((...((((....)))).(((((((..((.(...........(((((..--..)))))..).))..)))))))((((.---...))))..)))))).-.... ( -34.02) >consensus UCUUUUGGAGAGGAGUUGUAUUUGGGGCUUGCCCCACAACCAGGCCGAGAACAACAAAAACUGCCCU__AAGGCAGCGCCUGAACUGGUUGGAUCG___AAAGAUCAUUGCAACA_CAGC (((((....)))))((((((..(((((....)))))((((((((.......)........(((((......)))))........)))))))((((.......))))..))))))...... (-32.67 = -30.05 + -2.62)
| Location | 5,308,724 – 5,308,842 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.06 |
| Mean single sequence MFE | -34.12 |
| Consensus MFE | -20.65 |
| Energy contribution | -21.02 |
| Covariance contribution | 0.37 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.07 |
| SVM RNA-class probability | 0.566814 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
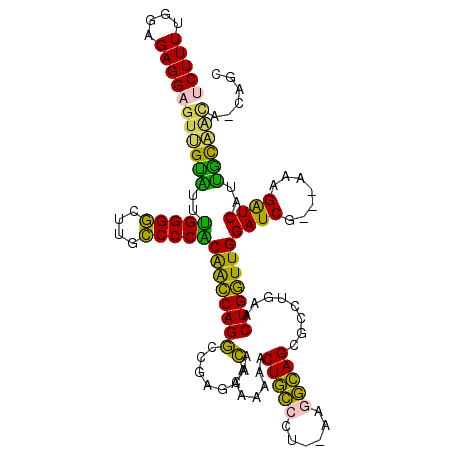
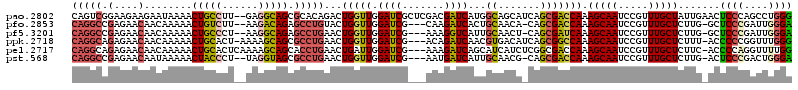
>pao.2802 5308724 118 + 6264404 CAGUCGGAAGAAGAAUAAAACUGCCUU--GAGGCAGCGCACAGACUGGUUGGAUCGCUCGACGAUCAUGGCAGCAUCAGCGACCAAAGCAAUCCGUUUGCUAUUGAACUCCCAGCCUGGG ...((....)).........(((((..--..)))))....(((.((((..((((((.....))))).(((..((....))..))).(((((.....)))))......)..)))).))).. ( -35.90) >pfo.2853 5425364 113 + 6438405 CAGGCCGAGAACAACAAAAACUGUCUU--AAGACAGAGCCUGUACUGGUUGGAUCG---CAAGAUCACUGCAACA-CAGCGACCAAAGCAAUCCGUUUGCUCUUG-GCUCCCGAUUGGGA ..((((((((.((((.....(((((..--..)))))....(((..((((((((((.---...)))).(((.....-)))))))))..)))....).))).)))))-)))(((....))). ( -40.80) >pf5.3201 6041097 113 + 7074893 CAGGCCGAGAACAACAAAAACUGCCCU--AAGGCAGAGCCUGAACUGGUUGGAUCG---AAAGGUCAUUGCAACU-CAGCGAUCAAAGCAAUCCGUUUGCUCUUG-GCUCCCGAUUGGGA ..((((((((..........(((((..--..))))).((..((((.(((((((((.---...))))(((((....-..))))).....))))).)))))))))))-)))(((....))). ( -41.00) >ppk.2718 5338412 115 + 6181863 CAGGCAGAGAACAACAAAAACUGCACU-AAAAGCAGCGCCUGAACUGGUUGGAUCG---ACAGAUCAACGUGACAUCAGCGGCCAAAGCAAUCCGUUUGCUCUUU-ACCCCCGGUUUGGG (((((.(....)........((((...-....)))).)))))...((((((((((.---...))))....((....)).)))))).(((((.....)))))....-...(((.....))) ( -27.60) >pel.2717 5029988 116 + 5888780 CAGGCAGAGAACAACAAAAACUGCACUCAAAAGCAGCACCUGAACUGAUUGGAUCG---AAAGAUCAGCAUCAUCUCGGCGACCAAAGCAAUCCGUUUGCUCUUC-ACCCCAGGUUUUGG ...((((.............)))).........(((.(((((...((((..((((.---...))))...))))....((.((....(((((.....)))))..))-.)).))))).))). ( -28.82) >pst.568 5350313 113 - 6397126 CAGGCCGAGAACAAUAAAAACUACCCU--UAGGUAGCGCCUGAACUGGUUGGAUCG---AAUGAUCAUUGCAACG-CAGCGACCAAAGCAAUCCGUUUGCUCUUG-ACUCCCGACUGGGA (((((.(....)........(((((..--..))))).)))))...((((((((((.---...)))).((((...)-))))))))).(((((.....)))))....-..((((....)))) ( -30.60) >consensus CAGGCCGAGAACAACAAAAACUGCCCU__AAGGCAGCGCCUGAACUGGUUGGAUCG___AAAGAUCAUUGCAACA_CAGCGACCAAAGCAAUCCGUUUGCUCUUG_ACUCCCGAUUGGGA (((((.(....)........(((((......))))).)))))...(((((.((((.......))))...((.......))))))).(((((.....))))).......((((....)))) (-20.65 = -21.02 + 0.37)

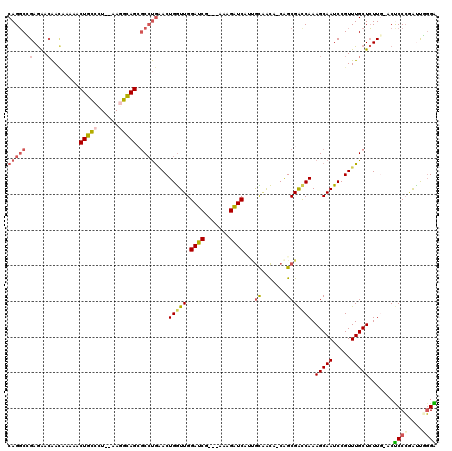
| Location | 5,308,724 – 5,308,842 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.06 |
| Mean single sequence MFE | -40.68 |
| Consensus MFE | -23.20 |
| Energy contribution | -21.68 |
| Covariance contribution | -1.52 |
| Combinations/Pair | 1.45 |
| Mean z-score | -2.12 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.98 |
| SVM RNA-class probability | 0.894386 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2802 5308724 118 - 6264404 CCCAGGCUGGGAGUUCAAUAGCAAACGGAUUGCUUUGGUCGCUGAUGCUGCCAUGAUCGUCGAGCGAUCCAACCAGUCUGUGCGCUGCCUC--AAGGCAGUUUUAUUCUUCUUCCGACUG ..(((..((((((...((((((..((((((((..(((((((((((((.(......).)))).))))).)))).))))))))))((((((..--..))))))..))))...)))))).))) ( -43.20) >pfo.2853 5425364 113 - 6438405 UCCCAAUCGGGAGC-CAAGAGCAAACGGAUUGCUUUGGUCGCUG-UGUUGCAGUGAUCUUG---CGAUCCAACCAGUACAGGCUCUGUCUU--AAGACAGUUUUUGUUGUUCUCGGCCUG ((((....))))((-(.(((((((..(((((((...((((((((-.....))))))))..)---))))))........((((..(((((..--..)))))..))))))))))).)))... ( -50.70) >pf5.3201 6041097 113 - 7074893 UCCCAAUCGGGAGC-CAAGAGCAAACGGAUUGCUUUGAUCGCUG-AGUUGCAAUGACCUUU---CGAUCCAACCAGUUCAGGCUCUGCCUU--AGGGCAGUUUUUGUUGUUCUCGGCCUG ((((....))))((-(.(((((((..((((((((((((..((((-.((((...(((....)---))...))))))))...(((...)))))--))))))))))...))))))).)))... ( -39.60) >ppk.2718 5338412 115 - 6181863 CCCAAACCGGGGGU-AAAGAGCAAACGGAUUGCUUUGGCCGCUGAUGUCACGUUGAUCUGU---CGAUCCAACCAGUUCAGGCGCUGCUUUU-AGUGCAGUUUUUGUUGUUCUCUGCCUG (((.....)))(((-(.(((((((..(((((((...(((((((((......(((((((...---.))).))))....))).)))..)))...-...)))))))...))))))).)))).. ( -35.10) >pel.2717 5029988 116 - 5888780 CCAAAACCUGGGGU-GAAGAGCAAACGGAUUGCUUUGGUCGCCGAGAUGAUGCUGAUCUUU---CGAUCCAAUCAGUUCAGGUGCUGCUUUUGAGUGCAGUUUUUGUUGUUCUCUGCCUG (((.....)))(((-..(((((((..(((((((...((.(((((((.((((...((((...---.))))..)))).))).))))))(((....))))))))))...)))))))..))).. ( -36.20) >pst.568 5350313 113 + 6397126 UCCCAGUCGGGAGU-CAAGAGCAAACGGAUUGCUUUGGUCGCUG-CGUUGCAAUGAUCAUU---CGAUCCAACCAGUUCAGGCGCUACCUA--AGGGUAGUUUUUAUUGUUCUCGGCCUG ((((....))))((-(.(((((((..((((((...((((((.((-(...))).))))))..---)))))).............((((((..--..)))))).....))))))).)))... ( -39.30) >consensus CCCAAACCGGGAGU_CAAGAGCAAACGGAUUGCUUUGGUCGCUG_UGUUGCAAUGAUCUUU___CGAUCCAACCAGUUCAGGCGCUGCCUU__AGGGCAGUUUUUGUUGUUCUCGGCCUG (((.....)))(((...(((((((.....)))))))....)))...........((((.......)))).........((((((((((((....))))))..............)))))) (-23.20 = -21.68 + -1.52)

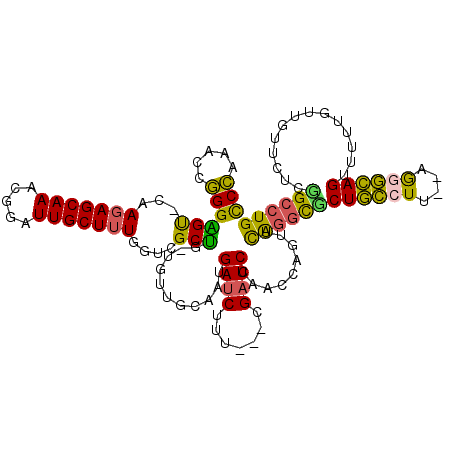
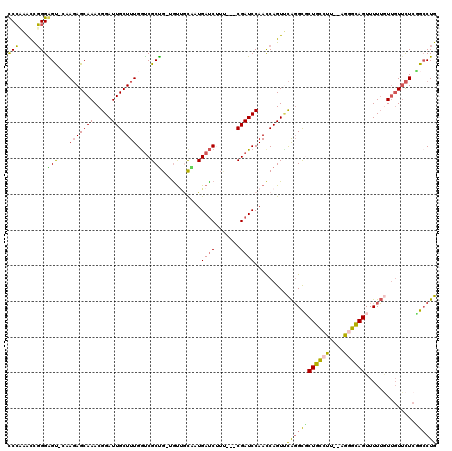
| Location | 5,308,735 – 5,308,851 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 79.15 |
| Mean single sequence MFE | -34.10 |
| Consensus MFE | -25.77 |
| Energy contribution | -24.80 |
| Covariance contribution | -0.97 |
| Combinations/Pair | 1.31 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.831318 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2802 5308735 116 + 6264404 AGAAUAAAACUGCCUU-GAGGCAGCGCACAGACUGGUUGGAUCGCUCGACGAUCAUGGCAGCAUCAGCGACCAAAGCAAUCCGUUUGCUAUUGAACUCCCAGCCUGGGAGAU--AUCCC .........(((((..-..)))))....(((((.((((((((((.....))))).(((..((....))..)))...))))).)))))........(((((.....)))))..--..... ( -37.50) >pfo.2853 5425375 109 + 6438405 CAACAAAAACUGUCUU-AAGACAGAGCCUGUACUGGUUGGAUCG---CAAGAUCACUGCAACA-CAGCGACCAAAGCAAUCCGUUUGCUCUUG-GCUCCCGAUUGGGAGG----GUCAU .........(((((..-..))))).((.(((....((..((((.---...))))...))...)-))))((((..(((((.....)))))....-.(((((....))))))----))).. ( -37.90) >pf5.3201 6041108 109 + 7074893 CAACAAAAACUGCCCU-AAGGCAGAGCCUGAACUGGUUGGAUCG---AAAGGUCAUUGCAACU-CAGCGAUCAAAGCAAUCCGUUUGCUCUUG-GCUCCCGAUUGGGAGG----GUCAU .........(((((..-..))))).((.(((....((..((((.---...))))...))...)-))))((((..(((((.....)))))....-.(((((....))))))----))).. ( -36.30) >ppk.2718 5338423 114 + 6181863 CAACAAAAACUGCACUAAAAGCAGCGCCUGAACUGGUUGGAUCG---ACAGAUCAACGUGACAUCAGCGGCCAAAGCAAUCCGUUUGCUCUUU-ACCCCCGGUUUGGGGG-UCGUCCAC .........((((.......))))(.((..(((((((((((((.---...))))..(((.......)))..)))(((((.....)))))....-....))))))..)).)-........ ( -29.80) >pss.485 5152724 110 - 6093698 CAACAAAAACUACCCU-UAGGUAGCGCCUGAACUGGUUGGAUCG---AAUGAUCAUUGCAACG-CAGCGACCAAAGCAAUCCGUUUGCUCUUG-ACUCCCGACUGGGAGG-U--UUCAC .........(((((..-..)))))....((((.((((((((((.---...)))).((((...)-))))))))).(((((.....)))))....-.(((((....))))).-.--)))). ( -29.60) >pst.568 5350324 110 - 6397126 CAAUAAAAACUACCCU-UAGGUAGCGCCUGAACUGGUUGGAUCG---AAUGAUCAUUGCAACG-CAGCGACCAAAGCAAUCCGUUUGCUCUUG-ACUCCCGACUGGGAGG-G--UUCAC .........(((((..-..)))))....(((((((((((((((.---...)))).((((...)-))))))))..(((((.....)))))....-.(((((....))))))-)--)))). ( -33.50) >consensus CAACAAAAACUGCCCU_AAGGCAGCGCCUGAACUGGUUGGAUCG___AAAGAUCAUUGCAACA_CAGCGACCAAAGCAAUCCGUUUGCUCUUG_ACUCCCGACUGGGAGG_U__UUCAC .........(((((.....))))).........(((((.((((.......))))...((.......))))))).(((((.....)))))......(((((....))))).......... (-25.77 = -24.80 + -0.97)

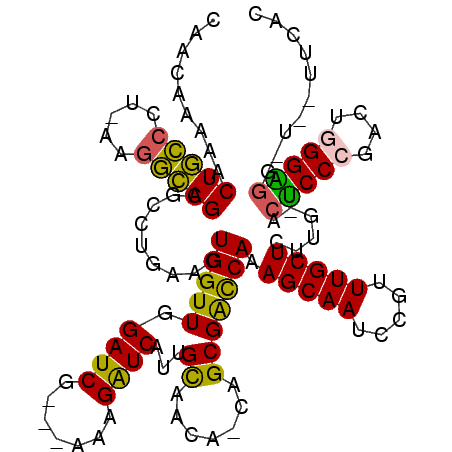
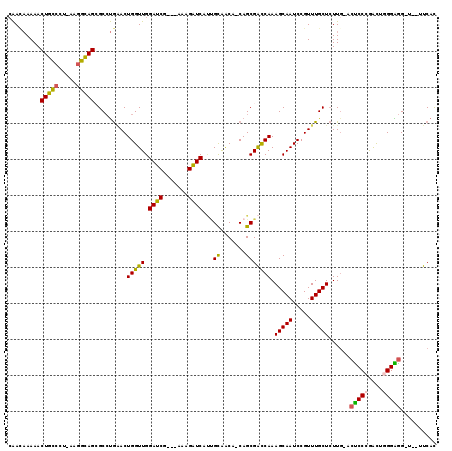
| Location | 5,308,735 – 5,308,851 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 79.15 |
| Mean single sequence MFE | -37.37 |
| Consensus MFE | -23.52 |
| Energy contribution | -24.33 |
| Covariance contribution | 0.81 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.49 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.626144 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2802 5308735 116 - 6264404 GGGAU--AUCUCCCAGGCUGGGAGUUCAAUAGCAAACGGAUUGCUUUGGUCGCUGAUGCUGCCAUGAUCGUCGAGCGAUCCAACCAGUCUGUGCGCUGCCUC-AAGGCAGUUUUAUUCU ((((.--...))))(((((((..((......))....(((((((((((((.((....)).))))........)))))))))..)))))))....((((((..-..))))))........ ( -44.90) >pfo.2853 5425375 109 - 6438405 AUGAC----CCUCCCAAUCGGGAGC-CAAGAGCAAACGGAUUGCUUUGGUCGCUG-UGUUGCAGUGAUCUUG---CGAUCCAACCAGUACAGGCUCUGUCUU-AAGACAGUUUUUGUUG .....----.(((((....))))).-(((((((....(((((((...((((((((-.....))))))))..)---))))))...............((((..-..)))))))))))... ( -41.10) >pf5.3201 6041108 109 - 7074893 AUGAC----CCUCCCAAUCGGGAGC-CAAGAGCAAACGGAUUGCUUUGAUCGCUG-AGUUGCAAUGACCUUU---CGAUCCAACCAGUUCAGGCUCUGCCUU-AGGGCAGUUUUUGUUG .....----.(((((....))))).-(((((((....((((((.((.(((((..(-.(((.....))))...---))))).)).))))))..((((((...)-))))).)))))))... ( -31.90) >ppk.2718 5338423 114 - 6181863 GUGGACGA-CCCCCAAACCGGGGGU-AAAGAGCAAACGGAUUGCUUUGGCCGCUGAUGUCACGUUGAUCUGU---CGAUCCAACCAGUUCAGGCGCUGCUUUUAGUGCAGUUUUUGUUG ((((.(.(-(((((.....))))))-..(((((((.....))))))).))))).........(((((((...---.))).)))).....((((.(((((.......))))).))))... ( -35.20) >pss.485 5152724 110 + 6093698 GUGAA--A-CCUCCCAGUCGGGAGU-CAAGAGCAAACGGAUUGCUUUGGUCGCUG-CGUUGCAAUGAUCAUU---CGAUCCAACCAGUUCAGGCGCUACCUA-AGGGUAGUUUUUGUUG .(((.--.-.(((((....))))))-))..((((((.((((((...((((((.((-(...))).))))))..---)))))).............((((((..-..)))))).)))))). ( -35.10) >pst.568 5350324 110 + 6397126 GUGAA--C-CCUCCCAGUCGGGAGU-CAAGAGCAAACGGAUUGCUUUGGUCGCUG-CGUUGCAAUGAUCAUU---CGAUCCAACCAGUUCAGGCGCUACCUA-AGGGUAGUUUUUAUUG .((((--(-.(((((....))))).-...........((((((...((((((.((-(...))).))))))..---)))))).....)))))...((((((..-..))))))........ ( -36.00) >consensus GUGAA__A_CCUCCCAAUCGGGAGU_CAAGAGCAAACGGAUUGCUUUGGUCGCUG_UGUUGCAAUGAUCUUU___CGAUCCAACCAGUUCAGGCGCUGCCUU_AGGGCAGUUUUUGUUG ..((......(((((....)))))....(((((((.....)))))))..))((((..((((....((((.......)))))))))))).((((..(((((.....)))))..))))... (-23.52 = -24.33 + 0.81)

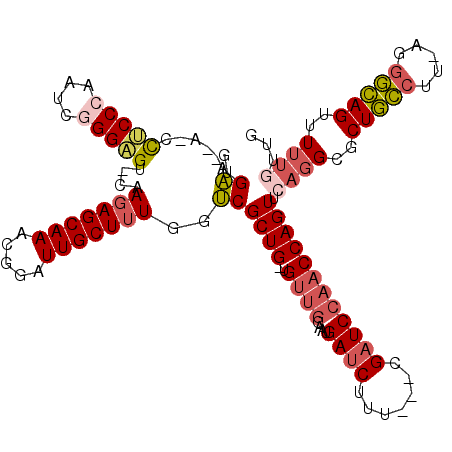
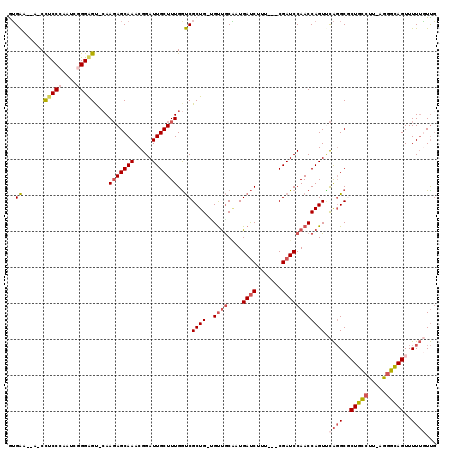
| Location | 5,308,851 – 5,308,962 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 69.32 |
| Mean single sequence MFE | -34.09 |
| Consensus MFE | -21.54 |
| Energy contribution | -24.37 |
| Covariance contribution | 2.83 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.39 |
| Structure conservation index | 0.63 |
| SVM decision value | 2.70 |
| SVM RNA-class probability | 0.996449 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2802 5308851 111 + 6264404 UGAAGCGACUGGCUCAAGGG-ACGGGUCGACAAACAAAAACAACAAGCCCG------AAAUCAUAAUAAAAACAAAGCACGCAC--CUACUUGGGGGGGAGCUUCGGCUCCCCCAGUAGC ....((.((((.((((((((-.(((((...................)))))------...................(....).)--)..))))))(((((((....))))))))))).)) ( -36.21) >pss.485 5152834 104 - 6093698 ---AGACGCAGGUCUCGUGA-AU-GGGCACUCAACAAAAACAAGAAGCCCAGGCAGAAAACAAUAAUAA----AGAGCACACAC-------UUUGGGGGAGCUUCGGCUCCCCCAGUAGC ---.(((....)))((.((.-.(-((((..................)))))..))))............----..........(-------(((((((((((....)))))))))).)). ( -35.67) >pel.2717 5030115 106 + 5888780 ---AGCUUCGAGAUUUGUGG-ACAGGGCACUCAACAAAAACAAGAAGCCCAGCAGAAAAAUAAU----A----AGAGCACACAA--CGACUUGGUGGGGAGCUUCGGCUCCCCAUGUCGU ---.((((....((((...(-...((((..................))))..)....))))...----.----.)))).....(--((((...(((((((((....)))))))))))))) ( -36.17) >ppk.2718 5338538 106 + 6181863 ---AGCUUCGAGAUUUGUGG-ACAGGGCACUCAACAAAAACAAGAAGCCCAGCAAAAAAACAAU----A----AGAGCACGCAA--CGACUUCUUGGGGAGCUUAGGCUCCCCUUGUCGU ---............((((.-...((((..................)))).((...........----.----...)).))))(--((((.....(((((((....)))))))..))))) ( -30.53) >pfo.2853 5425485 106 + 6438405 ---AAGGAAAAGCUUCAUGGCGA-GGGCACUCAACAAAAACAAGAAGCCCG------AAUCAAUAAUAA----AAAGAGCACGCAACUACUUCUUGGGGAGCUUCGGCUCCCCUUGUAGU ---...((...(((....)))..-((((..................)))).------..))........----............(((((.....(((((((....)))))))..))))) ( -31.27) >pst.568 5350434 104 - 6397126 ---AGACAAAAGUCUCGUGA-AU-GGGCACUCAACAAAAACAAGAAGCCCGUACAGAAAAUAAAAAUAA----AGAGCACACAC-------UUUGGGGGAGCUUCGGCUCCCCCAGUAGC ---.(((....)))((.((.-((-((((..................)))))).))))............----..........(-------(((((((((((....)))))))))).)). ( -34.67) >consensus ___AGCCACAAGAUUCGUGG_AC_GGGCACUCAACAAAAACAAGAAGCCCAG__A_AAAACAAUAAUAA____AGAGCACACAC__C_ACUUCUGGGGGAGCUUCGGCUCCCCCAGUAGC ........................((((..................))))............................................((((((((....))))))))...... (-21.54 = -24.37 + 2.83)



| Location | 5,308,851 – 5,308,962 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 69.32 |
| Mean single sequence MFE | -32.91 |
| Consensus MFE | -21.33 |
| Energy contribution | -21.42 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.79 |
| SVM RNA-class probability | 0.977360 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2802 5308851 111 - 6264404 GCUACUGGGGGAGCCGAAGCUCCCCCCCAAGUAG--GUGCGUGCUUUGUUUUUAUUAUGAUUU------CGGGCUUGUUGUUUUUGUUUGUCGACCCGU-CCCUUGAGCCAGUCGCUUCA .(((((((((((((....))))))))...)))))--..(((.((((............(....------)((((..((((...........))))..))-))...))))....))).... ( -33.40) >pss.485 5152834 104 + 6093698 GCUACUGGGGGAGCCGAAGCUCCCCCAAA-------GUGUGUGCUCU----UUAUUAUUGUUUUCUGCCUGGGCUUCUUGUUUUUGUUGAGUGCCC-AU-UCACGAGACCUGCGUCU--- ((....((((((((....))))))))(((-------(.(....).))----))......(((((.((..(((((..((((.......)))).))))-).-.)).)))))..))....--- ( -35.00) >pel.2717 5030115 106 - 5888780 ACGACAUGGGGAGCCGAAGCUCCCCACCAAGUCG--UUGUGUGCUCU----U----AUUAUUUUUCUGCUGGGCUUCUUGUUUUUGUUGAGUGCCCUGU-CCACAAAUCUCGAAGCU--- .((((.((((((((....))))))))....))))--(((((.((...----.----...........)).((((..((((.......)))).))))...-.)))))...........--- ( -31.46) >ppk.2718 5338538 106 - 6181863 ACGACAAGGGGAGCCUAAGCUCCCCAAGAAGUCG--UUGCGUGCUCU----U----AUUGUUUUUUUGCUGGGCUUCUUGUUUUUGUUGAGUGCCCUGU-CCACAAAUCUCGAAGCU--- .(((((((((((((....)))))))((((((((.--..(((.((...----.----...)).....)))..))))))))....)))))).(((......-.))).............--- ( -32.20) >pfo.2853 5425485 106 - 6438405 ACUACAAGGGGAGCCGAAGCUCCCCAAGAAGUAGUUGCGUGCUCUUU----UUAUUAUUGAUU------CGGGCUUCUUGUUUUUGUUGAGUGCCC-UCGCCAUGAAGCUUUUCCUU--- .......(((((((....)))))))..((((.((((.(((((.....----(((....)))..------.((((..((((.......)))).))))-..).)))).))))))))...--- ( -29.70) >pst.568 5350434 104 + 6397126 GCUACUGGGGGAGCCGAAGCUCCCCCAAA-------GUGUGUGCUCU----UUAUUUUUAUUUUCUGUACGGGCUUCUUGUUUUUGUUGAGUGCCC-AU-UCACGAGACUUUUGUCU--- .....(((((((((....)))))))))..-------(((.((((...----...............))))((((..((((.......)))).))))-..-.))).((((....))))--- ( -35.67) >consensus ACUACAGGGGGAGCCGAAGCUCCCCAAAAAGU_G__GUGUGUGCUCU____UUAUUAUUAUUUU_U__CCGGGCUUCUUGUUUUUGUUGAGUGCCC_GU_CCACGAAACUUGUAGCU___ ......((((((((....))))))))............................................((((..((((.......)))).))))........................ (-21.33 = -21.42 + 0.08)
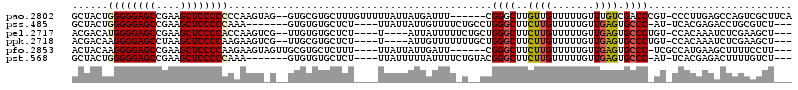

| Location | 5,308,860 – 5,308,971 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 70.32 |
| Mean single sequence MFE | -35.26 |
| Consensus MFE | -21.54 |
| Energy contribution | -24.37 |
| Covariance contribution | 2.83 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.75 |
| Structure conservation index | 0.61 |
| SVM decision value | 2.82 |
| SVM RNA-class probability | 0.997251 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2802 5308860 111 + 6264404 UGGCUCAAGGG-ACGGGUCGACAAACAAAAACAACAAGCCCG------AAAUCAUAAUAAAAACAAAGCACGCAC--CUACUUGGGGGGGAGCUUCGGCUCCCCCAGUAGCUUCACCCCC ........(((-.(((((...................)))))------.................((((((...(--(.....))((((((((....)))))))).)).))))..))).. ( -38.31) >pss.485 5152840 104 - 6093698 AGGUCUCGUGA-AU-GGGCACUCAACAAAAACAAGAAGCCCAGGCAGAAAACAAUAAUAA----AGAGCACACAC-------UUUGGGGGAGCUUCGGCUCCCCCAGUAGCUUCC-CC-- .((..((.((.-.(-((((..................)))))..))))............----.((((......-------.((((((((((....))))))))))..)))).)-).-- ( -35.57) >pel.2717 5030121 106 + 5888780 GAGAUUUGUGG-ACAGGGCACUCAACAAAAACAAGAAGCCCAGCAGAAAAAUAAU----A----AGAGCACACAA--CGACUUGGUGGGGAGCUUCGGCUCCCCAUGUCGUUUCU-CC-- ((((...(((.-...((((..................)))).((...........----.----...)).)))((--((((...(((((((((....))))))))))))))))))-).-- ( -39.53) >ppk.2718 5338544 106 + 6181863 GAGAUUUGUGG-ACAGGGCACUCAACAAAAACAAGAAGCCCAGCAAAAAAACAAU----A----AGAGCACGCAA--CGACUUCUUGGGGAGCUUAGGCUCCCCUUGUCGUUUCU-AC-- ((((.(((((.-...((((..................)))).((...........----.----...)).)))))--((((.....(((((((....)))))))..)))))))).-..-- ( -31.43) >pfo.2853 5425491 105 + 6438405 AAGCUUCAUGGCGA-GGGCACUCAACAAAAACAAGAAGCCCG------AAUCAAUAAUAA----AAAGAGCACGCAACUACUUCUUGGGGAGCUUCGGCUCCCCUUGUAGUUUCU-U--- ..(((....)))..-((((..................)))).------............----.........(.((((((.....(((((((....)))))))..)))))).).-.--- ( -32.67) >pst.568 5350440 103 - 6397126 AAGUCUCGUGA-AU-GGGCACUCAACAAAAACAAGAAGCCCGUACAGAAAAUAAAAAUAA----AGAGCACACAC-------UUUGGGGGAGCUUCGGCUCCCCCAGUAGCUUCC-A--- .....((.((.-((-((((..................)))))).))))............----.((((......-------.((((((((((....))))))))))..))))..-.--- ( -34.07) >consensus AAGAUUCGUGG_AC_GGGCACUCAACAAAAACAAGAAGCCCAG__A_AAAACAAUAAUAA____AGAGCACACAC__C_ACUUCUGGGGGAGCUUCGGCUCCCCCAGUAGCUUCU_CC__ ...............((((..................))))............................................((((((((....))))))))............... (-21.54 = -24.37 + 2.83)

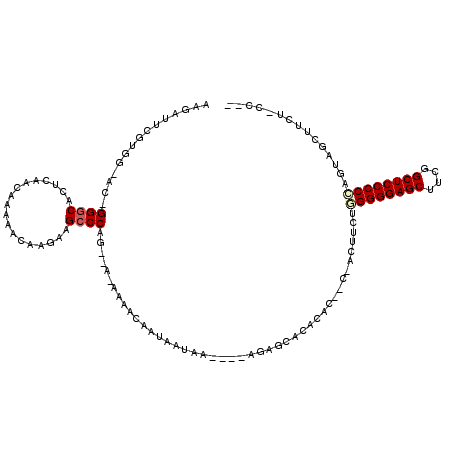
| Location | 5,308,860 – 5,308,971 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 70.32 |
| Mean single sequence MFE | -34.20 |
| Consensus MFE | -21.33 |
| Energy contribution | -21.42 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.966731 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2802 5308860 111 - 6264404 GGGGGUGAAGCUACUGGGGGAGCCGAAGCUCCCCCCCAAGUAG--GUGCGUGCUUUGUUUUUAUUAUGAUUU------CGGGCUUGUUGUUUUUGUUUGUCGACCCGU-CCCUUGAGCCA (((((..((((.((.((((((((....))))))))((.....)--)...))))))..)..............------((((.(((..............))))))))-)))........ ( -34.34) >pss.485 5152840 104 + 6093698 --GG-GGAAGCUACUGGGGGAGCCGAAGCUCCCCCAAA-------GUGUGUGCUCU----UUAUUAUUGUUUUCUGCCUGGGCUUCUUGUUUUUGUUGAGUGCCC-AU-UCACGAGACCU --((-(.(.(((..(((((((((....))))))))).)-------))...).))).----........(((((.((..(((((..((((.......)))).))))-).-.)).))))).. ( -37.60) >pel.2717 5030121 106 - 5888780 --GG-AGAAACGACAUGGGGAGCCGAAGCUCCCCACCAAGUCG--UUGUGUGCUCU----U----AUUAUUUUUCUGCUGGGCUUCUUGUUUUUGUUGAGUGCCCUGU-CCACAAAUCUC --.(-(((((((((.((((((((....))))))))....))))--))(((.((...----.----...........)).((((..((((.......)))).))))...-.)))...)))) ( -35.06) >ppk.2718 5338544 106 - 6181863 --GU-AGAAACGACAAGGGGAGCCUAAGCUCCCCAAGAAGUCG--UUGCGUGCUCU----U----AUUGUUUUUUUGCUGGGCUUCUUGUUUUUGUUGAGUGCCCUGU-CCACAAAUCUC --..-(((..(((((((((((((....)))))))((((((((.--..(((.((...----.----...)).....)))..))))))))....)))))).(((......-.)))...))). ( -32.50) >pfo.2853 5425491 105 - 6438405 ---A-AGAAACUACAAGGGGAGCCGAAGCUCCCCAAGAAGUAGUUGCGUGCUCUUU----UUAUUAUUGAUU------CGGGCUUCUUGUUUUUGUUGAGUGCCC-UCGCCAUGAAGCUU ---.-.(((.......(((((((....)))))))((((((.(((.....)))))))----))........))------)((((..((((.......)))).))))-..((......)).. ( -30.10) >pst.568 5350440 103 + 6397126 ---U-GGAAGCUACUGGGGGAGCCGAAGCUCCCCCAAA-------GUGUGUGCUCU----UUAUUUUUAUUUUCUGUACGGGCUUCUUGUUUUUGUUGAGUGCCC-AU-UCACGAGACUU ---.-(((((((..(((((((((....)))))))))..-------...(((((...----...............))))))))))))...((((((.((((....-))-))))))))... ( -35.57) >consensus __GG_AGAAACUACAGGGGGAGCCGAAGCUCCCCAAAAAGU_G__GUGUGUGCUCU____UUAUUAUUAUUUU_U__CCGGGCUUCUUGUUUUUGUUGAGUGCCC_GU_CCACGAAACUU ...............((((((((....))))))))............................................((((..((((.......)))).))))............... (-21.33 = -21.42 + 0.08)
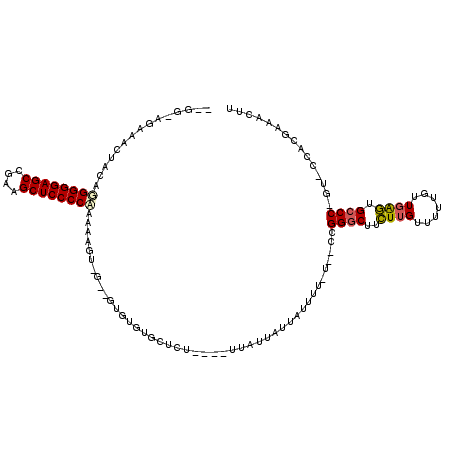
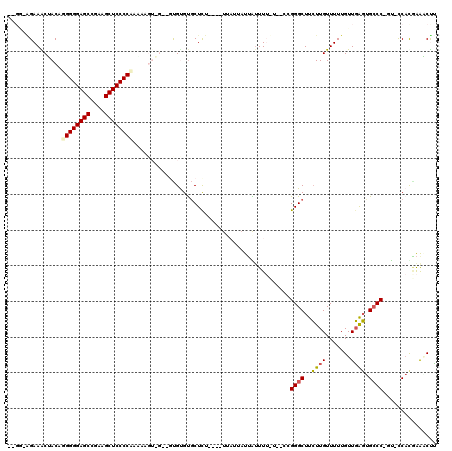
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:06:11 2007