
| Sequence ID | pao.2821 |
|---|---|
| Location | 5,344,903 – 5,345,048 |
| Length | 145 |
| Max. P | 0.971313 |

| Location | 5,344,903 – 5,345,009 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.99 |
| Mean single sequence MFE | -36.30 |
| Consensus MFE | -23.08 |
| Energy contribution | -23.80 |
| Covariance contribution | 0.72 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.31 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.764654 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2821 5344903 106 + 6264404 GAAGACCUCCCGUAAAUCAAGCCUGACUGGUCG--CGCAGG-UGUAAAAAAGCGGGAUGACACCGUGAAGAGUCAUCCCGCUUU--UUU-UCGAUUC--AUG--CUGCGCUGCUUU---- ...((((.....................))))(--((((((-((.((((((((((((((((.(......).)))))))))))))--)))-......)--)).--))))))......---- ( -41.00) >pst.2673 5073233 104 + 6397126 --AGACCUCCCGUAAAUAAAGCCUGAAAGGGCGAUCGCAGGUUGCAAAAAAGCGGAAUGACGUA--GUUACGUCACCCCGCUUU--CUUAUGGAAUC--AU---CUGCGC-GCUUU---- --........(((.......((((....))))....(((((((.((..(((((((..((((((.--...))))))..)))))))--....)).)...--))---))))))-)....---- ( -34.00) >psp.2444 4792700 108 + 5928787 --AGACCUCCCGUAAAUAAAACCUGAAAGGGCGAUCGCAGGCUGCAAAAAAGCGGAAUGACGUA--GUUACGUCACCCCGCUUU--CUUAUGGAAUC--AU---CUGCGC-GCUUUGCUU --.........(((((.....((.....))(((..((((((((..((.(((((((..((((((.--...))))))..)))))))--.))..))....--.)---))))))-))))))).. ( -32.50) >pf5.496 6115941 106 - 7074893 --AGACCUCCCGUAAAUAGAGCCUGAAAGGGCGAUCGCAGGUUGCAAAAAAGCGGAAUGACGUAUAGACACGUCACCCCGCUUC--UUCGUCGAAUU--AU---CUGCGC-GCUUU---- --........(((.......((((....))))....((((((.((.((.((((((..((((((......))))))..)))))).--)).))......--))---))))))-)....---- ( -33.80) >ppk.2736 5373169 113 + 6181863 --AGACCUCCCGUAAAUCAAGCAUGAAAGGGCGAUCGCAGGUUGCAAAAAAGCGGAGUGACGUAUGGACACGUCACCCCGCUUUCAUGUGCUGAAUUCAAUUCGCUGCGC-GCUUU---- --...(((..(((.........)))..)))(((..(((((...(((..(((((((.(((((((......))))))).)))))))....))).((((...)))).))))))-))...---- ( -38.00) >pel.392 5078600 107 - 5888780 --AGACCUCCCGUAAAUCAAGCCUGAAAGGGCGAUCGCAGGUUGCAAAAAAGCGGAAUGACGUAUGGACACGUCACCCCGCUUU--UAUGCUGAAUU-AGU---CUGCGC-GCUUU---- --........(((.......((((....))))....((((..((((.((((((((..((((((......))))))..)))))))--).)))......-)..---))))))-)....---- ( -38.50) >consensus __AGACCUCCCGUAAAUAAAGCCUGAAAGGGCGAUCGCAGGUUGCAAAAAAGCGGAAUGACGUAU_GACACGUCACCCCGCUUU__UUUGUCGAAUC__AU___CUGCGC_GCUUU____ ..........((((.....((((((...((....)).)))))).....(((((((..((((((......))))))..))))))).....................))))........... (-23.08 = -23.80 + 0.72)

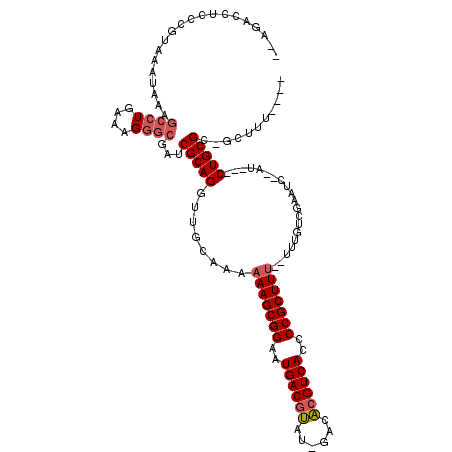
| Location | 5,344,903 – 5,345,009 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.99 |
| Mean single sequence MFE | -42.52 |
| Consensus MFE | -33.60 |
| Energy contribution | -32.99 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.24 |
| Mean z-score | -3.69 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.52 |
| SVM RNA-class probability | 0.961110 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2821 5344903 106 - 6264404 ----AAAGCAGCGCAG--CAU--GAAUCGA-AAA--AAAGCGGGAUGACUCUUCACGGUGUCAUCCCGCUUUUUUACA-CCUGCG--CGACCAGUCAGGCUUGAUUUACGGGAGGUCUUC ----.((((.((((((--..(--(......-(((--(((((((((((((.((....)).)))))))))))))))).))-.)))))--)((....))..))))(((((.....)))))... ( -40.50) >pst.2673 5073233 104 - 6397126 ----AAAGC-GCGCAG---AU--GAUUCCAUAAG--AAAGCGGGGUGACGUAAC--UACGUCAUUCCGCUUUUUUGCAACCUGCGAUCGCCCUUUCAGGCUUUAUUUACGGGAGGUCU-- ----...((-((((((---((--(....)))(((--(((((((((((((((...--.)))))))))))))))))).....)))))..)))......(((((((........)))))))-- ( -41.20) >psp.2444 4792700 108 - 5928787 AAGCAAAGC-GCGCAG---AU--GAUUCCAUAAG--AAAGCGGGGUGACGUAAC--UACGUCAUUCCGCUUUUUUGCAGCCUGCGAUCGCCCUUUCAGGUUUUAUUUACGGGAGGUCU-- ..((.....-))((((---((--(....)))(((--(((((((((((((((...--.)))))))))))))))))).....))))((((.(((.................))).)))).-- ( -40.93) >pf5.496 6115941 106 + 7074893 ----AAAGC-GCGCAG---AU--AAUUCGACGAA--GAAGCGGGGUGACGUGUCUAUACGUCAUUCCGCUUUUUUGCAACCUGCGAUCGCCCUUUCAGGCUCUAUUUACGGGAGGUCU-- ----...((-((((((---..--.....(.((((--((((((((((((((((....)))))))))))))))))))))...)))))..)))(((..(.............)..)))...-- ( -41.62) >ppk.2736 5373169 113 - 6181863 ----AAAGC-GCGCAGCGAAUUGAAUUCAGCACAUGAAAGCGGGGUGACGUGUCCAUACGUCACUCCGCUUUUUUGCAACCUGCGAUCGCCCUUUCAUGCUUGAUUUACGGGAGGUCU-- ----.....-..((((.((((...)))).(((...(((((((((((((((((....))))))))))))))))).)))...))))((((.(((..(((....))).....))).)))).-- ( -46.80) >pel.392 5078600 107 + 5888780 ----AAAGC-GCGCAG---ACU-AAUUCAGCAUA--AAAGCGGGGUGACGUGUCCAUACGUCAUUCCGCUUUUUUGCAACCUGCGAUCGCCCUUUCAGGCUUGAUUUACGGGAGGUCU-- ----.....-....((---(((-......(((.(--((((((((((((((((....))))))))))))))))).)))..((((.(((((((......)))..))))..)))).)))))-- ( -44.10) >consensus ____AAAGC_GCGCAG___AU__AAUUCAACAAA__AAAGCGGGGUGACGUGUC_AUACGUCAUUCCGCUUUUUUGCAACCUGCGAUCGCCCUUUCAGGCUUGAUUUACGGGAGGUCU__ .......((..(((((....................((((((((((((((((....))))))))))))))))........)))))...))((((((.............))))))..... (-33.60 = -32.99 + -0.61)

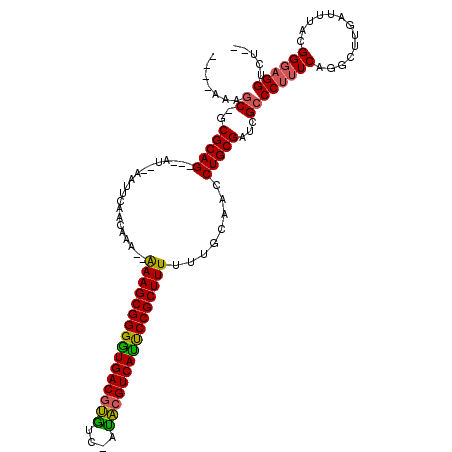
| Location | 5,344,941 – 5,345,048 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.08 |
| Mean single sequence MFE | -39.44 |
| Consensus MFE | -25.67 |
| Energy contribution | -25.82 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.83 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.64 |
| SVM RNA-class probability | 0.969569 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2821 5344941 107 + 6264404 G-UGUAAAAAAGCGGGAUGACACCGUGAAGAGUCAUCCCGCUUU--UUU-UCGAUUC--AUG--CUGCGCUGCUUU-----UAGUGGACACACUAAAGCUGUAGUUUACAGAAUCGCGAC .-...((((((((((((((((.(......).)))))))))))))--)))-.((((((--..(--(((((..(((((-----.((((....))))))))))))))).....)))))).... ( -44.40) >psp.2444 4792738 110 + 5928787 GCUGCAAAAAAGCGGAAUGACGUA--GUUACGUCACCCCGCUUU--CUUAUGGAAUC--AU---CUGCGC-GCUUUGCUUUUAGUGGACACACUAAAGCUUUAGCUUACAGAAAAGGCCC (((.....(((((((..((((((.--...))))))..)))))))--...........--.(---(((...-(((..(((((.((((....)))))))))...)))...))))...))).. ( -36.80) >pf5.496 6115979 107 - 7074893 GUUGCAAAAAAGCGGAAUGACGUAUAGACACGUCACCCCGCUUC--UUCGUCGAAUU--AU---CUGCGC-GCUUU-----UAGUGGACACACUAAAGCUGUAGCUUACAGAAAAAGGCU ...((....((((((..((((((......))))))..))))))(--((..((.....--..---(((((.-(((((-----.((((....))))))))))))))......))..))))). ( -35.12) >ppk.2736 5373207 114 + 6181863 GUUGCAAAAAAGCGGAGUGACGUAUGGACACGUCACCCCGCUUUCAUGUGCUGAAUUCAAUUCGCUGCGC-GCUUU-----UAGUGGACACACUAAAGCUGUAGCUUACAGAAAAGUGUG ...(((..(((((((.(((((((......))))))).)))))))..)))(((...(((.....((((((.-(((((-----.((((....))))))))))))))).....))).)))... ( -43.30) >pel.392 5078638 108 - 5888780 GUUGCAAAAAAGCGGAAUGACGUAUGGACACGUCACCCCGCUUU--UAUGCUGAAUU-AGU---CUGCGC-GCUUU-----UAGUGGACACACUAAAGCUGUAGCUUACAGAAAAAUGCG ...(((.((((((((..((((((......))))))..)))))))--)...(((....-((.---(((((.-(((((-----.((((....))))))))))))))))..))).....))). ( -39.70) >pfo.428 5539113 108 - 6438405 GUUGUAAAAAAGCGGAGUGACGUAUGGACACGUCACCCCGCUUC--UUCACUGAAUU--AUU--CUGCGC-GCUUU-----UAGUGGACACACUAAAGCUGUAGCUUACAGAAAAAGGGA .((((((..((((((.(((((((......))))))).)))))).--...........--...--(((((.-(((((-----.((((....)))))))))))))).))))))......... ( -37.30) >consensus GUUGCAAAAAAGCGGAAUGACGUAUGGACACGUCACCCCGCUUU__UUUGCUGAAUU__AU___CUGCGC_GCUUU_____UAGUGGACACACUAAAGCUGUAGCUUACAGAAAAAGGCC ((((((..(((((((..((((((......))))))..)))))))...........................((((......(((((....)))))))))))))))............... (-25.67 = -25.82 + 0.14)

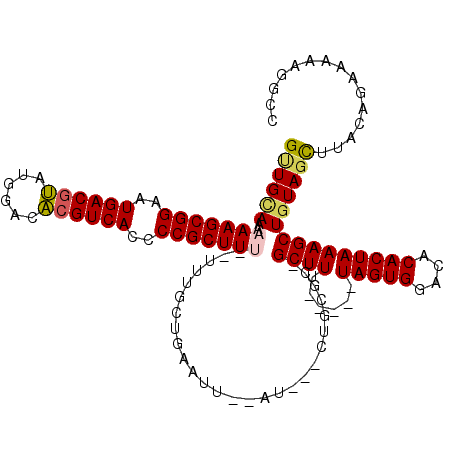
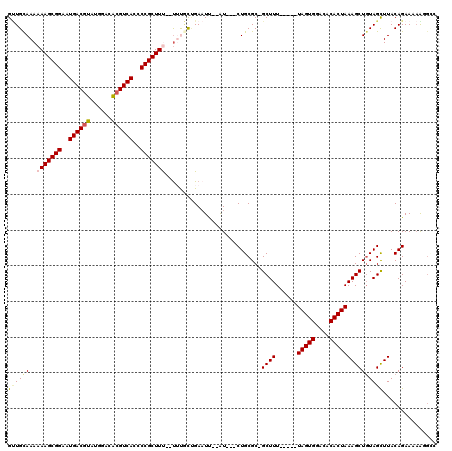
| Location | 5,344,941 – 5,345,048 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.08 |
| Mean single sequence MFE | -45.24 |
| Consensus MFE | -33.62 |
| Energy contribution | -32.35 |
| Covariance contribution | -1.27 |
| Combinations/Pair | 1.25 |
| Mean z-score | -5.73 |
| Structure conservation index | 0.74 |
| SVM decision value | 1.67 |
| SVM RNA-class probability | 0.971313 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.2821 5344941 107 - 6264404 GUCGCGAUUCUGUAAACUACAGCUUUAGUGUGUCCACUA-----AAAGCAGCGCAG--CAU--GAAUCGA-AAA--AAAGCGGGAUGACUCUUCACGGUGUCAUCCCGCUUUUUUACA-C ....(((((((((........(((((((((....)))).-----)))))......)--)).--)))))).-(((--(((((((((((((.((....)).))))))))))))))))...-. ( -42.04) >psp.2444 4792738 110 - 5928787 GGGCCUUUUCUGUAAGCUAAAGCUUUAGUGUGUCCACUAAAAGCAAAGC-GCGCAG---AU--GAUUCCAUAAG--AAAGCGGGGUGACGUAAC--UACGUCAUUCCGCUUUUUUGCAGC .((..((.(((((..(((...(((((((((....)))).)))))..)))-..))))---).--))..)).((((--(((((((((((((((...--.))))))))))))))))))).... ( -46.90) >pf5.496 6115979 107 + 7074893 AGCCUUUUUCUGUAAGCUACAGCUUUAGUGUGUCCACUA-----AAAGC-GCGCAG---AU--AAUUCGACGAA--GAAGCGGGGUGACGUGUCUAUACGUCAUUCCGCUUUUUUGCAAC ........(((((..(((.....(((((((....)))))-----)))))-..))))---).--.....(.((((--((((((((((((((((....)))))))))))))))))))))... ( -43.40) >ppk.2736 5373207 114 - 6181863 CACACUUUUCUGUAAGCUACAGCUUUAGUGUGUCCACUA-----AAAGC-GCGCAGCGAAUUGAAUUCAGCACAUGAAAGCGGGGUGACGUGUCCAUACGUCACUCCGCUUUUUUGCAAC ..((.(((.((((..(((.....(((((((....)))))-----)))))-..)))).))).))......(((...(((((((((((((((((....))))))))))))))))).)))... ( -45.30) >pel.392 5078638 108 + 5888780 CGCAUUUUUCUGUAAGCUACAGCUUUAGUGUGUCCACUA-----AAAGC-GCGCAG---ACU-AAUUCAGCAUA--AAAGCGGGGUGACGUGUCCAUACGUCAUUCCGCUUUUUUGCAAC .(.(((..(((((..(((.....(((((((....)))))-----)))))-..))))---)..-))).).(((.(--((((((((((((((((....))))))))))))))))).)))... ( -45.00) >pfo.428 5539113 108 + 6438405 UCCCUUUUUCUGUAAGCUACAGCUUUAGUGUGUCCACUA-----AAAGC-GCGCAG--AAU--AAUUCAGUGAA--GAAGCGGGGUGACGUGUCCAUACGUCACUCCGCUUUUUUACAAC .......((((((..(((.....(((((((....)))))-----)))))-..))))--)).--......(((((--((((((((((((((((....)))))))))))))))))))))... ( -48.80) >consensus CGCCCUUUUCUGUAAGCUACAGCUUUAGUGUGUCCACUA_____AAAGC_GCGCAG___AU__AAUUCAACAAA__AAAGCGGGGUGACGUGUCCAUACGUCAUUCCGCUUUUUUGCAAC ..........(((((((....(((((((((....)))))......))))...))......................((((((((((((((((....)))))))))))))))).))))).. (-33.62 = -32.35 + -1.27)

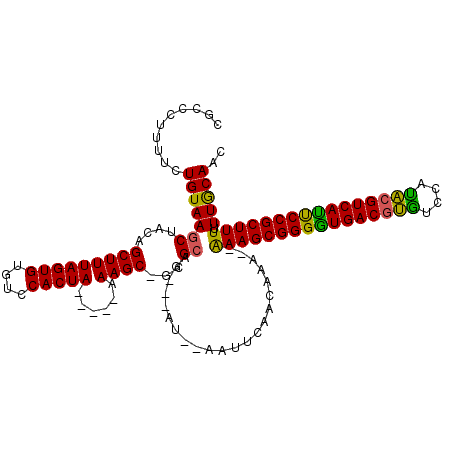
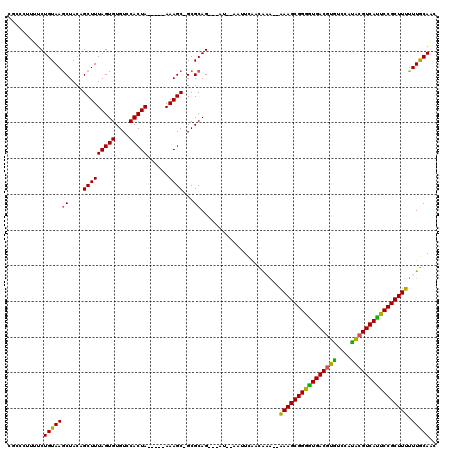
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:05:57 2007