
| Sequence ID | pao.1368 |
|---|---|
| Location | 2,549,458 – 2,549,563 |
| Length | 105 |
| Max. P | 0.996514 |

| Location | 2,549,458 – 2,549,561 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 68.08 |
| Mean single sequence MFE | -52.17 |
| Consensus MFE | -20.33 |
| Energy contribution | -20.80 |
| Covariance contribution | 0.47 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.78 |
| Structure conservation index | 0.39 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.707216 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
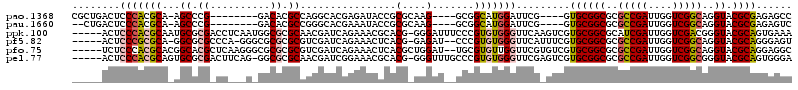
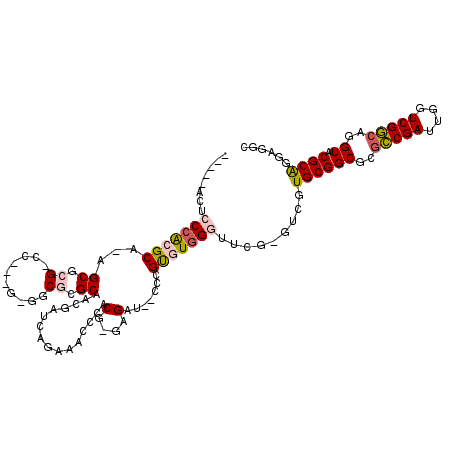
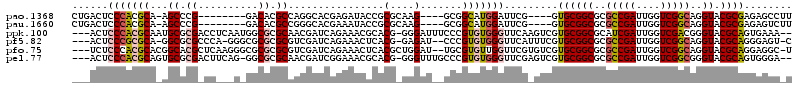
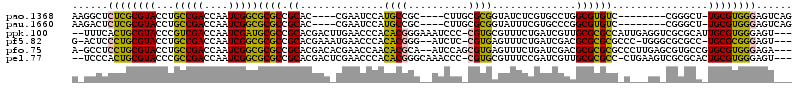
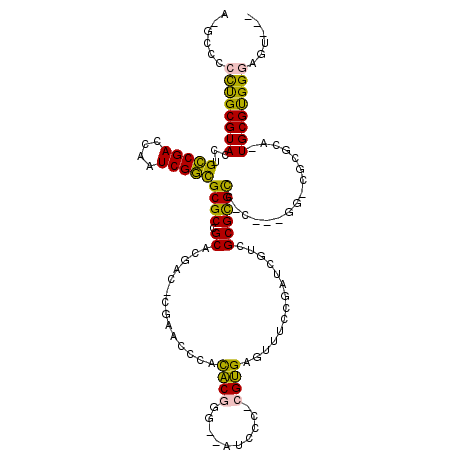
>pao.1368 2549458 103 + 6264404 CGCUGACUCCCACGCA-AGCCCG--------GACACGCCAGGCACGAGAUACCGCGCAAG----GCGGCAUGGAUUCG----GUGCGGCGCGCCGAUUGGUCGGCAGGUACGCGAGAGCC .(((((((.(((.((.-.(((.(--------(.....)).))).........(((.....----))))).)))..(((----((((...)))))))..))))))).(((.(....).))) ( -41.60) >pau.1660 3451278 101 - 6537648 --CUGACUCCCACGCA-AGCCCG--------GACACGCCGGGCACGAAAUACCGCGCAAG----GCGGCAUGGAUUCG----GUGCGGCGCGCCGAUUGGUCGGCAGGUACGCGAGAGUC --..(((((...(((.-.(((((--------(.....))))))......((((((((...----(((.(........)----.))).))))(((((....))))).)))).))).))))) ( -47.10) >ppk.100 5960641 114 - 6181863 -----ACUCCCACGCAAUGCGCGACCUCAAUGGCGCGCAACGAUCAGAAACGCACG-GGGAUUUCCCGUGUGGGUUCAAGUCGUGCGGCGCAUCGAUUGGUCGACGGGUACGCAGUGAAA -----.....(((((.((.(((((((......(((((((.((((..(((.((((((-((.....))))))))..)))..))))))).)))).......))))).)).))..)).)))... ( -49.12) >pf5.82 6914276 110 - 7074893 -----ACUCCCGCGCA-GGCGCGCCCA-GGGCGCGCGCGUCGAUCAGAAACUCACG-GAGAU--CCCGUGUGGGUUCAUUUCGUGCGGCGCGCCGAUUGGUCGGCAGGUACGCAGGGAGU -----((((((((((.-.(((((((..-.)))))))(((.(((...(((.(.((((-(....--.))))).)..)))...)))))).))))(((((....))))).........)))))) ( -57.80) >pfo.75 6272225 113 - 6438405 -----UCUCCCACGCACGGCACGCUCAAGGGCGCGCGCGUCGAUCAGAAACUCACGCUGGAU--UGCGUGUUGGUUCGUGUCGUGCGGCGCGCCGAUUGGUCGGCAGGUACGCAGGAGGC -----(((((..((.((.((.((..(((.((((((((((.((((..(((...(((((.....--.)))))....)))..))))))).)))))))..)))..))))..)).))..))))). ( -51.50) >pel.77 5733404 113 - 5888780 -----ACUCCCACGCAGUGCGCGACUUCAG-GGCGCGCAACGAUCGGAAACGCACG-GGGUUUGCCCGUGUGGGUUCGAGUCGUGCGGCGCGCCGAUUGGUCGGCGGGUACGCAGUGGGA -----..((((((((.((((((((((....-(((((((.(((((((((..((((((-((.....))))))))..))))).))))...)))))))....)))).))..)))))).)))))) ( -65.90) >consensus _____ACUCCCACGCA_AGCGCG_CC___G_GGCGCGCAACGAUCAGAAACCCACG_GAGAU__CCCGUGUGGGUUCG_GUCGUGCGGCGCGCCGAUUGGUCGGCAGGUACGCAGGAGGC ........(((((((...((.((..........)).))................(....).......))))))).........((((((..(((((....)))))..)).))))...... (-20.33 = -20.80 + 0.47)
| Location | 2,549,458 – 2,549,561 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 68.08 |
| Mean single sequence MFE | -52.02 |
| Consensus MFE | -25.88 |
| Energy contribution | -24.44 |
| Covariance contribution | -1.44 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.50 |
| SVM decision value | 2.60 |
| SVM RNA-class probability | 0.995617 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
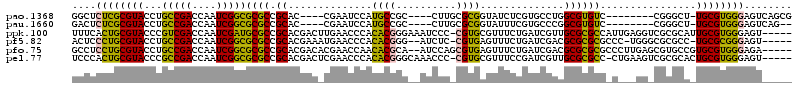
>pao.1368 2549458 103 - 6264404 GGCUCUCGCGUACCUGCCGACCAAUCGGCGCGCCGCAC----CGAAUCCAUGCCGC----CUUGCGCGGUAUCUCGUGCCUGGCGUGUC--------CGGGCU-UGCGUGGGAGUCAGCG ((((((((((((...(((((....))((((((((((((----.((....(((((((----.....))))))).))))))..))))))))--------..))).-)))))))))))).... ( -51.90) >pau.1660 3451278 101 + 6537648 GACUCUCGCGUACCUGCCGACCAAUCGGCGCGCCGCAC----CGAAUCCAUGCCGC----CUUGCGCGGUAUUUCGUGCCCGGCGUGUC--------CGGGCU-UGCGUGGGAGUCAG-- ((((((((((((...(((((....))((((((((((((----.(((...(((((((----.....))))))))))))))..))))))))--------..))).-))))))))))))..-- ( -53.90) >ppk.100 5960641 114 + 6181863 UUUCACUGCGUACCCGUCGACCAAUCGAUGCGCCGCACGACUUGAACCCACACGGGAAAUCCC-CGUGCGUUUCUGAUCGUUGCGCGCCAUUGAGGUCGCGCAUUGCGUGGGAGU----- .(((.(..((((..((.(((((..((((((.((((((((((..((((...((((((.....))-)))).))))..).))).)))).)))))))))))))))...))))..)))).----- ( -49.30) >pf5.82 6914276 110 + 7074893 ACUCCCUGCGUACCUGCCGACCAAUCGGCGCGCCGCACGAAAUGAACCCACACGGG--AUCUC-CGUGAGUUUCUGAUCGACGCGCGCGCCC-UGGGCGCGCC-UGCGCGGGAGU----- ((((((.(((((...(((((....)))))(((.((((.(((((.......(((((.--....)-)))).)))))))..)).)))(((((((.-..))))))).-)))))))))))----- ( -50.90) >pfo.75 6272225 113 + 6438405 GCCUCCUGCGUACCUGCCGACCAAUCGGCGCGCCGCACGACACGAACCAACACGCA--AUCCAGCGUGAGUUUCUGAUCGACGCGCGCGCCCUUGAGCGUGCCGUGCGUGGGAGA----- ..((((..(((((..((((..(((..(((((((((..(((((.((((...(((((.--.....))))).)))).)).))).)).))))))).)))..)).)).)))))..)))).----- ( -54.90) >pel.77 5733404 113 + 5888780 UCCCACUGCGUACCCGCCGACCAAUCGGCGCGCCGCACGACUCGAACCCACACGGGCAAACCC-CGUGCGUUUCCGAUCGUUGCGCGCC-CUGAAGUCGCGCACUGCGUGGGAGU----- ((((((.(((....(((.(((.....(((((((...((((.(((.(..(.((((((.....))-)))).)..).))))))).)))))))-.....))))))...)))))))))..----- ( -51.20) >consensus GCCCCCUGCGUACCUGCCGACCAAUCGGCGCGCCGCACGAC_CGAACCCACACGGG__AUCCC_CGUGAGUUUCCGAUCGUCGCGCGCC_C___GG_CGCGCA_UGCGUGGGAGU_____ ....((((((((...(((((....)))))((((.((..............((((..........))))..............))))))................))))))))........ (-25.88 = -24.44 + -1.44)
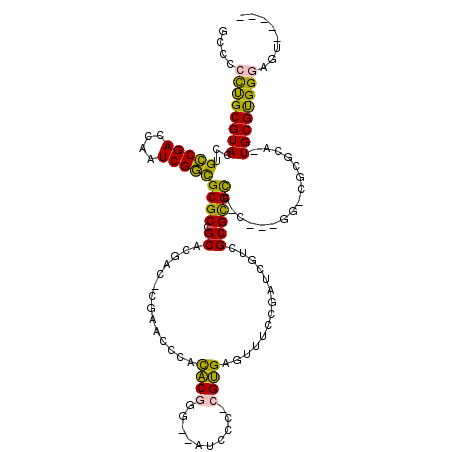

| Location | 2,549,460 – 2,549,563 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 67.80 |
| Mean single sequence MFE | -51.85 |
| Consensus MFE | -20.33 |
| Energy contribution | -20.80 |
| Covariance contribution | 0.47 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.39 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.625581 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1368 2549460 103 + 6264404 CUGACUCCCACGCA-AGCCCG--------GACACGCCAGGCACGAGAUACCGCGCAAG----GCGGCAUGGAUUCG----GUGCGGCGCGCCGAUUGGUCGGCAGGUACGCGAGAGCCUU ((((((.(((.((.-.(((.(--------(.....)).))).........(((.....----))))).)))..(((----((((...)))))))..)))))).((((.(....).)))). ( -38.40) >pau.1660 3451278 103 - 6537648 CUGACUCCCACGCA-AGCCCG--------GACACGCCGGGCACGAAAUACCGCGCAAG----GCGGCAUGGAUUCG----GUGCGGCGCGCCGAUUGGUCGGCAGGUACGCGAGAGUCUU ..(((((...(((.-.(((((--------(.....))))))......((((((((...----(((.(........)----.))).))))(((((....))))).)))).))).))))).. ( -48.30) >ppk.100 5960641 114 - 6181863 ---ACUCCCACGCAAUGCGCGACCUCAAUGGCGCGCAACGAUCAGAAACGCACG-GGGAUUUCCCGUGUGGGUUCAAGUCGUGCGGCGCAUCGAUUGGUCGACGGGUACGCAGUGAAA-- ---.....(((((.((.(((((((......(((((((.((((..(((.((((((-((.....))))))))..)))..))))))).)))).......))))).)).))..)).)))...-- ( -49.12) >pf5.82 6914276 111 - 7074893 ---ACUCCCGCGCA-GGCGCGCCCA-GGGCGCGCGCGUCGAUCAGAAACUCACG-GAGAU--CCCGUGUGGGUUCAUUUCGUGCGGCGCGCCGAUUGGUCGGCAGGUACGCAGGGAGU-C ---((((((((((.-.(((((((..-.)))))))(((.(((...(((.(.((((-(....--.))))).)..)))...)))))).))))(((((....))))).........))))))-. ( -57.90) >pfo.75 6272225 114 - 6438405 ---UCUCCCACGCACGGCACGCUCAAGGGCGCGCGCGUCGAUCAGAAACUCACGCUGGAU--UGCGUGUUGGUUCGUGUCGUGCGGCGCGCCGAUUGGUCGGCAGGUACGCAGGAGGC-U ---(((((..((.((.((.((..(((.((((((((((.((((..(((...(((((.....--.)))))....)))..))))))).)))))))..)))..))))..)).))..))))).-. ( -51.50) >pel.77 5733404 113 - 5888780 ---ACUCCCACGCAGUGCGCGACUUCAG-GGCGCGCAACGAUCGGAAACGCACG-GGGUUUGCCCGUGUGGGUUCGAGUCGUGCGGCGCGCCGAUUGGUCGGCGGGUACGCAGUGGGA-- ---..((((((((.((((((((((....-(((((((.(((((((((..((((((-((.....))))))))..))))).))))...)))))))....)))).))..)))))).))))))-- ( -65.90) >consensus ___ACUCCCACGCA_AGCGCG_CC___G_GGCGCGCAACGAUCAGAAACCCACG_GAGAU__CCCGUGUGGGUUCG_GUCGUGCGGCGCGCCGAUUGGUCGGCAGGUACGCAGGAGGC_U ......(((((((...((.((..........)).))................(....).......))))))).........((((((..(((((....)))))..)).))))........ (-20.33 = -20.80 + 0.47)
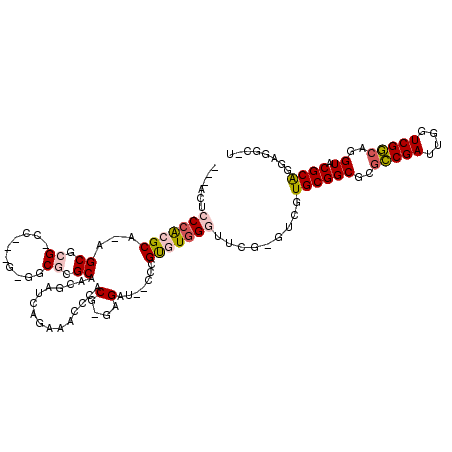
| Location | 2,549,460 – 2,549,563 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 67.80 |
| Mean single sequence MFE | -52.15 |
| Consensus MFE | -25.88 |
| Energy contribution | -24.44 |
| Covariance contribution | -1.44 |
| Combinations/Pair | 1.39 |
| Mean z-score | -2.65 |
| Structure conservation index | 0.50 |
| SVM decision value | 2.71 |
| SVM RNA-class probability | 0.996514 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1368 2549460 103 - 6264404 AAGGCUCUCGCGUACCUGCCGACCAAUCGGCGCGCCGCAC----CGAAUCCAUGCCGC----CUUGCGCGGUAUCUCGUGCCUGGCGUGUC--------CGGGCU-UGCGUGGGAGUCAG ..((((((((((((...(((((....))((((((((((((----.((....(((((((----.....))))))).))))))..))))))))--------..))).-)))))))))))).. ( -52.10) >pau.1660 3451278 103 + 6537648 AAGACUCUCGCGUACCUGCCGACCAAUCGGCGCGCCGCAC----CGAAUCCAUGCCGC----CUUGCGCGGUAUUUCGUGCCCGGCGUGUC--------CGGGCU-UGCGUGGGAGUCAG ..((((((((((((...(((((....))((((((((((((----.(((...(((((((----.....))))))))))))))..))))))))--------..))).-)))))))))))).. ( -54.10) >ppk.100 5960641 114 + 6181863 --UUUCACUGCGUACCCGUCGACCAAUCGAUGCGCCGCACGACUUGAACCCACACGGGAAAUCCC-CGUGCGUUUCUGAUCGUUGCGCGCCAUUGAGGUCGCGCAUUGCGUGGGAGU--- --.(((.(..((((..((.(((((..((((((.((((((((((..((((...((((((.....))-)))).))))..).))).)))).)))))))))))))))...))))..)))).--- ( -49.30) >pf5.82 6914276 111 + 7074893 G-ACUCCCUGCGUACCUGCCGACCAAUCGGCGCGCCGCACGAAAUGAACCCACACGGG--AUCUC-CGUGAGUUUCUGAUCGACGCGCGCGCCC-UGGGCGCGCC-UGCGCGGGAGU--- .-((((((.(((((...(((((....)))))(((.((((.(((((.......(((((.--....)-)))).)))))))..)).)))(((((((.-..))))))).-)))))))))))--- ( -51.30) >pfo.75 6272225 114 + 6438405 A-GCCUCCUGCGUACCUGCCGACCAAUCGGCGCGCCGCACGACACGAACCAACACGCA--AUCCAGCGUGAGUUUCUGAUCGACGCGCGCGCCCUUGAGCGUGCCGUGCGUGGGAGA--- .-..((((..(((((..((((..(((..(((((((((..(((((.((((...(((((.--.....))))).)))).)).))).)).))))))).)))..)).)).)))))..)))).--- ( -54.90) >pel.77 5733404 113 + 5888780 --UCCCACUGCGUACCCGCCGACCAAUCGGCGCGCCGCACGACUCGAACCCACACGGGCAAACCC-CGUGCGUUUCCGAUCGUUGCGCGCC-CUGAAGUCGCGCACUGCGUGGGAGU--- --((((((.(((....(((.(((.....(((((((...((((.(((.(..(.((((((.....))-)))).)..).))))))).)))))))-.....))))))...)))))))))..--- ( -51.20) >consensus A_GCCCCCUGCGUACCUGCCGACCAAUCGGCGCGCCGCACGAC_CGAACCCACACGGG__AUCCC_CGUGAGUUUCCGAUCGUCGCGCGCC_C___GG_CGCGCA_UGCGUGGGAGU___ ......((((((((...(((((....)))))((((.((..............((((..........))))..............))))))................))))))))...... (-25.88 = -24.44 + -1.44)
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:16:10 2007