
| Sequence ID | pao.3201 |
|---|---|
| Location | 6,043,825 – 6,044,664 |
| Length | 839 |
| Max. P | 0.999984 |

| Location | 6,043,825 – 6,043,945 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.39 |
| Mean single sequence MFE | -39.40 |
| Consensus MFE | -40.64 |
| Energy contribution | -39.58 |
| Covariance contribution | -1.05 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.23 |
| Structure conservation index | 1.03 |
| SVM decision value | 2.63 |
| SVM RNA-class probability | 0.995883 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
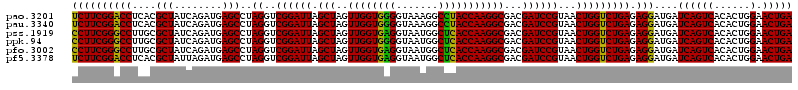
>pao.3201 6043825 120 - 6264404 CACGCCGUAAACGAUGUCGACUAGCCGUUGGGAUCCUUGAGAUCAUAGUGGCGCAGCUAACGCGAUAAGUCGACCGCCUGGGGAGUACGGCCGCAAGGUUAAAACUCAAAUGAAUUGACG ((.((((....))..(((((((.(((((((.((((.....)))).)))))))...((....))....))))))).)).))..((((..((((....))))...))))............. ( -36.90) >pau.3340 6316744 120 - 6537648 CACGCCGUAAACGAUGUCGACUAGCCGUUGGGAUCCUUGAGAUCUUAGUGGCGCAGCUAACGCGAUAAGUCGACCGCCUGGGGAGUACGGCCGCAAGGUUAAAACUCAAAUGAAUUGACG ((.((((....))..(((((((.((((((((((((.....))))))))))))...((....))....))))))).)).))..((((..((((....))))...))))............. ( -40.60) >ppk.94 6004143 120 + 6181863 CACGCCGUAAACGAUGUCAACUAGCCGUUGGAAUCCUUGAGAUUUUAGUGGCGCAGCUAACGCAUUAAGUUGACCGCCUGGGGAGUACGGCCGCAAGGUUAAAACUCAAAUGAAUUGACG ((.((((....))..(((((((.((((((((((((.....))))))))))))...((....))....))))))).)).))..((((..((((....))))...))))............. ( -36.50) >pst.2031 3873761 120 - 6397126 CACGCCGUAAACGAUGUCAACUAGCCGUUGGGAGCCUUGAGCUCUUAGUGGCGCAGCUAACGCAUUAAGUUGACCGCCUGGGGAGUACGGCCGCAAGGUUAAAACUCAAAUGAAUUGACG ((.((((....))..(((((((.((((((((((((.....))))))))))))...((....))....))))))).)).))..((((..((((....))))...))))............. ( -40.80) >pfo.3002 5730641 120 - 6438405 CACGCCGUAAACGAUGUCAACUAGCCGUUGGGAGCCUUGAGCUCUUAGUGGCGCAGCUAACGCAUUAAGUUGACCGCCUGGGGAGUACGGCCGCAAGGUUAAAACUCAAAUGAAUUGACG ((.((((....))..(((((((.((((((((((((.....))))))))))))...((....))....))))))).)).))..((((..((((....))))...))))............. ( -40.80) >pf5.3378 6387032 120 - 7074893 CACGCCGUAAACGAUGUCAACUAGCCGUUGGGAGCCUUGAGCUCUUAGUGGCGCAGCUAACGCAUUAAGUUGACCGCCUGGGGAGUACGGCCGCAAGGUUAAAACUCAAAUGAAUUGACG ((.((((....))..(((((((.((((((((((((.....))))))))))))...((....))....))))))).)).))..((((..((((....))))...))))............. ( -40.80) >consensus CACGCCGUAAACGAUGUCAACUAGCCGUUGGGAGCCUUGAGAUCUUAGUGGCGCAGCUAACGCAUUAAGUUGACCGCCUGGGGAGUACGGCCGCAAGGUUAAAACUCAAAUGAAUUGACG ((.((((....))..(((((((.((((((((((((.....))))))))))))...((....))....))))))).)).))..((((..((((....))))...))))............. (-40.64 = -39.58 + -1.05)

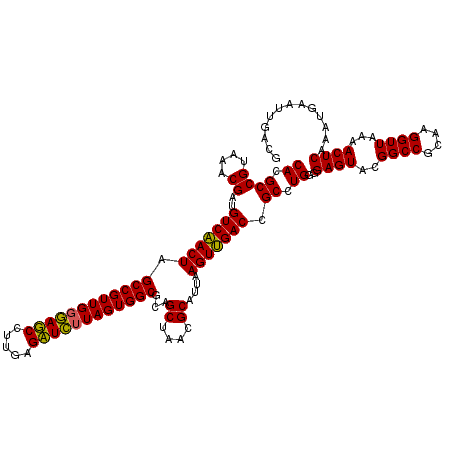
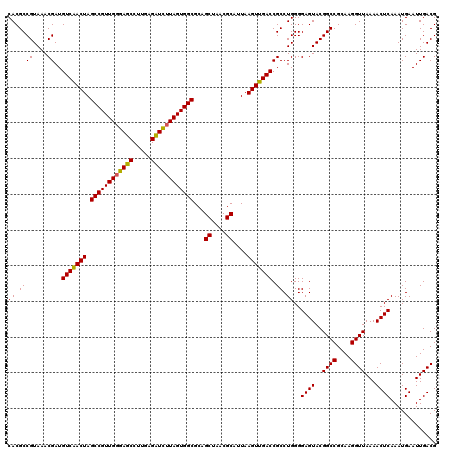
| Location | 6,043,865 – 6,043,985 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.39 |
| Mean single sequence MFE | -37.65 |
| Consensus MFE | -38.77 |
| Energy contribution | -37.55 |
| Covariance contribution | -1.22 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.17 |
| Structure conservation index | 1.03 |
| SVM decision value | 2.42 |
| SVM RNA-class probability | 0.993682 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043865 120 + 6264404 CAGGCGGUCGACUUAUCGCGUUAGCUGCGCCACUAUGAUCUCAAGGAUCCCAACGGCUAGUCGACAUCGUUUACGGCGUGGACUACCAGGGUAUCUAAUCCUGUUUGCUCCCCACGCUUU .((((((((((((..((((((.....))))......((((.....)))).....))..)))))))..)))))..(((((((.....((((((.....))))))........))))))).. ( -38.52) >pau.3340 6316784 120 + 6537648 CAGGCGGUCGACUUAUCGCGUUAGCUGCGCCACUAAGAUCUCAAGGAUCCCAACGGCUAGUCGACAUCGUUUACGGCGUGGACUACCAGGGUAUCUAAUCCUGUUUGCUCCCCACGCUUU .((((((((((((..((((((.....))))......((((.....)))).....))..)))))))..)))))..(((((((.....((((((.....))))))........))))))).. ( -38.72) >ppk.94 6004183 120 - 6181863 CAGGCGGUCAACUUAAUGCGUUAGCUGCGCCACUAAAAUCUCAAGGAUUCCAACGGCUAGUUGACAUCGUUUACGGCGUGGACUACCAGGGUAUCUAAUCCUGUUUGCUCCCCACGCUUU .((((((((((((....((....))...(((.....((((.....)))).....))).)))))))..)))))..(((((((.....((((((.....))))))........))))))).. ( -34.02) >pst.2031 3873801 120 + 6397126 CAGGCGGUCAACUUAAUGCGUUAGCUGCGCCACUAAGAGCUCAAGGCUCCCAACGGCUAGUUGACAUCGUUUACGGCGUGGACUACCAGGGUAUCUAAUCCUGUUUGCUCCCCACGCUUU .((((((((((((....((....))...(((.....((((.....)))).....))).)))))))..)))))..(((((((.....((((((.....))))))........))))))).. ( -38.22) >pfo.3002 5730681 120 + 6438405 CAGGCGGUCAACUUAAUGCGUUAGCUGCGCCACUAAGAGCUCAAGGCUCCCAACGGCUAGUUGACAUCGUUUACGGCGUGGACUACCAGGGUAUCUAAUCCUGUUUGCUCCCCACGCUUU .((((((((((((....((....))...(((.....((((.....)))).....))).)))))))..)))))..(((((((.....((((((.....))))))........))))))).. ( -38.22) >pf5.3378 6387072 120 + 7074893 CAGGCGGUCAACUUAAUGCGUUAGCUGCGCCACUAAGAGCUCAAGGCUCCCAACGGCUAGUUGACAUCGUUUACGGCGUGGACUACCAGGGUAUCUAAUCCUGUUUGCUCCCCACGCUUU .((((((((((((....((....))...(((.....((((.....)))).....))).)))))))..)))))..(((((((.....((((((.....))))))........))))))).. ( -38.22) >consensus CAGGCGGUCAACUUAAUGCGUUAGCUGCGCCACUAAGAGCUCAAGGAUCCCAACGGCUAGUUGACAUCGUUUACGGCGUGGACUACCAGGGUAUCUAAUCCUGUUUGCUCCCCACGCUUU .((((((((((((....((....))...(((.....((((.....)))).....))).)))))))..)))))..(((((((.....((((((.....))))))........))))))).. (-38.77 = -37.55 + -1.22)

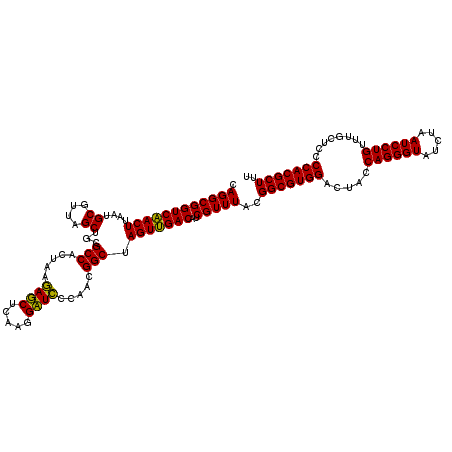
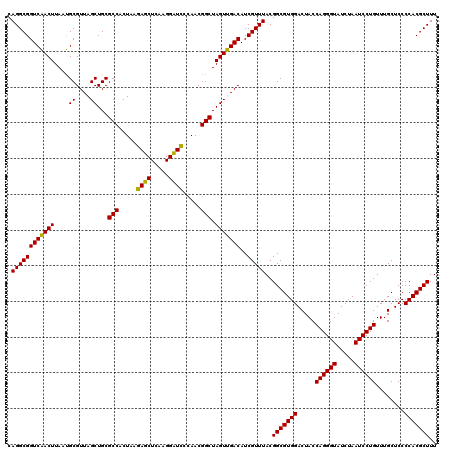
| Location | 6,043,865 – 6,043,985 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.39 |
| Mean single sequence MFE | -42.30 |
| Consensus MFE | -43.54 |
| Energy contribution | -42.48 |
| Covariance contribution | -1.05 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.72 |
| Structure conservation index | 1.03 |
| SVM decision value | 4.05 |
| SVM RNA-class probability | 0.999776 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043865 120 - 6264404 AAAGCGUGGGGAGCAAACAGGAUUAGAUACCCUGGUAGUCCACGCCGUAAACGAUGUCGACUAGCCGUUGGGAUCCUUGAGAUCAUAGUGGCGCAGCUAACGCGAUAAGUCGACCGCCUG ...(((((((..((...((((.........))))))..)))))))((....))..(((((((.(((((((.((((.....)))).)))))))...((....))....)))))))...... ( -39.80) >pau.3340 6316784 120 - 6537648 AAAGCGUGGGGAGCAAACAGGAUUAGAUACCCUGGUAGUCCACGCCGUAAACGAUGUCGACUAGCCGUUGGGAUCCUUGAGAUCUUAGUGGCGCAGCUAACGCGAUAAGUCGACCGCCUG ...(((((((..((...((((.........))))))..)))))))((....))..(((((((.((((((((((((.....))))))))))))...((....))....)))))))...... ( -43.50) >ppk.94 6004183 120 + 6181863 AAAGCGUGGGGAGCAAACAGGAUUAGAUACCCUGGUAGUCCACGCCGUAAACGAUGUCAACUAGCCGUUGGAAUCCUUGAGAUUUUAGUGGCGCAGCUAACGCAUUAAGUUGACCGCCUG ...(((((((..((...((((.........))))))..)))))))((....))..(((((((.((((((((((((.....))))))))))))...((....))....)))))))...... ( -39.40) >pst.2031 3873801 120 - 6397126 AAAGCGUGGGGAGCAAACAGGAUUAGAUACCCUGGUAGUCCACGCCGUAAACGAUGUCAACUAGCCGUUGGGAGCCUUGAGCUCUUAGUGGCGCAGCUAACGCAUUAAGUUGACCGCCUG ...(((((((..((...((((.........))))))..)))))))((....))..(((((((.((((((((((((.....))))))))))))...((....))....)))))))...... ( -43.70) >pfo.3002 5730681 120 - 6438405 AAAGCGUGGGGAGCAAACAGGAUUAGAUACCCUGGUAGUCCACGCCGUAAACGAUGUCAACUAGCCGUUGGGAGCCUUGAGCUCUUAGUGGCGCAGCUAACGCAUUAAGUUGACCGCCUG ...(((((((..((...((((.........))))))..)))))))((....))..(((((((.((((((((((((.....))))))))))))...((....))....)))))))...... ( -43.70) >pf5.3378 6387072 120 - 7074893 AAAGCGUGGGGAGCAAACAGGAUUAGAUACCCUGGUAGUCCACGCCGUAAACGAUGUCAACUAGCCGUUGGGAGCCUUGAGCUCUUAGUGGCGCAGCUAACGCAUUAAGUUGACCGCCUG ...(((((((..((...((((.........))))))..)))))))((....))..(((((((.((((((((((((.....))))))))))))...((....))....)))))))...... ( -43.70) >consensus AAAGCGUGGGGAGCAAACAGGAUUAGAUACCCUGGUAGUCCACGCCGUAAACGAUGUCAACUAGCCGUUGGGAGCCUUGAGAUCUUAGUGGCGCAGCUAACGCAUUAAGUUGACCGCCUG ...(((((((..((...((((.........))))))..)))))))((....))..(((((((.((((((((((((.....))))))))))))...((....))....)))))))...... (-43.54 = -42.48 + -1.05)
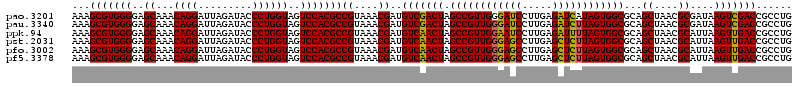
| Location | 6,043,985 – 6,044,105 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.11 |
| Mean single sequence MFE | -42.30 |
| Consensus MFE | -41.00 |
| Energy contribution | -40.75 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.88 |
| Structure conservation index | 0.97 |
| SVM decision value | 4.93 |
| SVM RNA-class probability | 0.999963 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043985 120 - 6264404 CUGAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGGCGAAGGCGACCACCUGGACUGAUACUGACACUGAGGUGCG ((.((....((((((((..(((.(((((...((((((((((.(((......)))....))))))))))...)))))((.((....)).))..)))..)))).))))....)).))..... ( -42.50) >pau.3340 6316904 120 - 6537648 CUGAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGGCGAAGGCGACCACCUGGACUGAUACUGACACUGAGGUGCG ((.((....((((((((..(((.(((((...((((((((((.(((......)))....))))))))))...)))))((.((....)).))..)))..)))).))))....)).))..... ( -42.50) >ppk.94 6004303 120 + 6181863 GCAAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGGCGAAGGCGACCACCUGGACUGAUACUGACACUGAGGUGCG (((...(((((((((((..(((.(((((...((((((((((.(((......)))....))))))))))...)))))((.((....)).))..)))..)))).))))....)))...))). ( -43.50) >pst.2031 3873921 120 - 6397126 GCAAGCUAGAGUAUGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGGCGAAGGCGACCACCUGGACUGAUACUGACACUGAGGUGCG (((...(((((((((((..(((.(((((...((((((((((.(((......)))....))))))))))...)))))((.((....)).))..)))..))).)))))....)))...))). ( -42.00) >pfo.3002 5730801 120 - 6438405 GCGAGCUAGAGUAUGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGGCGAAGGCGACCACCUGGACUGAUACUGACACUGAGGUGCG (((...(((((((((((..(((.(((((...((((((((((.(((......)))....))))))))))...)))))((.((....)).))..)))..))).)))))....)))...))). ( -41.30) >pf5.3378 6387192 120 - 7074893 GCAAGCUAGAGUAUGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGGCGAAGGCGACCACCUGGACUGAUACUGACACUGAGGUGCG (((...(((((((((((..(((.(((((...((((((((((.(((......)))....))))))))))...)))))((.((....)).))..)))..))).)))))....)))...))). ( -42.00) >consensus GCAAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGGCGAAGGCGACCACCUGGACUGAUACUGACACUGAGGUGCG .........((((((((..(((.(((((...((((((((((.(((......)))....))))))))))...)))))((.((....)).))..)))..)))).))))(.(((....)))). (-41.00 = -40.75 + -0.25)

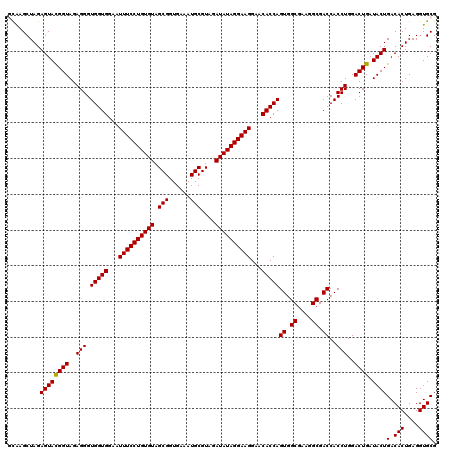
| Location | 6,044,025 – 6,044,145 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.22 |
| Mean single sequence MFE | -41.33 |
| Consensus MFE | -39.71 |
| Energy contribution | -39.10 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.57 |
| Structure conservation index | 0.96 |
| SVM decision value | 4.48 |
| SVM RNA-class probability | 0.999906 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044025 120 - 6264404 GAAAUCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUACUGAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGG ......((((((.....))))))..(..(((((....((((((.((....)).)))))).))))((((...((((((((((.(((......)))....))))))))))...)))).)..) ( -43.90) >pau.3340 6316944 120 - 6537648 GAAAUCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUACUGAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGG ......((((((.....))))))..(..(((((....((((((.((....)).)))))).))))((((...((((((((((.(((......)))....))))))))))...)))).)..) ( -43.90) >ppk.94 6004343 120 + 6181863 GAAAGCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUGGCAAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGG ......((((((.....))))))..(..(((((...((((((........)).))))...))))((((...((((((((((.(((......)))....))))))))))...)))).)..) ( -39.70) >pst.2031 3873961 120 - 6397126 GAAAUCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUGGCAAGCUAGAGUAUGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGG ......((((((.....))))))..(..(((((....(((.((.((....)).)).))).))))((((...((((((((((.(((......)))....))))))))))...)))).)..) ( -40.40) >pfo.3002 5730841 120 - 6438405 GAAAUCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUGGCGAGCUAGAGUAUGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGG ......((((((.....))))))..(..(((((....(((.((.((....)).)).))).))))((((...((((((((((.(((......)))....))))))))))...)))).)..) ( -39.70) >pf5.3378 6387232 120 - 7074893 GAAAGCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUGGCAAGCUAGAGUAUGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGG ......((((((.....))))))..(..(((((....(((.((.((....)).)).))).))))((((...((((((((((.(((......)))....))))))))))...)))).)..) ( -40.40) >consensus GAAAUCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUGGCAAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUGUAGCGGUGAAAUGCGUAGAUAUAGGAAGGAACACCAGUGG ......((((((.....))))))..(..(((((....(((.((.((....)).)).))).))))((((...((((((((((.(((......)))....))))))))))...)))).)..) (-39.71 = -39.10 + -0.61)

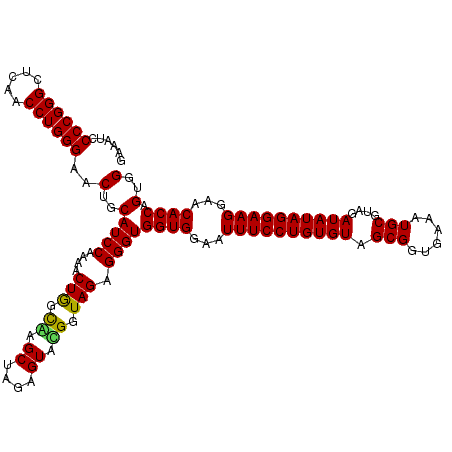
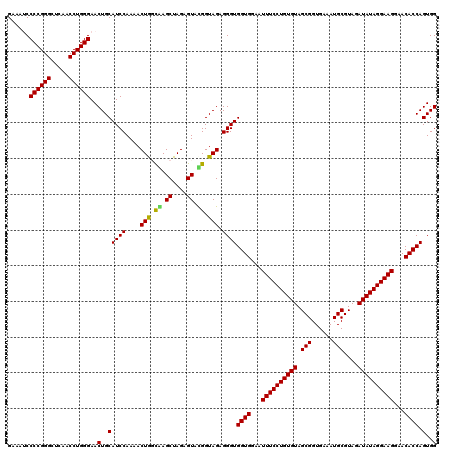
| Location | 6,044,065 – 6,044,185 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.11 |
| Mean single sequence MFE | -39.75 |
| Consensus MFE | -39.55 |
| Energy contribution | -38.42 |
| Covariance contribution | -1.14 |
| Combinations/Pair | 1.11 |
| Mean z-score | -0.97 |
| Structure conservation index | 1.00 |
| SVM decision value | 1.22 |
| SVM RNA-class probability | 0.932211 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044065 120 - 6264404 GGGCGUAAAGCGCGCGUAGGUGGUUCAGCAAGUUGGAUGUGAAAUCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUACUGAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUG (.(((.....))).).((((..((((.((...(((((((((...((((.((......)).))))..)))))))))..((((((.((....)).))))))......)).))))..)))).. ( -43.60) >pau.3340 6316984 120 - 6537648 GGGCGUAAAGCGCGCGUAGGUGGUUCAGCAAGUUGGAUGUGAAAUCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUACUGAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUG (.(((.....))).).((((..((((.((...(((((((((...((((.((......)).))))..)))))))))..((((((.((....)).))))))......)).))))..)))).. ( -43.60) >ppk.94 6004383 120 + 6181863 GGGCGUAAAGCGCGCGUAGGUGGUUUGUUAAGUUGGAUGUGAAAGCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUGGCAAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUG ..(((.....))).((((..((((((((((..((((((((......((((((.....))))))....))))))))...))))))))))...)))).((.((((..........)))).)) ( -40.10) >pst.2031 3874001 120 - 6397126 GGGCGUAAAGCGCGCGUAGGUGGUUUGUUAAGUUGAAUGUGAAAUCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUGGCAAGCUAGAGUAUGGUAGAGGGUGGUGGAAUUUCCUGUG (.(((.....))).).....((((((((((..(((.(((((...((((.((......)).))))..))))).)))...))))))))))........((.((((..........)))).)) ( -32.00) >pfo.3002 5730881 120 - 6438405 GGGCGUAAAGCGCGCGUAGGUGGUUCGUUAAGUUGGAUGUGAAAUCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUGGCGAGCUAGAGUAUGGUAGAGGGUGGUGGAAUUUCCUGUG (.(((.....))).).....((((((((((..(((((((((...((((.((......)).))))..)))))))))...))))))))))........((.((((..........)))).)) ( -40.60) >pf5.3378 6387272 120 - 7074893 GGGCGUAAAGCGCGCGUAGGUGGUUUGUUAAGUUGGAUGUGAAAGCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUGGCAAGCUAGAGUAUGGUAGAGGGUGGUGGAAUUUCCUGUG (.(((.....))).).....((((((((((..((((((((......((((((.....))))))....))))))))...))))))))))........((.((((..........)))).)) ( -38.60) >consensus GGGCGUAAAGCGCGCGUAGGUGGUUCGUUAAGUUGGAUGUGAAAUCCCCGGGCUCAACCUGGGAACUGCAUCCAAAACUGGCAAGCUAGAGUACGGUAGAGGGUGGUGGAAUUUCCUGUG ..(((.....))).((((..((((((((((..((((((((......((((((.....))))))....))))))))...))))))))))...)))).((.((((..........)))).)) (-39.55 = -38.42 + -1.14)
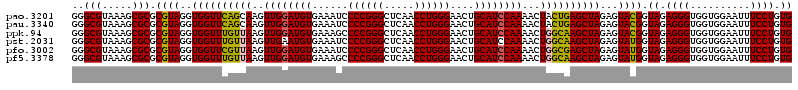
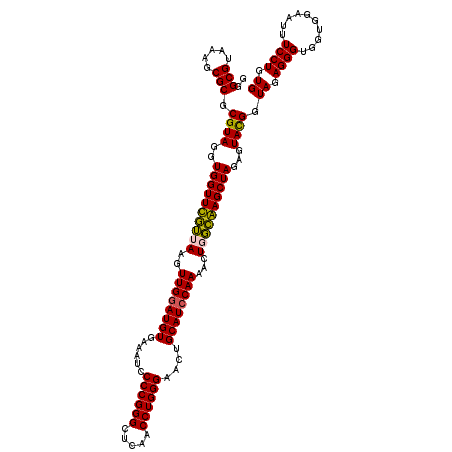
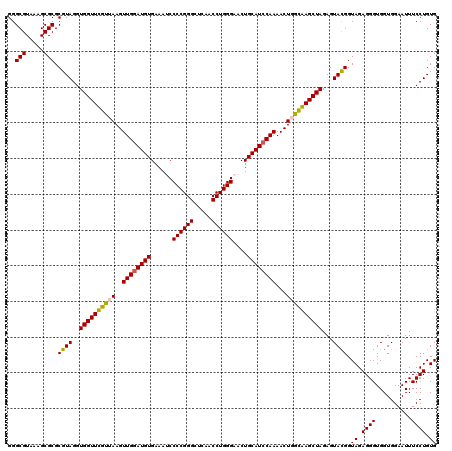
| Location | 6,044,145 – 6,044,265 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.50 |
| Mean single sequence MFE | -38.97 |
| Consensus MFE | -37.33 |
| Energy contribution | -36.30 |
| Covariance contribution | -1.03 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.11 |
| Structure conservation index | 0.96 |
| SVM decision value | 1.08 |
| SVM RNA-class probability | 0.911543 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044145 120 - 6264404 UACCAACAGAAUAAGCACCGGCUAACUUCGUGCCAGCAGCCGCGGUAAUACGAAGGGUGCAAGCGUUAAUCGGAAUUACUGGGCGUAAAGCGCGCGUAGGUGGUUCAGCAAGUUGGAUGU ..(((((.......(((((......(((((((((.((....)))))...)))))))))))..(((.....).(((((((((.((((.....)))).)).))))))).))..))))).... ( -39.30) >pau.3340 6317064 120 - 6537648 UACCAACAGAAUAAGCACCGGCUAACUUCGUGCCAGCAGCCGCGGUAAUACGAAGGGUGCAAGCGUUAAUCGGAAUUACUGGGCGUAAAGCGCGCGUAGGUGGUUCAGCAAGUUGGAUGU ..(((((.......(((((......(((((((((.((....)))))...)))))))))))..(((.....).(((((((((.((((.....)))).)).))))))).))..))))).... ( -39.30) >pss.1919 3852077 120 - 6093698 UACCGACAGAAUAAGCACCGGCUAACUCUGUGCCAGCAGCCGCGGUAAUACAGAGGGUGCAAGCGUUAAUCGGAAUUACUGGGCGUAAAGCGCGCGUAGGUGGUUUGUUAAGUUGAAUGU ...((((..((((((((((.((...(((((((((.((....)))))...))))))...))..((((...((((.....))))(((.....))))))).))).)))))))..))))..... ( -37.40) >pst.2031 3874081 120 - 6397126 UACCGACAGAAUAAGCACCGGCUAACUCUGUGCCAGCAGCCGCGGUAAUACAGAGGGUGCAAGCGUUAAUCGGAAUUACUGGGCGUAAAGCGCGCGUAGGUGGUUUGUUAAGUUGAAUGU ...((((..((((((((((.((...(((((((((.((....)))))...))))))...))..((((...((((.....))))(((.....))))))).))).)))))))..))))..... ( -37.40) >pfo.3002 5730961 120 - 6438405 UACCGACAGAAUAAGCACCGGCUAACUCUGUGCCAGCAGCCGCGGUAAUACAGAGGGUGCAAGCGUUAAUCGGAAUUACUGGGCGUAAAGCGCGCGUAGGUGGUUCGUUAAGUUGGAUGU ..(((((.......(((((......(((((((((.((....)))))...))))))))))).....((((.((((....(((.((((.....)))).)))....))))))))))))).... ( -39.50) >pf5.3378 6387352 120 - 7074893 UACCGACAGAAUAAGCACCGGCUAACUCUGUGCCAGCAGCCGCGGUAAUACAGAGGGUGCAAGCGUUAAUCGGAAUUACUGGGCGUAAAGCGCGCGUAGGUGGUUUGUUAAGUUGGAUGU ..(((((..((((((((((.((...(((((((((.((....)))))...))))))...))..((((...((((.....))))(((.....))))))).))).)))))))..))))).... ( -40.90) >consensus UACCGACAGAAUAAGCACCGGCUAACUCUGUGCCAGCAGCCGCGGUAAUACAGAGGGUGCAAGCGUUAAUCGGAAUUACUGGGCGUAAAGCGCGCGUAGGUGGUUCGUUAAGUUGGAUGU ..(((((.......(((((......(((((((((.((....)))))...)))))))))))............(((((((((.((((.....)))).)).))))))).....))))).... (-37.33 = -36.30 + -1.03)

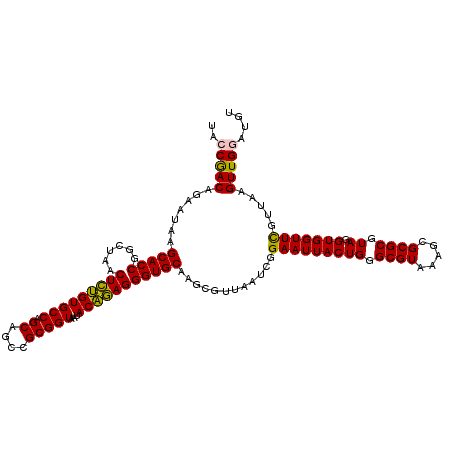
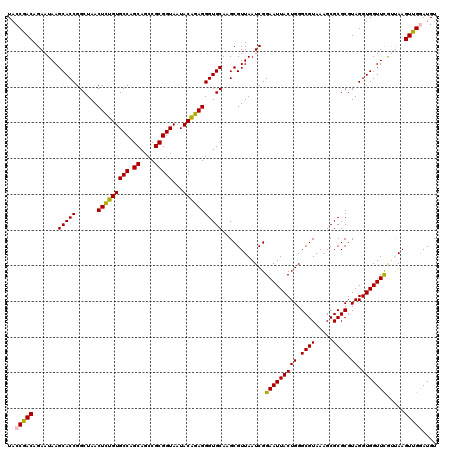
| Location | 6,044,225 – 6,044,345 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.61 |
| Mean single sequence MFE | -34.63 |
| Consensus MFE | -35.45 |
| Energy contribution | -33.57 |
| Covariance contribution | -1.88 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.00 |
| Structure conservation index | 1.02 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.963839 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044225 120 - 6264404 GUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUAAGUUAAUACCUUGCUGUUUUGACGUUACCAACAGAAUAAGCACCGGCUAACUUCGUGCCAGCAGC ((((......((((....)))).....)))).....(((((.(.(((((((((((((.......)))))))))....................(((....)))..)))).).)))))... ( -34.80) >pau.3340 6317144 120 - 6537648 GUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUAAGUUAAUACCUUGCUGUUUUGACGUUACCAACAGAAUAAGCACCGGCUAACUUCGUGCCAGCAGC ((((......((((....)))).....)))).....(((((.(.(((((((((((((.......)))))))))....................(((....)))..)))).).)))))... ( -34.80) >ppk.94 6004543 120 + 6181863 GUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUAAGUUAAUACCUUGCUGUUUUGACGUUACCGACAGAAUAAGCACCGGCUAACUCUGUGCCAGCAGC ((((......((((....)))).....)))).....(((((..(..(((((((((((.......)))))))))))..).......(((((.(.(((....))).).))))).)))))... ( -36.50) >psp.1833 3636173 120 - 5928787 GUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUUACCUAAUACGUGAUUGUUUUGACGUUACCGACAGAAUAAGCACCGGCUAACUCUGUGCCAGCAGC ((((......((((....)))).....)))).....(((((..(..(((((((((((.......)))))))))))..).......(((((.(.(((....))).).))))).)))))... ( -34.30) >pst.2031 3874161 120 - 6397126 GUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUUACCUAAUACGUAUCUGUUUUGACGUUACCGACAGAAUAAGCACCGGCUAACUCUGUGCCAGCAGC ((((......((((....)))).....)))).....(((((..(..(((((((.(((.......))).)))))))..).......(((((.(.(((....))).).))))).)))))... ( -32.40) >pfo.3002 5731041 120 - 6438405 GUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGUUGUAGAUUAAUACUCUGCAAUUUUGACGUUACCGACAGAAUAAGCACCGGCUAACUCUGUGCCAGCAGC ((((......((((....)))).....)))).....(((((..(..(((((((((((.......)))))))))))..).......(((((.(.(((....))).).))))).)))))... ( -35.00) >consensus GUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUAAGUUAAUACCUUGCUGUUUUGACGUUACCGACAGAAUAAGCACCGGCUAACUCUGUGCCAGCAGC ((((......((((....)))).....)))).....(((((..(..(((((((((((.......)))))))))))..).......(((((.(.(((....))).).))))).)))))... (-35.45 = -33.57 + -1.88)

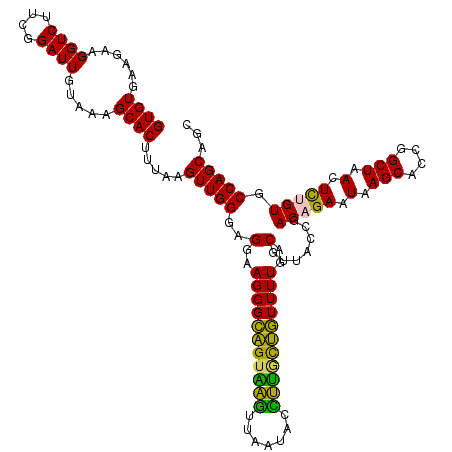
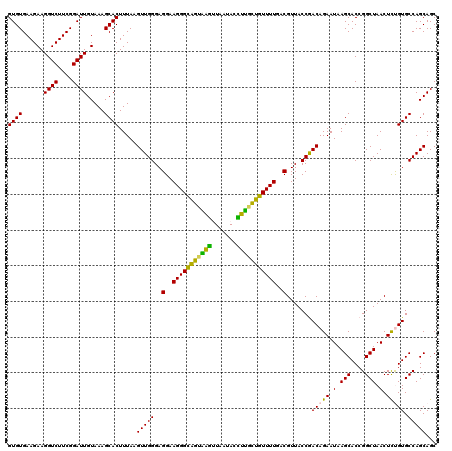
| Location | 6,044,265 – 6,044,385 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.72 |
| Mean single sequence MFE | -35.77 |
| Consensus MFE | -38.04 |
| Energy contribution | -35.93 |
| Covariance contribution | -2.11 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.13 |
| Structure conservation index | 1.06 |
| SVM decision value | 2.64 |
| SVM RNA-class probability | 0.995969 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044265 120 - 6264404 AUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUAAGUUAAUACCUUGCUGUUUUGACGU .(((((...(((((....))))).)))))(((.((.....((((......((((....)))).....)))).....)))))..(..(((((((((((.......)))))))))))..).. ( -37.80) >pss.1919 3852197 120 - 6093698 AUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUUACCUAAUACGUGAUUGUUUUGACGU .(((((...(((((....))))).)))))(((.((.....((((......((((....)))).....)))).....)))))..(..(((((((((((.......)))))))))))..).. ( -35.60) >psp.1833 3636213 120 - 5928787 AUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUUACCUAAUACGUGAUUGUUUUGACGU .(((((...(((((....))))).)))))(((.((.....((((......((((....)))).....)))).....)))))..(..(((((((((((.......)))))))))))..).. ( -35.60) >pst.2031 3874201 120 - 6397126 AUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUUACCUAAUACGUAUCUGUUUUGACGU .(((((...(((((....))))).)))))(((.((.....((((......((((....)))).....)))).....)))))..(..(((((((.(((.......))).)))))))..).. ( -33.70) >pfo.3002 5731081 120 - 6438405 AUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGUUGUAGAUUAAUACUCUGCAAUUUUGACGU .(((((...(((((....))))).)))))(((.((.....((((......((((....)))).....)))).....)))))..(..(((((((((((.......)))))))))))..).. ( -36.30) >pf5.3378 6387472 120 - 7074893 AUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUUACCUAAUACGUGAUUGUUUUGACGU .(((((...(((((....))))).)))))(((.((.....((((......((((....)))).....)))).....)))))..(..(((((((((((.......)))))))))))..).. ( -35.60) >consensus AUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUGGGAGGAAGGGCAGUUACCUAAUACGUGACUGUUUUGACGU .(((((...(((((....))))).)))))(((.((.....((((......((((....)))).....)))).....)))))..(..(((((((((((.......)))))))))))..).. (-38.04 = -35.93 + -2.11)

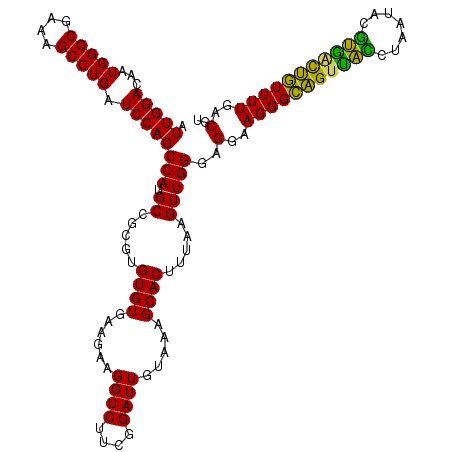
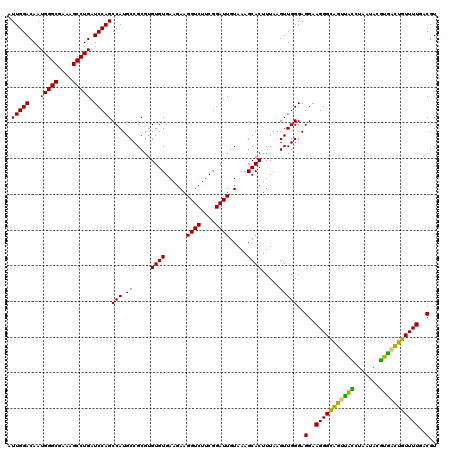
| Location | 6,044,305 – 6,044,425 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 100.00 |
| Mean single sequence MFE | -31.10 |
| Consensus MFE | -31.10 |
| Energy contribution | -31.10 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -0.74 |
| Structure conservation index | 1.00 |
| SVM decision value | 0.41 |
| SVM RNA-class probability | 0.723730 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044305 120 + 6264404 CAACUUAAAGUGCUUUACAAUCCGAAGACCUUCUUCACACACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUC .......................((((.....))))........(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))) ( -31.10) >ppk.94 6004623 120 - 6181863 CAACUUAAAGUGCUUUACAAUCCGAAGACCUUCUUCACACACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUC .......................((((.....))))........(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))) ( -31.10) >psp.1833 3636253 120 + 5928787 CAACUUAAAGUGCUUUACAAUCCGAAGACCUUCUUCACACACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUC .......................((((.....))))........(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))) ( -31.10) >pst.2031 3874241 120 + 6397126 CAACUUAAAGUGCUUUACAAUCCGAAGACCUUCUUCACACACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUC .......................((((.....))))........(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))) ( -31.10) >pfo.3002 5731121 120 + 6438405 CAACUUAAAGUGCUUUACAAUCCGAAGACCUUCUUCACACACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUC .......................((((.....))))........(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))) ( -31.10) >pf5.3378 6387512 120 + 7074893 CAACUUAAAGUGCUUUACAAUCCGAAGACCUUCUUCACACACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUC .......................((((.....))))........(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))) ( -31.10) >consensus CAACUUAAAGUGCUUUACAAUCCGAAGACCUUCUUCACACACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUC .......................((((.....))))........(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))) (-31.10 = -31.10 + -0.00)
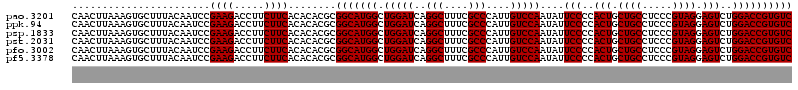
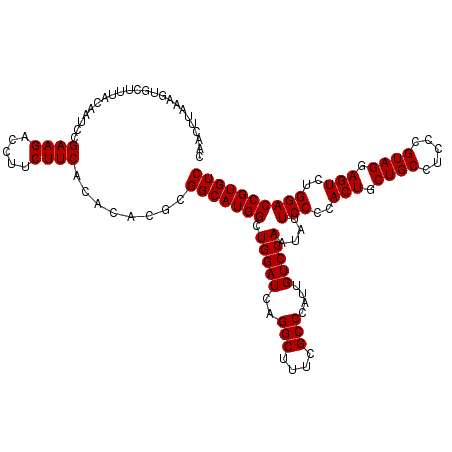
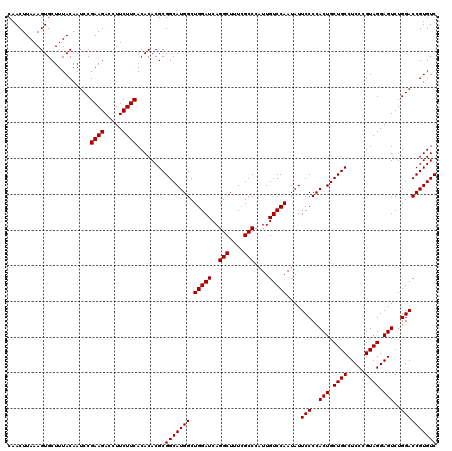
| Location | 6,044,305 – 6,044,425 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 100.00 |
| Mean single sequence MFE | -41.10 |
| Consensus MFE | -41.10 |
| Energy contribution | -41.10 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.35 |
| Structure conservation index | 1.00 |
| SVM decision value | 2.18 |
| SVM RNA-class probability | 0.989671 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044305 120 - 6264404 GACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUG ...(((((((...((((....))))(((.((((((.(....(((((...(((((....))))).)))))...).)))).)))))((((.....)))))))))))................ ( -41.10) >ppk.94 6004623 120 + 6181863 GACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUG ...(((((((...((((....))))(((.((((((.(....(((((...(((((....))))).)))))...).)))).)))))((((.....)))))))))))................ ( -41.10) >psp.1833 3636253 120 - 5928787 GACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUG ...(((((((...((((....))))(((.((((((.(....(((((...(((((....))))).)))))...).)))).)))))((((.....)))))))))))................ ( -41.10) >pst.2031 3874241 120 - 6397126 GACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUG ...(((((((...((((....))))(((.((((((.(....(((((...(((((....))))).)))))...).)))).)))))((((.....)))))))))))................ ( -41.10) >pfo.3002 5731121 120 - 6438405 GACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUG ...(((((((...((((....))))(((.((((((.(....(((((...(((((....))))).)))))...).)))).)))))((((.....)))))))))))................ ( -41.10) >pf5.3378 6387512 120 - 7074893 GACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUG ...(((((((...((((....))))(((.((((((.(....(((((...(((((....))))).)))))...).)))).)))))((((.....)))))))))))................ ( -41.10) >consensus GACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGUGUGUGAAGAAGGUCUUCGGAUUGUAAAGCACUUUAAGUUG ...(((((((...((((....))))(((.((((((.(....(((((...(((((....))))).)))))...).)))).)))))((((.....)))))))))))................ (-41.10 = -41.10 + 0.00)

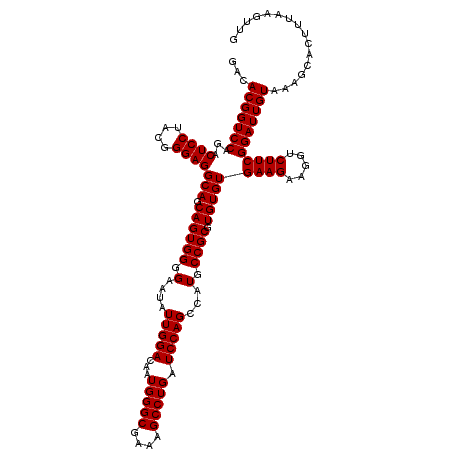
| Location | 6,044,345 – 6,044,465 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 100.00 |
| Mean single sequence MFE | -36.20 |
| Consensus MFE | -36.20 |
| Energy contribution | -36.20 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -0.93 |
| Structure conservation index | 1.00 |
| SVM decision value | 0.96 |
| SVM RNA-class probability | 0.889489 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044345 120 + 6264404 ACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUAC ..(.(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))).).........(((((((.((.........)).))))))) ( -36.20) >ppk.94 6004663 120 - 6181863 ACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUAC ..(.(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))).).........(((((((.((.........)).))))))) ( -36.20) >psp.1833 3636293 120 + 5928787 ACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUAC ..(.(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))).).........(((((((.((.........)).))))))) ( -36.20) >pst.2031 3874281 120 + 6397126 ACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUAC ..(.(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))).).........(((((((.((.........)).))))))) ( -36.20) >pfo.3002 5731161 120 + 6438405 ACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUAC ..(.(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))).).........(((((((.((.........)).))))))) ( -36.20) >pf5.3378 6387552 120 + 7074893 ACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUAC ..(.(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))).).........(((((((.((.........)).))))))) ( -36.20) >consensus ACGCGGCAUGGCUGGAUCAGGCUUUCGCCCAUUGUCCAAUAUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUAC ..(.(((((((.(((((..(((....)))....)))))....(((..(((.((((.....)))).)))..)))))))))).).........(((((((.((.........)).))))))) (-36.20 = -36.20 + -0.00)

| Location | 6,044,345 – 6,044,465 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 100.00 |
| Mean single sequence MFE | -47.10 |
| Consensus MFE | -47.10 |
| Energy contribution | -47.10 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.61 |
| Structure conservation index | 1.00 |
| SVM decision value | 4.73 |
| SVM RNA-class probability | 0.999943 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044345 120 - 6264404 GUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGU ((.(((((((.........))))))).)).(((((.(((.....)))))))).((((....))))((.(((.(((......(((((...(((((....))))).))))))))))).)).. ( -47.10) >ppk.94 6004663 120 + 6181863 GUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGU ((.(((((((.........))))))).)).(((((.(((.....)))))))).((((....))))((.(((.(((......(((((...(((((....))))).))))))))))).)).. ( -47.10) >psp.1833 3636293 120 - 5928787 GUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGU ((.(((((((.........))))))).)).(((((.(((.....)))))))).((((....))))((.(((.(((......(((((...(((((....))))).))))))))))).)).. ( -47.10) >pst.2031 3874281 120 - 6397126 GUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGU ((.(((((((.........))))))).)).(((((.(((.....)))))))).((((....))))((.(((.(((......(((((...(((((....))))).))))))))))).)).. ( -47.10) >pfo.3002 5731161 120 - 6438405 GUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGU ((.(((((((.........))))))).)).(((((.(((.....)))))))).((((....))))((.(((.(((......(((((...(((((....))))).))))))))))).)).. ( -47.10) >pf5.3378 6387552 120 - 7074893 GUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGU ((.(((((((.........))))))).)).(((((.(((.....)))))))).((((....))))((.(((.(((......(((((...(((((....))))).))))))))))).)).. ( -47.10) >consensus GUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGGACAAUGGGCGAAAGCCUGAUCCAGCCAUGCCGCGU ((.(((((((.........))))))).)).(((((.(((.....)))))))).((((....))))((.(((.(((......(((((...(((((....))))).))))))))))).)).. (-47.10 = -47.10 + 0.00)
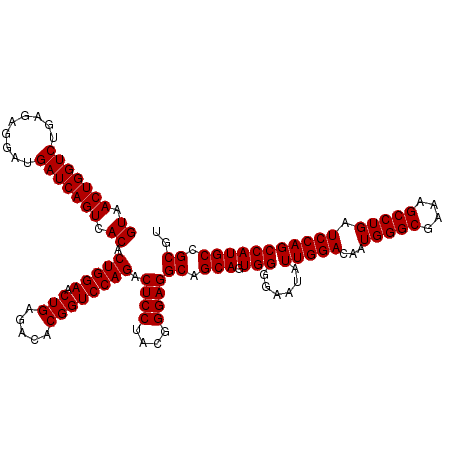
| Location | 6,044,385 – 6,044,505 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.17 |
| Mean single sequence MFE | -32.60 |
| Consensus MFE | -32.78 |
| Energy contribution | -32.95 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.03 |
| Mean z-score | -0.75 |
| Structure conservation index | 1.01 |
| SVM decision value | 0.64 |
| SVM RNA-class probability | 0.807300 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044385 120 + 6264404 AUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUACGGAUCGUCGCCUUGGUAGGCCUUUACCCCACCAACUAGCU ..........((((....((((((..(((((((..(((..((((((.......)))))).)))..)).)))))...)))))).........(((((.((.......)).))))).)))). ( -30.40) >pau.3340 6317304 120 + 6537648 AUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUACGGAUCGUCGCCUUGGUAGGCCUUUACCCCACCAACUAGCU ..........((((....((((((..(((((((..(((..((((((.......)))))).)))..)).)))))...)))))).........(((((.((.......)).))))).)))). ( -30.40) >ppk.94 6004703 120 - 6181863 AUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUACGGAUCGUCGCCUUGGUGAGCCAUUACCCCACCAACUAGCU ..........((((....((((((..(((((((..(((..((((((.......)))))).)))..)).)))))...)))))).........((((((...........)))))).)))). ( -30.40) >pst.2031 3874321 120 + 6397126 AUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUACGGAUCGUCGCCUUGGUGAGCCAUUACCUCACCAACUAGCU ..........((((....((((((..(((((((..(((..((((((.......)))))).)))..)).)))))...)))))).........((((((((.......)))))))).)))). ( -34.80) >pfo.3002 5731201 120 + 6438405 AUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUACGGAUCGUCGCCUUGGUGAGCCAUUACCUCACCAACUAGCU ..........((((....((((((..(((((((..(((..((((((.......)))))).)))..)).)))))...)))))).........((((((((.......)))))))).)))). ( -34.80) >pf5.3378 6387592 120 + 7074893 AUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUACGGAUCGUCGCCUUGGUGAGCCAUUACCUCACCAACUAGCU ..........((((....((((((..(((((((..(((..((((((.......)))))).)))..)).)))))...)))))).........((((((((.......)))))))).)))). ( -34.80) >consensus AUUCCCCACUGCUGCCUCCCGUAGGAGUCUGGACCGUGUCUCAGUUCCAGUGUGACUGAUCAUCCUCUCAGACCAGUUACGGAUCGUCGCCUUGGUGAGCCAUUACCCCACCAACUAGCU ..........((((....((((((..(((((((..(((..((((((.......)))))).)))..)).)))))...)))))).........((((((((.......)))))))).)))). (-32.78 = -32.95 + 0.17)
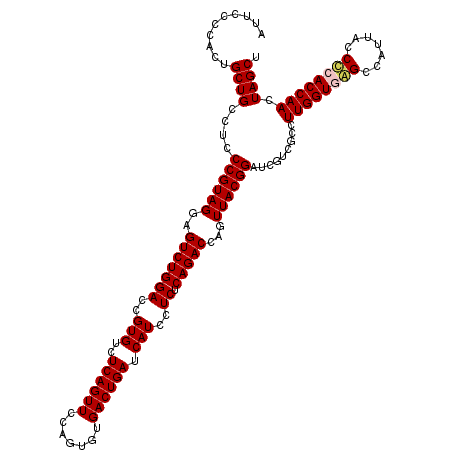
| Location | 6,044,385 – 6,044,505 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.17 |
| Mean single sequence MFE | -44.78 |
| Consensus MFE | -45.48 |
| Energy contribution | -44.78 |
| Covariance contribution | -0.69 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.17 |
| Structure conservation index | 1.02 |
| SVM decision value | 4.71 |
| SVM RNA-class probability | 0.999941 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044385 120 - 6264404 AGCUAGUUGGUGGGGUAAAGGCCUACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAU .(((..((((((((.......))))))))))).....(((...(((((((.........)))))))(((.(((((.(((.....)))))))).((((....))))......))).))).. ( -44.80) >pau.3340 6317304 120 - 6537648 AGCUAGUUGGUGGGGUAAAGGCCUACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAU .(((..((((((((.......))))))))))).....(((...(((((((.........)))))))(((.(((((.(((.....)))))))).((((....))))......))).))).. ( -44.80) >ppk.94 6004703 120 + 6181863 AGCUAGUUGGUGGGGUAAUGGCUCACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAU .(((..((((((((.......))))))))))).....(((...(((((((.........)))))))(((.(((((.(((.....)))))))).((((....))))......))).))).. ( -44.10) >pst.2031 3874321 120 - 6397126 AGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAU .(((..((((((((.......))))))))))).....(((...(((((((.........)))))))(((.(((((.(((.....)))))))).((((....))))......))).))).. ( -45.00) >pfo.3002 5731201 120 - 6438405 AGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAU .(((..((((((((.......))))))))))).....(((...(((((((.........)))))))(((.(((((.(((.....)))))))).((((....))))......))).))).. ( -45.00) >pf5.3378 6387592 120 - 7074893 AGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAU .(((..((((((((.......))))))))))).....(((...(((((((.........)))))))(((.(((((.(((.....)))))))).((((....))))......))).))).. ( -45.00) >consensus AGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGAGACACGGUCCAGACUCCUACGGGAGGCAGCAGUGGGGAAU .(((..((((((((.......))))))))))).....(((...(((((((.........)))))))(((.(((((.(((.....)))))))).((((....))))......))).))).. (-45.48 = -44.78 + -0.69)

| Location | 6,044,425 – 6,044,545 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.89 |
| Mean single sequence MFE | -39.93 |
| Consensus MFE | -40.68 |
| Energy contribution | -39.48 |
| Covariance contribution | -1.19 |
| Combinations/Pair | 1.13 |
| Mean z-score | -0.81 |
| Structure conservation index | 1.02 |
| SVM decision value | 1.02 |
| SVM RNA-class probability | 0.901995 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044425 120 - 6264404 UCUUCGGACCUCACGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGGGGUAAAGGCCUACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGA ...((((.((((..(((........)))((((..((((((.(((..((((((((.......)))))))))))...))))))...)))).....))))....((((.....))))..)))) ( -39.40) >pau.3340 6317344 120 - 6537648 UCUUCGGACCUCACGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGGGGUAAAGGCCUACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGA ...((((.((((..(((........)))((((..((((((.(((..((((((((.......)))))))))))...))))))...)))).....))))....((((.....))))..)))) ( -39.40) >pss.1919 3852357 120 - 6093698 CCUUCGGGCCUUGCGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGA ((((((((((....(((........)))..((..((((((.(((..((((((((.......)))))))))))...))))))...))))))))).)))....((((((......).))))) ( -40.70) >ppk.94 6004743 120 + 6181863 CCUUCGGGCCUUGCGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGGGGUAAUGGCUCACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGA ((((((((((....(((........)))..((..((((((.(((..((((((((.......)))))))))))...))))))...))))))))).)))....((((((......).))))) ( -39.80) >pfo.3002 5731241 120 - 6438405 CCUUCGGGCCUUGCGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGA ((((((((((....(((........)))..((..((((((.(((..((((((((.......)))))))))))...))))))...))))))))).)))....((((((......).))))) ( -40.70) >pf5.3378 6387632 120 - 7074893 UCUUCGGACCUCACGCUAUUAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGA ...((((.((((..(((........)))((((..((((((.(((..((((((((.......)))))))))))...))))))...)))).....))))....((((.....))))..)))) ( -39.60) >consensus CCUUCGGACCUCACGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCCGUAACUGGUCUGAGAGGAUGAUCAGUCACACUGGAACUGA ((((((((((....(((........)))..((..((((((.(((..((((((((.......)))))))))))...))))))...))))))))).)))....((((((......).))))) (-40.68 = -39.48 + -1.19)
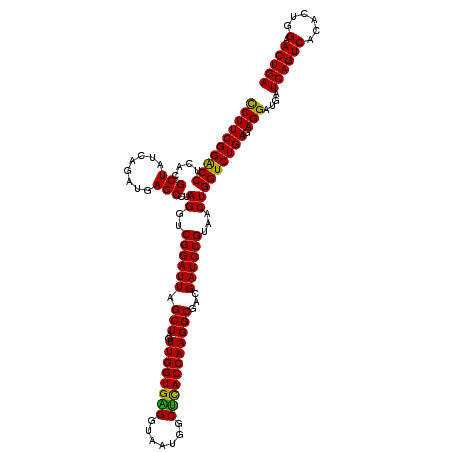
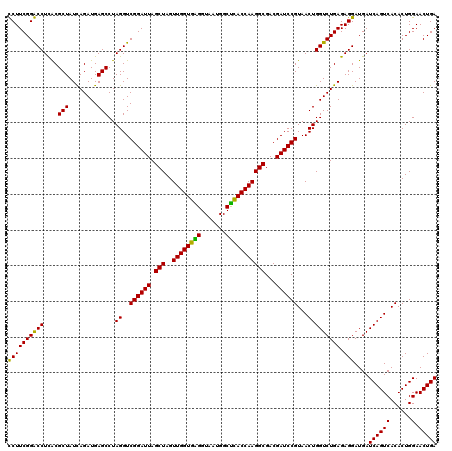
| Location | 6,044,465 – 6,044,585 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.50 |
| Mean single sequence MFE | -42.17 |
| Consensus MFE | -42.09 |
| Energy contribution | -39.90 |
| Covariance contribution | -2.19 |
| Combinations/Pair | 1.18 |
| Mean z-score | -0.87 |
| Structure conservation index | 1.00 |
| SVM decision value | 0.76 |
| SVM RNA-class probability | 0.842527 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044465 120 - 6264404 GCGCUAAUACCGCAUACGUCCUGAGGGAGAAAGUGGGGGAUCUUCGGACCUCACGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGGGGUAAAGGCCUACCAAGGCGACGAUCC ((((((((.(((....((((.(((...((...((((((..((....)))))))).)).))))))).((....)))))))))))...((((((((.......)))))))).))........ ( -42.10) >pau.3340 6317384 120 - 6537648 GCGCUAAUACCGCAUACGUCCUGAGGGAGAAAGUGGGGGAUCUUCGGACCUCACGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGGGGUAAAGGCCUACCAAGGCGACGAUCC ((((((((.(((....((((.(((...((...((((((..((....)))))))).)).))))))).((....)))))))))))...((((((((.......)))))))).))........ ( -42.10) >ppk.94 6004783 120 + 6181863 ACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGACCUUCGGGCCUUGCGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGGGGUAAUGGCUCACCAAGGCGACGAUCC ..((((((.((((.....(((....)))((.(((.((((.((....))))))..))).))............).)))))))))...((((((((.......))))))))(....)..... ( -41.30) >pst.2031 3874401 120 - 6397126 ACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGACCUUCGGGCCUUGCGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCC ..((((((.((((.....(((....)))((.(((.((((.((....))))))..))).))............).)))))))))...((((((((.......))))))))(....)..... ( -42.20) >pfo.3002 5731281 120 - 6438405 ACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGACCUUCGGGCCUUGCGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCC ..((((((.((((.....(((....)))((.(((.((((.((....))))))..))).))............).)))))))))...((((((((.......))))))))(....)..... ( -42.20) >pf5.3378 6387672 120 - 7074893 GCGCUAAUACCGCAUACGUCCUACGGGAGAAAGUGGGGGAUCUUCGGACCUCACGCUAUUAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCC ((.((((((..((.....(((....)))....((((((..((....))))))))))))))))....))...(((((.....(((..((((((((.......)))))))))))..))))). ( -43.10) >consensus ACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGACCUUCGGACCUCACGCUAUCAGAUGAGCCUAGGUCGGAUUAGCUAGUUGGUGAGGUAAUGGCUCACCAAGGCGACGAUCC ..((((((.((((.....(((....)))....((((((..((....))))))))(((........)))....).)))))))))...((((((((.......))))))))(....)..... (-42.09 = -39.90 + -2.19)
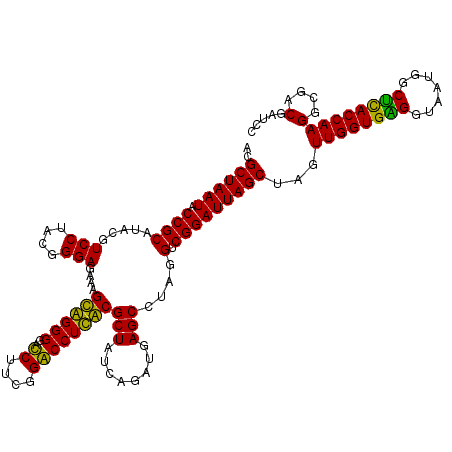
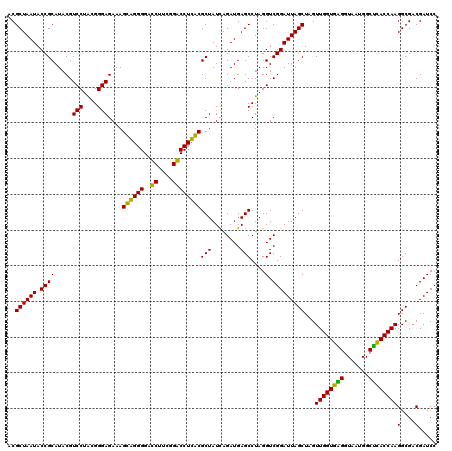
| Location | 6,044,505 – 6,044,625 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.06 |
| Mean single sequence MFE | -48.35 |
| Consensus MFE | -48.74 |
| Energy contribution | -45.97 |
| Covariance contribution | -2.77 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.73 |
| Structure conservation index | 1.01 |
| SVM decision value | 5.36 |
| SVM RNA-class probability | 0.999984 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044505 120 - 6264404 AGGAAUCUGCCUGGUAGUGGGGGAUAACGUCCGGAAACGGGCGCUAAUACCGCAUACGUCCUGAGGGAGAAAGUGGGGGAUCUUCGGACCUCACGCUAUCAGAUGAGCCUAGGUCGGAUU ...((((((((((((.(((.((..((.((((((....)))))).))...)).))).((((.(((...((...((((((..((....)))))))).)).))))))).))).))).)))))) ( -50.80) >pau.3340 6317424 120 - 6537648 AGGAAUCUGCCUGGUAGUGGGGGAUAACGUCCGGAAACGGGCGCUAAUACCGCAUACGUCCUGAGGGAGAAAGUGGGGGAUCUUCGGACCUCACGCUAUCAGAUGAGCCUAGGUCGGAUU ...((((((((((((.(((.((..((.((((((....)))))).))...)).))).((((.(((...((...((((((..((....)))))))).)).))))))).))).))).)))))) ( -50.80) >ppk.94 6004823 120 + 6181863 AGGAAUCUGCCUGGUAGUGGGGGACAACGUUUCGAAAGGAACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGACCUUCGGGCCUUGCGCUAUCAGAUGAGCCUAGGUCGGAUU ...((((((((((((.(((.((.....((((((....))))))......)).)))...(((....)))((.(((.((((.((....))))))..))).))......))).))).)))))) ( -46.90) >pst.2031 3874441 120 - 6397126 AGGAAUCUGCCUGGUAGUGGGGGAUAACGCUCGGAAACGGACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGACCUUCGGGCCUUGCGCUAUCAGAUGAGCCUAGGUCGGAUU ...((((((((((((.(((.((..((.((..((....))..)).))...)).)))...(((....)))((.(((.((((.((....))))))..))).))......))).))).)))))) ( -45.40) >pfo.3002 5731321 120 - 6438405 AGGAAUCUGCCUGGUAGUGGGGGACAACGUUUCGAAAGGAACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGACCUUCGGGCCUUGCGCUAUCAGAUGAGCCUAGGUCGGAUU ...((((((((((((.(((.((.....((((((....))))))......)).)))...(((....)))((.(((.((((.((....))))))..))).))......))).))).)))))) ( -46.90) >pf5.3378 6387712 120 - 7074893 AGGAAUCUGCCUAGUAGUGGGGGAUAACGUCCGGAAACGGGCGCUAAUACCGCAUACGUCCUACGGGAGAAAGUGGGGGAUCUUCGGACCUCACGCUAUUAGAUGAGCCUAGGUCGGAUU ...(((((((((((..((((((.....((((((....))))))......((.(((...(((....)))....))).))..((....))))))))(((........)))))))).)))))) ( -49.30) >consensus AGGAAUCUGCCUGGUAGUGGGGGAUAACGUCCGGAAACGGACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGACCUUCGGACCUCACGCUAUCAGAUGAGCCUAGGUCGGAUU ...(((((((((((..(((.((.....((((((....))))))......)).)))...(((....)))....((((((..((....))))))))(((........)))))))).)))))) (-48.74 = -45.97 + -2.77)

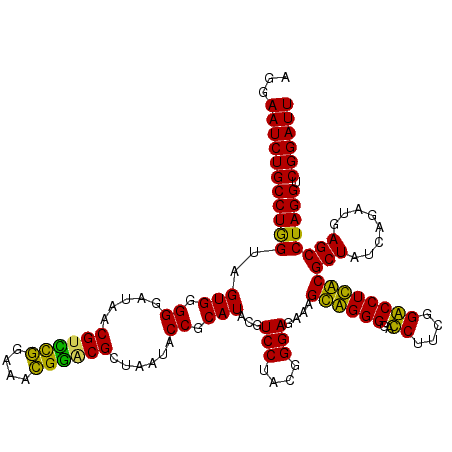
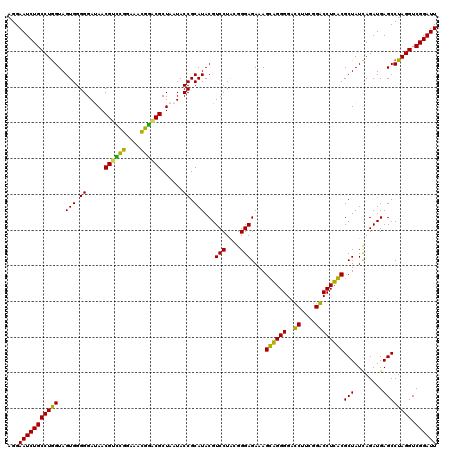
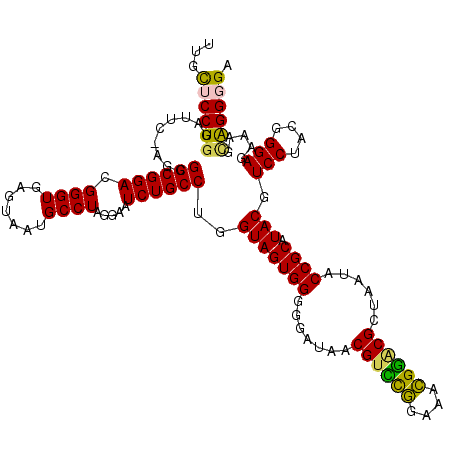
| Location | 6,044,545 – 6,044,664 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.36 |
| Mean single sequence MFE | -42.78 |
| Consensus MFE | -39.94 |
| Energy contribution | -38.67 |
| Covariance contribution | -1.27 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.93 |
| SVM decision value | 0.77 |
| SVM RNA-class probability | 0.846702 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6044545 119 - 6264404 UUGCUCCUGGAUUC-AGCGGCGGACGGGUGAGUAAUGCCUAGGAAUCUGCCUGGUAGUGGGGGAUAACGUCCGGAAACGGGCGCUAAUACCGCAUACGUCCUGAGGGAGAAAGUGGGGGA ...((((((..(((-..(..((((((.........((((.(((......)))))))(((.((..((.((((((....)))))).))...)).))).))))).)..)..)))..)))))). ( -42.70) >pau.3340 6317464 119 - 6537648 UUGCUCCUGGAUUC-AGCGGCGGACGGGUGAGUAAUGCCUAGGAAUCUGCCUGGUAGUGGGGGAUAACGUCCGGAAACGGGCGCUAAUACCGCAUACGUCCUGAGGGAGAAAGUGGGGGA ...((((((..(((-..(..((((((.........((((.(((......)))))))(((.((..((.((((((....)))))).))...)).))).))))).)..)..)))..)))))). ( -42.70) >ppk.94 6004863 119 + 6181863 UUGCUCCUUGAUUC-AGCGGCGGACGGGUGAGUAAUGCCUAGGAAUCUGCCUGGUAGUGGGGGACAACGUUUCGAAAGGAACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGA ....((((((.(((-.((((((((.((((.......)))).....)))))).((((...(....)..((((((....))))))....)))))).....(((....))))))..)))))). ( -42.00) >pst.2031 3874481 120 - 6397126 UUGUACCUGGUGGCGAGCGGCGGACGGGUGAGUAAUGCCUAGGAAUCUGCCUGGUAGUGGGGGAUAACGCUCGGAAACGGACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGA .....((((((((..(((((((...((((.......))))....((((.((.......)).))))..)))(((....))).))))....)))).....(((....))).....))))... ( -40.50) >pfo.3002 5731361 119 - 6438405 UUGCUCCUGGAUUC-AGCGGCGGACGGGUGAGUAAUGCCUAGGAAUCUGCCUGGUAGUGGGGGACAACGUUUCGAAAGGAACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGA ...((((((..(((-.((((((((.((((.......)))).....)))))).((((...(....)..((((((....))))))....)))))).....(((....))))))..)))))). ( -45.00) >pf5.3378 6387752 120 - 7074893 UUGUACCUGGUGGCGAGCGGCGGACGGGUGAGUAAUGCCUAGGAAUCUGCCUAGUAGUGGGGGAUAACGUCCGGAAACGGGCGCUAAUACCGCAUACGUCCUACGGGAGAAAGUGGGGGA ..(((..(((((...((((.(((((((((.......)))).(..((((.((.......)).))))..)))))(....).).))))..)))))..))).((((((........)))))).. ( -43.80) >consensus UUGCUCCUGGAUUC_AGCGGCGGACGGGUGAGUAAUGCCUAGGAAUCUGCCUGGUAGUGGGGGAUAACGUCCGGAAACGGACGCUAAUACCGCAUACGUCCUACGGGAGAAAGCAGGGGA ...((((((.........((((((.((((.......)))).....))))))..(((((((.......((((((....))))))......)))).))).(((....))).....)))))). (-39.94 = -38.67 + -1.27)

Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:05:10 2007