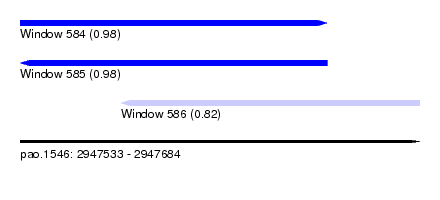
| Sequence ID | pao.1546 |
|---|---|
| Location | 2,947,533 – 2,947,684 |
| Length | 151 |
| Max. P | 0.984004 |

| Location | 2,947,533 – 2,947,649 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.29 |
| Mean single sequence MFE | -43.22 |
| Consensus MFE | -35.06 |
| Energy contribution | -32.82 |
| Covariance contribution | -2.24 |
| Combinations/Pair | 1.35 |
| Mean z-score | -3.59 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.77 |
| SVM RNA-class probability | 0.976209 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1546 2947533 116 + 6264404 CCCCGAAAAUG-CAAAAGCCCCGC-UCAGCGGGGCUUUUCAGUACGCGGAUAU--GGCGGAGGCGGUGAGAUUCGAACUCACGGAAGAGUCUCCCCUUCGACGGUUUUCAAGACCGUUGC ..(((....((-.(((((((((((-...))))))))))))).....)))....--((.((((((..((((.......))))(....).))))))))..((((((((.....)))))))). ( -50.50) >pel.1270 3551102 107 - 5888780 CCCCAAAAACGCAAAAAGCCCUGAAUAAUCAGGGCUUUG---------AAUGU--GGCGGAAGCGUAGAGAUUCGAACUCUAGGAUAGUUG--CCCAUCGACGGUUUUCAAGACCGUUGC ..((.....((((.((((((((((....)))))))))).---------..)))--)((....)).(((((.......))))))).......--.....((((((((.....)))))))). ( -39.50) >ppk.2304 4501271 107 + 6181863 CCUCAGAAACGCAAAAAGCCCUGAAUAAUCAGGGCUUUG---------AAUAU--GGCGGAAGCGUAGAGAUUCGAACUCUAGGAUAGUUG--CCCAUCGACGGUUUUCAAGACCGUUGC ..............((((((((((....)))))))))).---------.....--(((((...(.(((((.......))))).)....)))--))...((((((((.....)))))))). ( -37.60) >pst.1986 2625355 114 - 6397126 CCCCAGAAACG-ACAAAGCCCUGAAUAAUCAGGGCUUUGCGCUAC-CAAAUAU--GGCGGAGGCAUAGAGAUUCGAACUCUAGGACCUGUU--ACAGUCGGCAGUUUUCAAGACUGCUGC ..((......(-.(((((((((((....))))))))))))((((.-......)--)))))((((.(((((.......))))).).)))...--.....((((((((.....)))))))). ( -41.40) >pf5.2270 2736423 112 - 7074893 -GACAGACACA-AAAAAGCCCUGAAAAAUCAGGGCUUUUUCGU----UGAGAUUUGGCGGAGGCGUAGAGAUUCGAACUCUAGGACCUGUU--ACAGUCGGCGGUUUUCAAGACCGCUGC -....(((...-((((((((((((....))))))))))))(((----..(...)..))).((((.(((((.......))))).).)))...--...)))(((((((.....))))))).. ( -43.20) >pfo.1458 2845455 113 + 6438405 -GUCAGAAACG-ACAAAGCCCUGAAUAAUCAGGGCUUUGUCGUAC-UUAAGAU--GGCGGAGGCGAUGGGAUUCGAACUCAUGGACCUGUU--ACAGUCGACGGUUUUCAAGACCGUUGC -((((.(((((-((((((((((((....)))))))))))))))..-))....)--)))..((((.(((((.......))))).).)))...--.....((((((((.....)))))))). ( -47.10) >consensus CCCCAGAAACG_AAAAAGCCCUGAAUAAUCAGGGCUUUG__GU_____AAUAU__GGCGGAGGCGUAGAGAUUCGAACUCUAGGACCUGUU__ACAGUCGACGGUUUUCAAGACCGUUGC .............(((((((((((....))))))))))).................((....)).(((((.......)))))................((((((((.....)))))))). (-35.06 = -32.82 + -2.24)

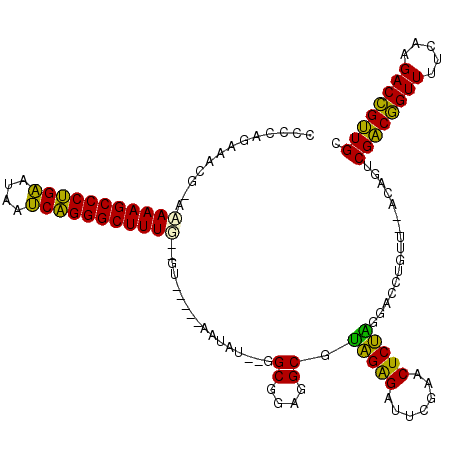
| Location | 2,947,533 – 2,947,649 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.29 |
| Mean single sequence MFE | -39.73 |
| Consensus MFE | -32.15 |
| Energy contribution | -29.05 |
| Covariance contribution | -3.10 |
| Combinations/Pair | 1.44 |
| Mean z-score | -2.94 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.96 |
| SVM RNA-class probability | 0.984004 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1546 2947533 116 - 6264404 GCAACGGUCUUGAAAACCGUCGAAGGGGAGACUCUUCCGUGAGUUCGAAUCUCACCGCCUCCGCC--AUAUCCGCGUACUGAAAAGCCCCGCUGA-GCGGGGCUUUUG-CAUUUUCGGGG ((.(((((.......))))).((((((....)))))).(((((.......))))).))((((((.--......))....(((((((((((((...-))))))))))).-)).....)))) ( -45.00) >pel.1270 3551102 107 + 5888780 GCAACGGUCUUGAAAACCGUCGAUGGG--CAACUAUCCUAGAGUUCGAAUCUCUACGCUUCCGCC--ACAUU---------CAAAGCCCUGAUUAUUCAGGGCUUUUUGCGUUUUUGGGG ((.(((((.......))))).(((((.--...))))).(((((.......))))).))..((.((--(.(..---------(((((((((((....))))))))))....)..).))))) ( -35.00) >ppk.2304 4501271 107 - 6181863 GCAACGGUCUUGAAAACCGUCGAUGGG--CAACUAUCCUAGAGUUCGAAUCUCUACGCUUCCGCC--AUAUU---------CAAAGCCCUGAUUAUUCAGGGCUUUUUGCGUUUCUGAGG ((((.(((...(((.......(((((.--...))))).(((((.......)))))...))).)))--.....---------.((((((((((....)))))))))))))).......... ( -33.70) >pst.1986 2625355 114 + 6397126 GCAGCAGUCUUGAAAACUGCCGACUGU--AACAGGUCCUAGAGUUCGAAUCUCUAUGCCUCCGCC--AUAUUUG-GUAGCGCAAAGCCCUGAUUAUUCAGGGCUUUGU-CGUUUCUGGGG ((((((((.......))))).((((..--....)))).(((((.......))))))))(((((((--(....))-))(((((((((((((((....))))))))))).-))))...)))) ( -45.10) >pf5.2270 2736423 112 + 7074893 GCAGCGGUCUUGAAAACCGCCGACUGU--AACAGGUCCUAGAGUUCGAAUCUCUACGCCUCCGCCAAAUCUCA----ACGAAAAAGCCCUGAUUUUUCAGGGCUUUUU-UGUGUCUGUC- ((((((((..(....)..)))).))))--...((((..(((((.......))))).)))).............----(((((((((((((((....))))))))))))-))).......- ( -40.00) >pfo.1458 2845455 113 - 6438405 GCAACGGUCUUGAAAACCGUCGACUGU--AACAGGUCCAUGAGUUCGAAUCCCAUCGCCUCCGCC--AUCUUAA-GUACGACAAAGCCCUGAUUAUUCAGGGCUUUGU-CGUUUCUGAC- ((...((..(((((...(((.((((..--....)))).)))..)))))..))....)).......--......(-(.(((((((((((((((....))))))))))))-)))..))...- ( -39.60) >consensus GCAACGGUCUUGAAAACCGUCGACGGG__AACAGGUCCUAGAGUUCGAAUCUCUACGCCUCCGCC__AUAUU_____AC__CAAAGCCCUGAUUAUUCAGGGCUUUUU_CGUUUCUGGGG ((.(((((.......))))).((((........)))).(((((.......))))).)).......................(((((((((((....)))))))))))............. (-32.15 = -29.05 + -3.10)
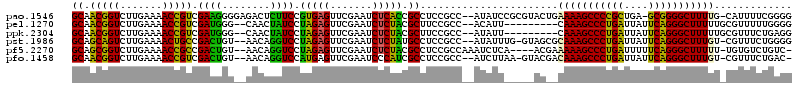
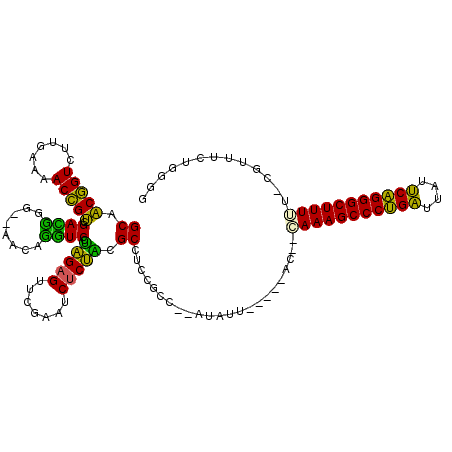
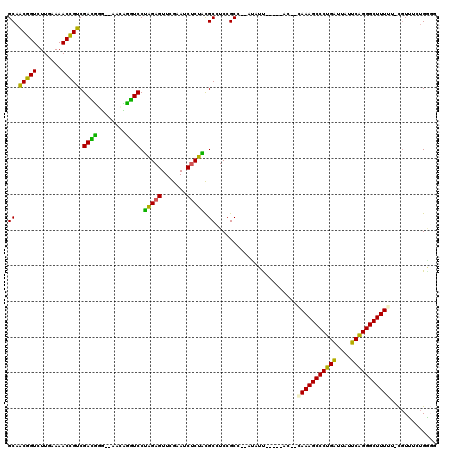
| Location | 2,947,571 – 2,947,684 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 77.63 |
| Mean single sequence MFE | -39.13 |
| Consensus MFE | -33.58 |
| Energy contribution | -30.58 |
| Covariance contribution | -3.00 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.19 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.821377 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1546 2947571 113 - 6264404 CCC----CGCGAUGCGGAGGUGUGGCCGAGUGGUUUAAGGCAACGGUCUUGAAAACCGUCGAAGGGGAGACUCUUCCGUGAGUUCGAAUCUCACCGCCUCCGCC--AUAUCCGCGUACU ...----(((((((((((((((.....((.(((((((((((....)))))))..))))))((((((....)))))).(((((.......)))))))))))))).--..)).)))).... ( -47.40) >pau.1463 3903309 113 + 6537648 CCC----CGCGAUGCGGAGGUGUGGCCGAGUGGUUUAAGGCAACGGUCUUGAAAACCGUCGAAGGGGAGACUCUUCCGUGAGUUCGAAUCUCACCGCCUCCGCC--AUAUCCGCGUACU ...----(((((((((((((((.....((.(((((((((((....)))))))..))))))((((((....)))))).(((((.......)))))))))))))).--..)).)))).... ( -47.40) >pel.1270 3551141 107 + 5888780 GCCGCAAAACCAGCUGGAAGCGUGGCCGAGUGGUUUAAGGCAACGGUCUUGAAAACCGUCGAUGGG--CAACUAUCCUAGAGUUCGAAUCUCUACGCUUCCGCC--ACAUU-------- ............((.((((((((((..((.(((((((((((....)))))))..))))))(((((.--...)))))...............)))))))))))).--.....-------- ( -35.90) >ppk.2304 4501310 107 - 6181863 GCCGCAAAACGAGCUGGAAGCGUGGCCGAGUGGUUUAAGGCAACGGUCUUGAAAACCGUCGAUGGG--CAACUAUCCUAGAGUUCGAAUCUCUACGCUUCCGCC--AUAUU-------- ((((.....)).)).((((((((((..((.(((((((((((....)))))))..))))))(((((.--...)))))...............))))))))))...--.....-------- ( -36.60) >pf5.2270 2736461 113 + 7074893 CCCGCAAUGCGAUCUGGAGGUGUGGCCGAGUGGUUUAAGGCAGCGGUCUUGAAAACCGCCGACUGU--AACAGGUCCUAGAGUUCGAAUCUCUACGCCUCCGCCAAAUCUCA----ACG ..(((...))).....(((((.((((((.((((((((((((....)))))))..))))))).....--...((((..(((((.......))))).))))..)))).))))).----... ( -35.50) >pfo.1458 2845493 112 - 6438405 CCCGCAACGCAA--CGGAGGUGUGGCCGAGUGGUUUAAGGCAACGGUCUUGAAAACCGUCGACUGU--AACAGGUCCAUGAGUUCGAAUCCCAUCGCCUCCGCC--AUCUUAA-GUACG ............--(((((((((((.(((((..((((((((....)))))))).))(((.((((..--....)))).)))...)))....))).))))))))..--.......-..... ( -32.00) >consensus CCCGCAACGCGAUCCGGAGGUGUGGCCGAGUGGUUUAAGGCAACGGUCUUGAAAACCGUCGAAGGG__AACACAUCCUAGAGUUCGAAUCUCAACGCCUCCGCC__AUAUC_____AC_ ..............((((((((.(((.(.((..((((((((....)))))))).))))))(((((......))))).(((((.......)))))))))))))................. (-33.58 = -30.58 + -3.00)

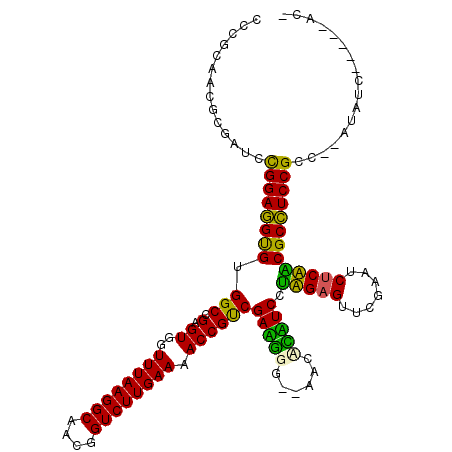
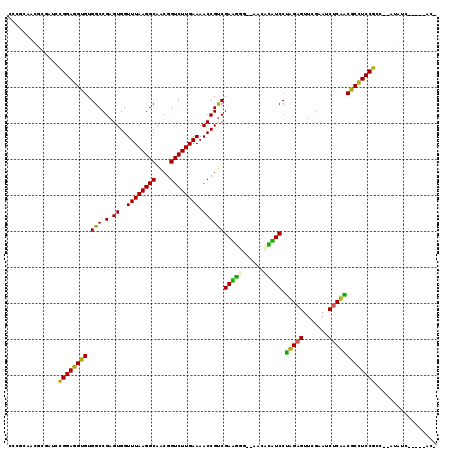
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:13:51 2007