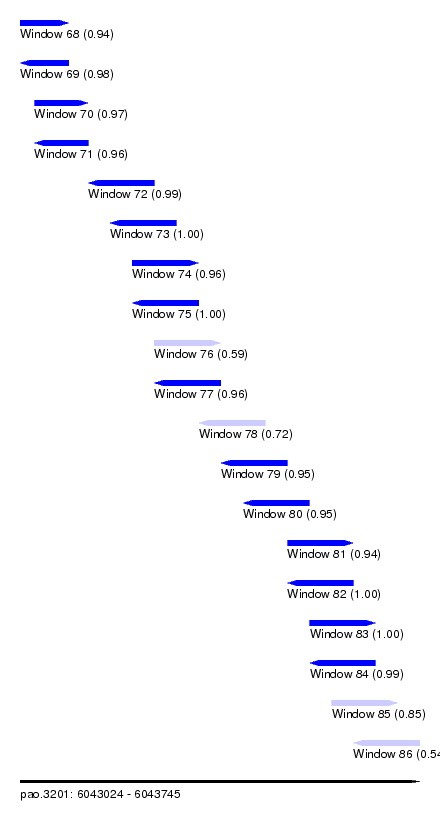
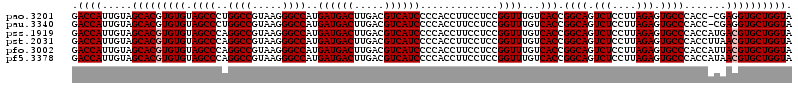
| Sequence ID | pao.3201 |
|---|---|
| Location | 6,043,024 – 6,043,745 |
| Length | 721 |
| Max. P | 0.999985 |

| Location | 6,043,024 – 6,043,112 |
|---|---|
| Length | 88 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 77.18 |
| Mean single sequence MFE | -28.87 |
| Consensus MFE | -23.41 |
| Energy contribution | -23.27 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.27 |
| SVM RNA-class probability | 0.938849 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
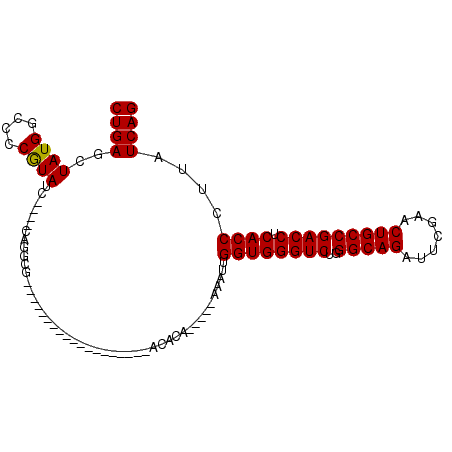
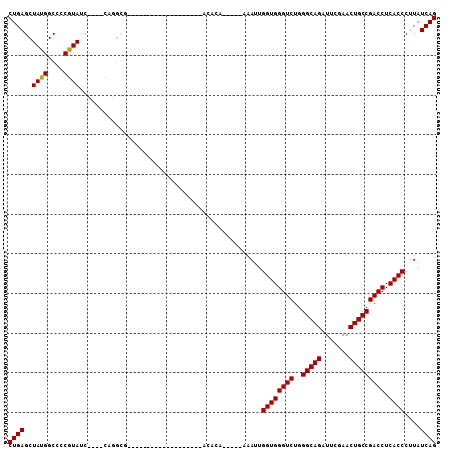
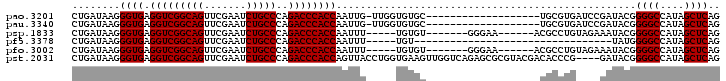
>pao.3201 6043024 88 + 6264404 CUGAGCUAUGGCCCCGUAUCGGAUCACGCA-------------------GCACACCAA-CAAUUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAG (((((((.((..((......))..))...)-------------------)).......-.....((((((((..(((((.......))))))))).))))....)))) ( -27.50) >pau.3340 6315943 88 + 6537648 CUGAGCUAUGGCCCCGUAUCGGAUCACGCA-------------------GCACACCAA-CAAUUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAG (((((((.((..((......))..))...)-------------------)).......-.....((((((((..(((((.......))))))))).))))....)))) ( -27.50) >psp.1833 3634910 90 + 5928787 CUGAGCUAUGGCCCCGUAUUUCUACAGGCGU------UUCCC-------ACACA-----AAAUUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAG ((((......(((..(((....))).)))..------.....-------.....-----.....((((((((..(((((.......))))))))).))))....)))) ( -26.60) >pf5.3378 6386189 70 + 7074893 CUGAGCUAUGGCCCCAUA---------------------------------ACA-----AAAUUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAG (((((.((((....))))---------------------------------.).-----.....((((((((..(((((.......))))))))).))))....)))) ( -24.30) >pfo.3002 5729820 90 + 6438405 CUGAGCUAUGGCCCCGUAUUUCUACAGGCGU------UUCCC-------ACACA-----AAAUUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAG ((((......(((..(((....))).)))..------.....-------.....-----.....((((((((..(((((.......))))))))).))))....)))) ( -26.60) >pst.2031 3872911 104 + 6397126 CUGAGCUAUGGCCCCGUAUC----CGGGUGUCGUACGCGCUCUGACCAACUUCACCAGGUAACUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAG ((((((((((((((((....----)))).)))))).))....(((.......((((((....))))))((((..(((((.......))))))))))))......)))) ( -40.70) >consensus CUGAGCUAUGGCCCCGUAUC____CAGGCG___________________ACACA_____AAAUUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAG ((((..((((....))))..............................................((((((((..(((((.......))))))))).))))....)))) (-23.41 = -23.27 + -0.14)

| Location | 6,043,024 – 6,043,112 |
|---|---|
| Length | 88 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 77.18 |
| Mean single sequence MFE | -34.38 |
| Consensus MFE | -24.50 |
| Energy contribution | -24.50 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.71 |
| SVM decision value | 1.85 |
| SVM RNA-class probability | 0.980140 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043024 88 - 6264404 CUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAAUUG-UUGGUGUGC-------------------UGCGUGAUCCGAUACGGGGCCAUAGCUCAG ((((....((((.(((((((((.......)))))..))))))))....(-(((.((.((-------------------(.((((......)))).))))))))))))) ( -33.60) >pau.3340 6315943 88 - 6537648 CUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAAUUG-UUGGUGUGC-------------------UGCGUGAUCCGAUACGGGGCCAUAGCUCAG ((((....((((.(((((((((.......)))))..))))))))....(-(((.((.((-------------------(.((((......)))).))))))))))))) ( -33.60) >psp.1833 3634910 90 - 5928787 CUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAAUUU-----UGUGU-------GGGAA------ACGCCUGUAGAAAUACGGGGCCAUAGCUCAG ((((....((((.(((((((((.......)))))..)))))))).....-----..(((-------((...------.).((((((....))))))..))))..)))) ( -35.40) >pf5.3378 6386189 70 - 7074893 CUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAAUUU-----UGU---------------------------------UAUGGGGCCAUAGCUCAG ((((....((((.(((((((((.......)))))..)))))))).....-----.((---------------------------------((((....)))))))))) ( -28.70) >pfo.3002 5729820 90 - 6438405 CUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAAUUU-----UGUGU-------GGGAA------ACGCCUGUAGAAAUACGGGGCCAUAGCUCAG ((((....((((.(((((((((.......)))))..)))))))).....-----..(((-------((...------.).((((((....))))))..))))..)))) ( -35.40) >pst.2031 3872911 104 - 6397126 CUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAGUUACCUGGUGAAGUUGGUCAGAGCGCGUACGACACCCG----GAUACGGGGCCAUAGCUCAG .......((((...((.(((.((.((..((((.((((...((((((....))))))...)))).))))...)).))....((((----....))))))))).)))).. ( -39.60) >consensus CUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAAUUU_____UGUGC___________________CGCCUG____GAUACGGGGCCAUAGCUCAG ........((((.(((((((((.......)))))..))))))))..................................................((((....)))).. (-24.50 = -24.50 + 0.00)
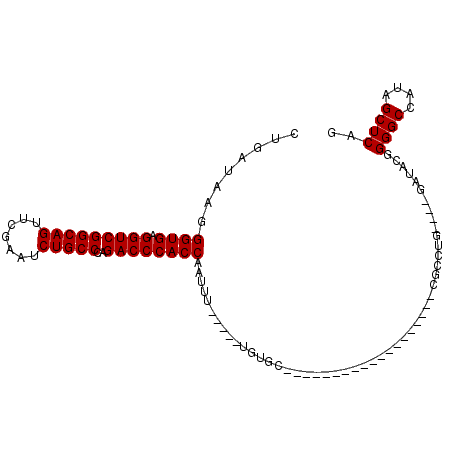
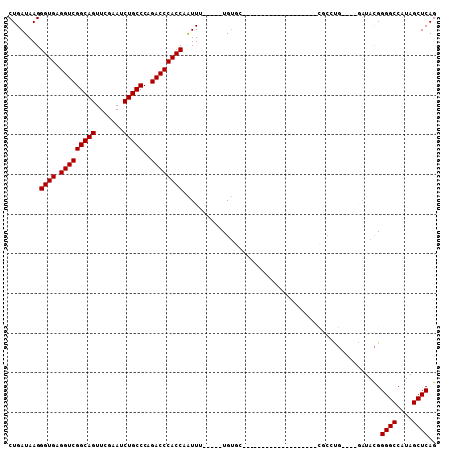
| Location | 6,043,050 – 6,043,147 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.41 |
| Mean single sequence MFE | -39.67 |
| Consensus MFE | -36.03 |
| Energy contribution | -35.62 |
| Covariance contribution | -0.41 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.78 |
| Structure conservation index | 0.91 |
| SVM decision value | 1.59 |
| SVM RNA-class probability | 0.965732 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043050 97 + 6264404 CGCA----------------------GCACACCAA-CAAUUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAGGGGUGCGCUCUAACCAACUGAGCUACAGACCCAAU ....----------------------....((((.-....))))((((((((((((.......))))).....((((((.....)))))).((((.........))))..)))))))... ( -38.00) >pss.1919 3850945 114 + 6093698 GGCGU------CUCUCUGACCAGCUAACUCACCAGUAAACUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAGGGGUGCGCUCUAACCAACUGAGCUACAGACCCGAA (((((------(.....)))..)))...(((((((....)))))((((((((((((.......))))).....((((((.....)))))).((((.........))))..))))))))). ( -45.80) >pel.69 5771594 84 - 5888780 ------------------------------------AUAUUGGUAGGUCUGGGCAGAUUUGAACUGCCGACCUCACCCUUAUCAGGGGUGCGCUCUAACCAACUGAGCUACAGACCUAUA ------------------------------------......((((((((((((((.......))))).....((((((.....)))))).((((.........))))..))))))))). ( -35.60) >pf5.3378 6386207 87 + 7074893 ----------------------------ACA-----AAAUUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAGGGGUGCGCUCUAACCAACUGAGCUACAGACCCAAU ----------------------------...-----.......(((((((((((((.......))))).....((((((.....)))))).((((.........))))..)))))))).. ( -36.10) >pfo.3002 5729846 99 + 6438405 GGCGU------UUCCC----------ACACA-----AAAUUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAGGGGUGCGCUCUAACCAACUGAGCUACAGACCCAAU .....------...((----------(....-----....)))(((((((((((((.......))))).....((((((.....)))))).((((.........))))..)))))))).. ( -37.00) >pst.2031 3872933 117 + 6397126 GGUGUCGUACGCGCUCUGACCAA---CUUCACCAGGUAACUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAGGGGUGCGCUCUAACCAACUGAGCUACAGACCCAAA ((.((((((.(((((((((....---...((((((....))))))((((..(((((.......))))))))).........))).))))))((((.........))))))).)))))... ( -45.50) >consensus GGCG_______________________CACA_____AAAUUGGUGGGUCUGGGCAGAUUCGAACUGCCGACCUCACCCUUAUCAGGGGUGCGCUCUAACCAACUGAGCUACAGACCCAAA ...........................................(((((((((((((.......))))).....((((((.....)))))).((((.........))))..)))))))).. (-36.03 = -35.62 + -0.41)

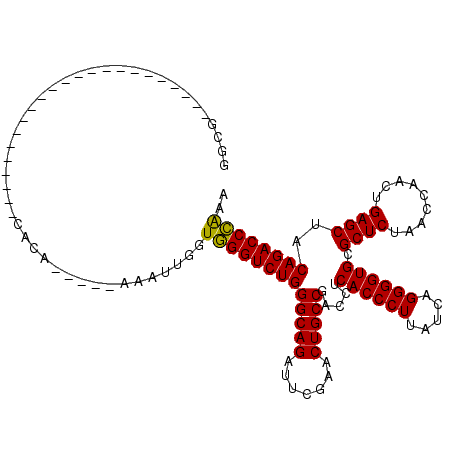
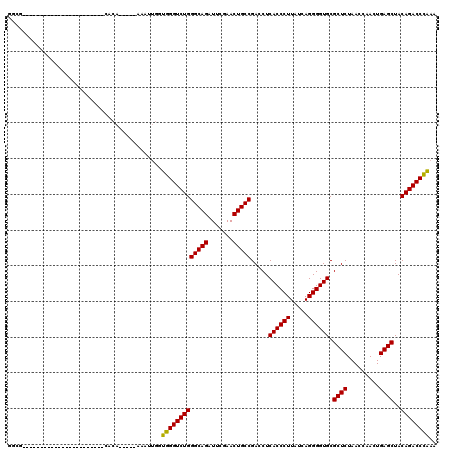
| Location | 6,043,050 – 6,043,147 |
|---|---|
| Length | 97 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.41 |
| Mean single sequence MFE | -38.18 |
| Consensus MFE | -33.33 |
| Energy contribution | -33.22 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.10 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.50 |
| SVM RNA-class probability | 0.959059 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043050 97 - 6264404 AUUGGGUCUGUAGCUCAGUUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAAUUG-UUGGUGUGC----------------------UGCG (((((((.....))))))).......((((((((...(((((.((((.(((((((((.......)))))..))))))))..)))-)))))))))----------------------)... ( -39.10) >pss.1919 3850945 114 - 6093698 UUCGGGUCUGUAGCUCAGUUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAGUUUACUGGUGAGUUAGCUGGUCAGAGAG------ACGCC (((((((((((((((.....)))))((.(.(((((.......))))).).))(((((.......))))))))))))(((((((.(((....))).)))))))..)))..------..... ( -44.60) >pel.69 5771594 84 + 5888780 UAUAGGUCUGUAGCUCAGUUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGCAGUUCAAAUCUGCCCAGACCUACCAAUAU------------------------------------ ..(((((((((((((.....)))))((.(.(((((.......))))).).))(((((.......))))))))))))).......------------------------------------ ( -32.10) >pf5.3378 6386207 87 - 7074893 AUUGGGUCUGUAGCUCAGUUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAAUUU-----UGU---------------------------- ..(((((((((((((.....)))))((.(.(((((.......))))).).))(((((.......))))))))))))).......-----...---------------------------- ( -34.00) >pfo.3002 5729846 99 - 6438405 AUUGGGUCUGUAGCUCAGUUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAAUUU-----UGUGU----------GGGAA------ACGCC ..(((((((((((((.....)))))((.(.(((((.......))))).).))(((((.......))))))))))))).......-----.((((----------.....------)))). ( -36.70) >pst.2031 3872933 117 - 6397126 UUUGGGUCUGUAGCUCAGUUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAGUUACCUGGUGAAG---UUGGUCAGAGCGCGUACGACACC ...(((((.(((((((.........((.(.(((((.......))))).).))(((((.......)))))..((((((((((....))))))...---..)))).))))...)))))).)) ( -42.60) >consensus AUUGGGUCUGUAGCUCAGUUGGUUAGAGCGCACCCCUGAUAAGGGUGAGGUCGGCAGUUCGAAUCUGCCCAGACCCACCAAUUU_____UGUG_______________________CGCC ..(((((((((((((.....)))))((.(.(((((.......))))).).))(((((.......)))))))))))))........................................... (-33.33 = -33.22 + -0.11)

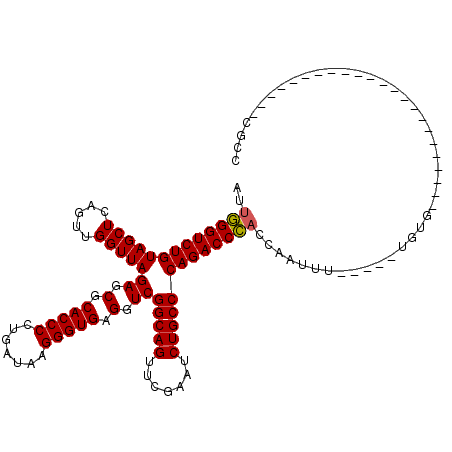
| Location | 6,043,147 – 6,043,266 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.66 |
| Mean single sequence MFE | -41.79 |
| Consensus MFE | -38.36 |
| Energy contribution | -38.50 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.10 |
| Structure conservation index | 0.92 |
| SVM decision value | 2.51 |
| SVM RNA-class probability | 0.994736 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043147 119 - 6264404 UGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGAAGAUCUCAGCUUCUUCAUAAGCUCCCACACGAAUUGCUUGAUUCACUGGUUA-GACG .(((((((.((....))....((((((((((....))))))))))..))))))).(((((((((((........))))).(((((.............))))).......)))))-)... ( -40.72) >pau.3340 6316066 119 - 6537648 UGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGAAGAUCUCAGCUUCUUCAUAAGCUCCCACACGAAUUGCUUGAUUCACUGGUUA-GACG .(((((((.((....))....((((((((((....))))))))))..))))))).(((((((((((........))))).(((((.............))))).......)))))-)... ( -40.72) >ppk.94 6003463 120 + 6181863 UGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGACGACAUCAGCCUGCUGAUGAGCUCCCACACGAAUUGCUUGAUUCAUUGUCAAAGACG ((((((...((((....((((.(((((((((....)))))))))((....)).))))...))))..((((((....)))))).)).))))...(((((....)))))...(((...))). ( -45.60) >pst.2031 3873082 119 - 6397126 UGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGACGAC-UCAGCUGCACCAUAAGCACCCACACGAAUUGCUUGAUUCAUUGAAGAAGACG ((((.(((.(((((...((((.(((((((((....)))))))))((....)).)))).....)))))-)))(((........))).))))...(((((....)))))............. ( -42.00) >pfo.3002 5729961 120 - 6438405 UGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGACGACAUCAGCUGCUCCAUAAGUUCCCACACGAAUUGCUUGAUUCAUUGAAGAAGACG ((((((((.(((((...((((.(((((((((....)))))))))((....)).)))).....))))).)))(((........))))))))...(((((....)))))............. ( -39.70) >pf5.3378 6386352 120 - 7074893 UGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGACGACAUCAGCUGCUUCAUAAGCUCCCACACGAAUUGCUUGAUUCAUUGAAGAAGACG ((((((((.(((((...((((.(((((((((....)))))))))((....)).)))).....))))).)))(((........))))))))...(((((....)))))............. ( -42.00) >consensus UGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGACGACAUCAGCUGCUUCAUAAGCUCCCACACGAAUUGCUUGAUUCAUUGAAGAAGACG ((((.(((.(((((...((((.(((((((((....)))))))))((....)).)))).....))))).)))(((........))).))))...(((((....)))))............. (-38.36 = -38.50 + 0.14)
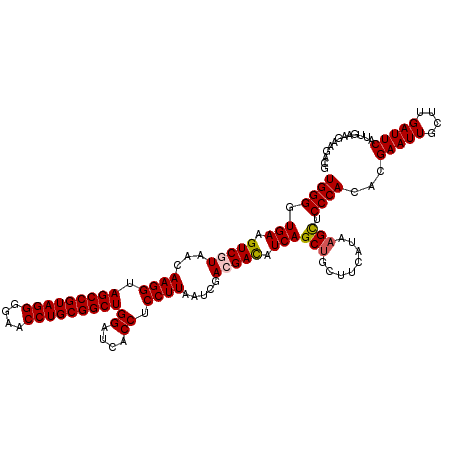
| Location | 6,043,186 – 6,043,306 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.39 |
| Mean single sequence MFE | -47.80 |
| Consensus MFE | -42.97 |
| Energy contribution | -42.97 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.89 |
| SVM RNA-class probability | 0.997592 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043186 120 - 6264404 UAACCGCAAGGGGGACGGUUACCACGGAGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGAAGAUCUCAGCUUCUUCA ...((....))((........))..((((((((((((((((.......))))))........(((((((((....))))))))))))))).))))......(((((........))))). ( -49.30) >pau.3340 6316105 120 - 6537648 UAACCGCAAGGGGGACGGUUACCACGGAGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGAAGAUCUCAGCUUCUUCA ...((....))((........))..((((((((((((((((.......))))))........(((((((((....))))))))))))))).))))......(((((........))))). ( -49.30) >ppk.94 6003503 120 + 6181863 UAACCUUCGGGAGGACGGUUACCACGGUGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGACGACAUCAGCCUGCUGA ...((....))....((((((....((.(((((((((((((.......))))))........(((((((((....))))))))))))))))..)).))))))......((((....)))) ( -46.50) >psp.1833 3635134 119 - 5928787 UAACCUUCGGGGGGACGGUUACCACGGUGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGACGAC-UCAGCUGCACCA ...((....))((..((((((((.(((((((...))).)))).)))((.(((((...((((.(((((((((....)))))))))((....)).)))).....)))))-)))))))..)). ( -48.00) >pst.2031 3873122 119 - 6397126 UAACCUUCGGGGGGACGGUUACCACGGUGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGACGAC-UCAGCUGCACCA ...((....))((..((((((((.(((((((...))).)))).)))((.(((((...((((.(((((((((....)))))))))((....)).)))).....)))))-)))))))..)). ( -48.00) >pf5.3378 6386392 120 - 7074893 UAACCUUCGGGAGGACGGUUACCACGGUGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGACGACAUCAGCUGCUUCA ...((....)).(((((((((((.(((((((...))).)))).)))((.(((((...((((.(((((((((....)))))))))((....)).)))).....))))).))))))).))). ( -45.70) >consensus UAACCUUCGGGGGGACGGUUACCACGGUGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGCGGCUGGAUCACCUCCUUAAUCGACGAC_UCAGCUGCUUCA ...((....))....((((((....((.(((((((((((((.......))))))........(((((((((....))))))))))))))))))...)))))).................. (-42.97 = -42.97 + -0.00)

| Location | 6,043,226 – 6,043,346 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.89 |
| Mean single sequence MFE | -34.83 |
| Consensus MFE | -31.23 |
| Energy contribution | -31.90 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.90 |
| SVM decision value | 1.48 |
| SVM RNA-class probability | 0.957643 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043226 120 + 6264404 GCAGGUUCCCCUACGGCUACCUUGUUACGACUUCACCCCAGUCAUGAAUCACUCCGUGGUAACCGUCCCCCUUGCGGUUAGACUAGCUACUUCUGGAGCAACCCACUCCCAUGGUGUGAC ((((((..((....))..))).)))...((((.......)))).....((((.(((((((((((((.......))))))).....(((.(....).))).........)))))).)))). ( -33.50) >pau.3340 6316145 120 + 6537648 GCAGGUUCCCCUACGGCUACCUUGUUACGACUUCACCCCAGUCAUGAAUCACUCCGUGGUAACCGUCCCCCUUGCGGUUAGACUAGCUACUUCUGGAGCAACCCACUCCCAUGGUGUGAC ((((((..((....))..))).)))...((((.......)))).....((((.(((((((((((((.......))))))).....(((.(....).))).........)))))).)))). ( -33.50) >psp.1833 3635173 120 + 5928787 GCAGGUUCCCCUACGGCUACCUUGUUACGACUUCACCCCAGUCAUGAAUCACACCGUGGUAACCGUCCCCCCGAAGGUUAGACUAGCUACUUCUGGUGCAACCCACUCCCAUGGUGUGAC ((((((..((....))..))).)))...((((.......)))).....((((((((((((((((.((.....)).))))).(((((......)))))...........))))))))))). ( -35.50) >pst.2031 3873161 120 + 6397126 GCAGGUUCCCCUACGGCUACCUUGUUACGACUUCACCCCAGUCAUGAAUCACACCGUGGUAACCGUCCCCCCGAAGGUUAGACUAGCUACUUCUGGUGCAACCCACUCCCAUGGUGUGAC ((((((..((....))..))).)))...((((.......)))).....((((((((((((((((.((.....)).))))).(((((......)))))...........))))))))))). ( -35.50) >pfo.3002 5730041 120 + 6438405 GCAGGUUCCCCUACGGCUACCUUGUUACGACUUCACCCCAGUCAUGAAUCACACCGUGGUAACCGUCCUCCCGAAGGUUAGACUAGCUACUUCUGGUGCAACCCACUCCCAUGGUGUGAC ((((((..((....))..))).)))...((((.......)))).....((((((((((((((((.((.....)).))))).(((((......)))))...........))))))))))). ( -35.50) >pf5.3378 6386432 120 + 7074893 GCAGGUUCCCCUACGGCUACCUUGUUACGACUUCACCCCAGUCAUGAAUCACACCGUGGUAACCGUCCUCCCGAAGGUUAGACUAGCUACUUCUGGUGCAACCCACUCCCAUGGUGUGAC ((((((..((....))..))).)))...((((.......)))).....((((((((((((((((.((.....)).))))).(((((......)))))...........))))))))))). ( -35.50) >consensus GCAGGUUCCCCUACGGCUACCUUGUUACGACUUCACCCCAGUCAUGAAUCACACCGUGGUAACCGUCCCCCCGAAGGUUAGACUAGCUACUUCUGGUGCAACCCACUCCCAUGGUGUGAC ((((((..((....))..))).)))...((((.......)))).....((((((((((((((((...........))))).(((((......)))))...........))))))))))). (-31.23 = -31.90 + 0.67)

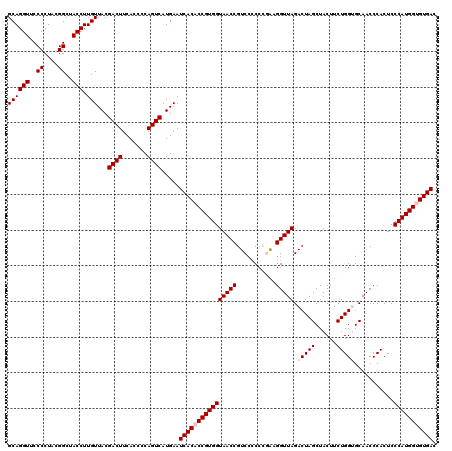
| Location | 6,043,226 – 6,043,346 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.89 |
| Mean single sequence MFE | -47.17 |
| Consensus MFE | -47.61 |
| Energy contribution | -47.17 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.51 |
| Structure conservation index | 1.01 |
| SVM decision value | 5.36 |
| SVM RNA-class probability | 0.999985 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043226 120 - 6264404 GUCACACCAUGGGAGUGGGUUGCUCCAGAAGUAGCUAGUCUAACCGCAAGGGGGACGGUUACCACGGAGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGC .((((.((.(((..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))..)))))))))..................(((((....))))) ( -47.80) >pau.3340 6316145 120 - 6537648 GUCACACCAUGGGAGUGGGUUGCUCCAGAAGUAGCUAGUCUAACCGCAAGGGGGACGGUUACCACGGAGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGC .((((.((.(((..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))..)))))))))..................(((((....))))) ( -47.80) >psp.1833 3635173 120 - 5928787 GUCACACCAUGGGAGUGGGUUGCACCAGAAGUAGCUAGUCUAACCUUCGGGGGGACGGUUACCACGGUGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGC .((((.((.(((..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))..)))))))))..................(((((....))))) ( -47.20) >pst.2031 3873161 120 - 6397126 GUCACACCAUGGGAGUGGGUUGCACCAGAAGUAGCUAGUCUAACCUUCGGGGGGACGGUUACCACGGUGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGC .((((.((.(((..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))..)))))))))..................(((((....))))) ( -47.20) >pfo.3002 5730041 120 - 6438405 GUCACACCAUGGGAGUGGGUUGCACCAGAAGUAGCUAGUCUAACCUUCGGGAGGACGGUUACCACGGUGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGC .((((.((.(((..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))..)))))))))..................(((((....))))) ( -46.50) >pf5.3378 6386432 120 - 7074893 GUCACACCAUGGGAGUGGGUUGCACCAGAAGUAGCUAGUCUAACCUUCGGGAGGACGGUUACCACGGUGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGC .((((.((.(((..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))..)))))))))..................(((((....))))) ( -46.50) >consensus GUCACACCAUGGGAGUGGGUUGCACCAGAAGUAGCUAGUCUAACCUUCGGGGGGACGGUUACCACGGUGUGAUUCAUGACUGGGGUGAAGUCGUAACAAGGUAGCCGUAGGGGAACCUGC .((((.((.(((..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))..)))))))))..................(((((....))))) (-47.61 = -47.17 + -0.44)


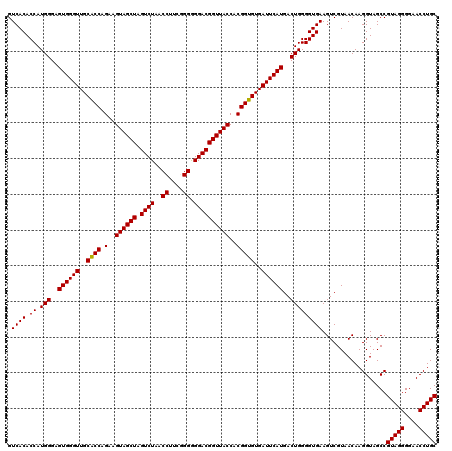
| Location | 6,043,266 – 6,043,386 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.44 |
| Mean single sequence MFE | -43.53 |
| Consensus MFE | -38.79 |
| Energy contribution | -39.23 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.20 |
| Structure conservation index | 0.89 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.588732 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043266 120 + 6264404 GUCAUGAAUCACUCCGUGGUAACCGUCCCCCUUGCGGUUAGACUAGCUACUUCUGGAGCAACCCACUCCCAUGGUGUGACGGGCGGUGUGUACAAGGCCCGGGAACGUAUUCACCGUGAC ((((((..((((.(((((((((((((.......))))))).....(((.(....).))).........)))))).))))(((((..((....))..))))).............)))))) ( -43.40) >pau.3340 6316185 120 + 6537648 GUCAUGAAUCACUCCGUGGUAACCGUCCCCCUUGCGGUUAGACUAGCUACUUCUGGAGCAACCCACUCCCAUGGUGUGACGGGCGGUGUGUACAAGGCCCGGGAACGUAUUCACCGUGAC ((((((..((((.(((((((((((((.......))))))).....(((.(....).))).........)))))).))))(((((..((....))..))))).............)))))) ( -43.40) >psp.1833 3635213 120 + 5928787 GUCAUGAAUCACACCGUGGUAACCGUCCCCCCGAAGGUUAGACUAGCUACUUCUGGUGCAACCCACUCCCAUGGUGUGACGGGCGGUGUGUACAAGGCCCGGGAACGUAUUCACCGCGAC (((.....((((((((((((((((.((.....)).))))).(((((......)))))...........)))))))))))...((((((.((((....(....)...)))).))))))))) ( -43.60) >pst.2031 3873201 120 + 6397126 GUCAUGAAUCACACCGUGGUAACCGUCCCCCCGAAGGUUAGACUAGCUACUUCUGGUGCAACCCACUCCCAUGGUGUGACGGGCGGUGUGUACAAGGCCCGGGAACGUAUUCACCGCGAC (((.....((((((((((((((((.((.....)).))))).(((((......)))))...........)))))))))))...((((((.((((....(....)...)))).))))))))) ( -43.60) >pfo.3002 5730081 120 + 6438405 GUCAUGAAUCACACCGUGGUAACCGUCCUCCCGAAGGUUAGACUAGCUACUUCUGGUGCAACCCACUCCCAUGGUGUGACGGGCGGUGUGUACAAGGCCCGGGAACGUAUUCACCGCGAC (((.....((((((((((((((((.((.....)).))))).(((((......)))))...........)))))))))))...((((((.((((....(....)...)))).))))))))) ( -43.60) >pf5.3378 6386472 120 + 7074893 GUCAUGAAUCACACCGUGGUAACCGUCCUCCCGAAGGUUAGACUAGCUACUUCUGGUGCAACCCACUCCCAUGGUGUGACGGGCGGUGUGUACAAGGCCCGGGAACGUAUUCACCGCGAC (((.....((((((((((((((((.((.....)).))))).(((((......)))))...........)))))))))))...((((((.((((....(....)...)))).))))))))) ( -43.60) >consensus GUCAUGAAUCACACCGUGGUAACCGUCCCCCCGAAGGUUAGACUAGCUACUUCUGGUGCAACCCACUCCCAUGGUGUGACGGGCGGUGUGUACAAGGCCCGGGAACGUAUUCACCGCGAC (((.....((((((((((((((((...........))))).(((((......)))))...........)))))))))))...((((((.((((....(....)...)))).))))))))) (-38.79 = -39.23 + 0.44)

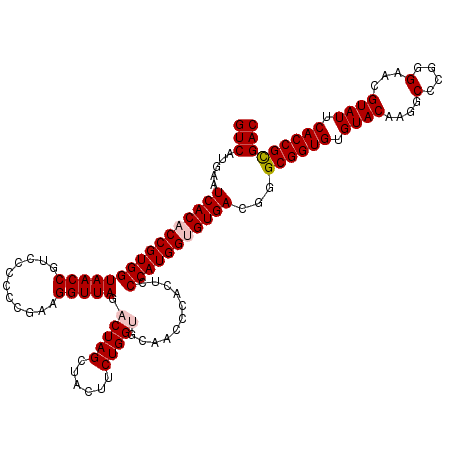
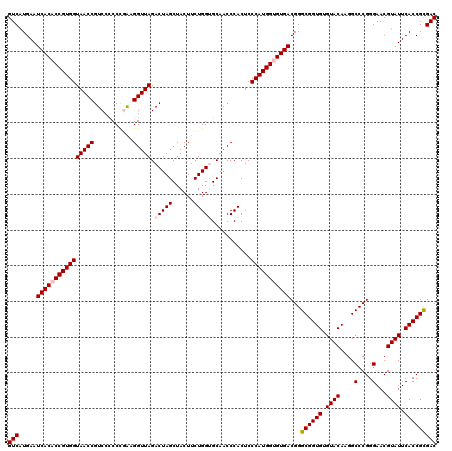
| Location | 6,043,266 – 6,043,386 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.44 |
| Mean single sequence MFE | -45.17 |
| Consensus MFE | -45.03 |
| Energy contribution | -44.37 |
| Covariance contribution | -0.66 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.06 |
| Structure conservation index | 1.00 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.964271 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043266 120 - 6264404 GUCACGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCCGUCACACCAUGGGAGUGGGUUGCUCCAGAAGUAGCUAGUCUAACCGCAAGGGGGACGGUUACCACGGAGUGAUUCAUGAC (((.(((((..((((..(....)...))))..)))))(((((......))))).((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))))) ( -45.40) >pau.3340 6316185 120 - 6537648 GUCACGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCCGUCACACCAUGGGAGUGGGUUGCUCCAGAAGUAGCUAGUCUAACCGCAAGGGGGACGGUUACCACGGAGUGAUUCAUGAC (((.(((((..((((..(....)...))))..)))))(((((......))))).((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))))) ( -45.40) >psp.1833 3635213 120 - 5928787 GUCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCCGUCACACCAUGGGAGUGGGUUGCACCAGAAGUAGCUAGUCUAACCUUCGGGGGGACGGUUACCACGGUGUGAUUCAUGAC (((((((((..((((..(....)...))))..))))))((((......))))..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))))) ( -45.40) >pst.2031 3873201 120 - 6397126 GUCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCCGUCACACCAUGGGAGUGGGUUGCACCAGAAGUAGCUAGUCUAACCUUCGGGGGGACGGUUACCACGGUGUGAUUCAUGAC (((((((((..((((..(....)...))))..))))))((((......))))..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))))) ( -45.40) >pfo.3002 5730081 120 - 6438405 GUCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCCGUCACACCAUGGGAGUGGGUUGCACCAGAAGUAGCUAGUCUAACCUUCGGGAGGACGGUUACCACGGUGUGAUUCAUGAC (((((((((..((((..(....)...))))..))))))((((......))))..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))))) ( -44.70) >pf5.3378 6386472 120 - 7074893 GUCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCCGUCACACCAUGGGAGUGGGUUGCACCAGAAGUAGCUAGUCUAACCUUCGGGAGGACGGUUACCACGGUGUGAUUCAUGAC (((((((((..((((..(....)...))))..))))))((((......))))..((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))))) ( -44.70) >consensus GUCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCCGUCACACCAUGGGAGUGGGUUGCACCAGAAGUAGCUAGUCUAACCUUCGGGGGGACGGUUACCACGGUGUGAUUCAUGAC ((((((.......)))((((((((..((....))..))))(......)..))))((((((..((((.(..((((((.((((..((....)).))))))))))..)))))..))))))))) (-45.03 = -44.37 + -0.66)

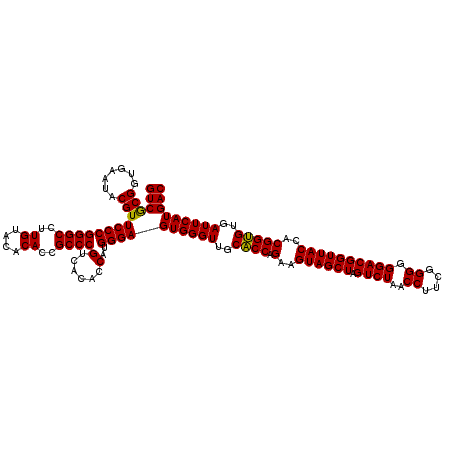
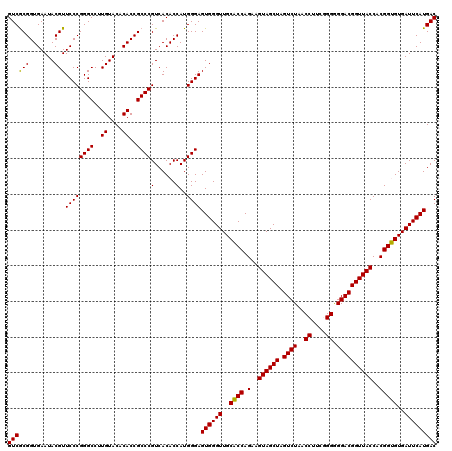
| Location | 6,043,346 – 6,043,466 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.67 |
| Mean single sequence MFE | -37.10 |
| Consensus MFE | -37.54 |
| Energy contribution | -37.10 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.06 |
| Mean z-score | -0.66 |
| Structure conservation index | 1.01 |
| SVM decision value | 0.40 |
| SVM RNA-class probability | 0.721938 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043346 120 - 6264404 AUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGUGAAUCAGAAUGUCACGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCC ........((.((((..((.....((((((........))))))((((..((.((.((((.(....).)))).)).))...))))))....)))).))..((((..((....))..)))) ( -36.10) >pau.3340 6316265 120 - 6537648 AUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGUGAAUCAGAAUGUCACGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCC ........((.((((..((.....((((((........))))))((((..((.((.((((.(....).)))).)).))...))))))....)))).))..((((..((....))..)))) ( -36.10) >psp.1833 3635293 120 - 5928787 ACAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGAAUCAGAAUGUCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCC ........((.((((..((.....((((((........))))))((((..((.((.((((.(....).)))).)).))...))))))....)))).))..((((..((....))..)))) ( -37.60) >pst.2031 3873281 120 - 6397126 ACAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGAAUCAGAAUGUCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCC ........((.((((..((.....((((((........))))))((((..((.((.((((.(....).)))).)).))...))))))....)))).))..((((..((....))..)))) ( -37.60) >pfo.3002 5730161 120 - 6438405 AUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGAAUCAGAAUGUCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCC ........((.((((..((.....((((((........))))))((((..((.((.((((.(....).)))).)).))...))))))....)))).))..((((..((....))..)))) ( -37.60) >pf5.3378 6386552 120 - 7074893 AUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGAAUCAGAAUGUCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCC ........((.((((..((.....((((((........))))))((((..((.((.((((.(....).)))).)).))...))))))....)))).))..((((..((....))..)))) ( -37.60) >consensus AUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGAAUCAGAAUGUCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCC ........((.((((..((.....((((((........))))))((((..((.((.((((.(....).)))).)).))...))))))....)))).))..((((..((....))..)))) (-37.54 = -37.10 + -0.44)

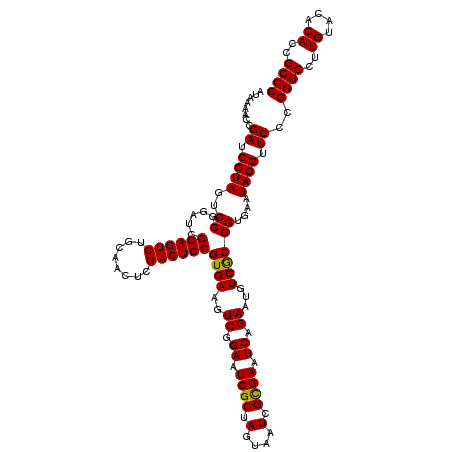
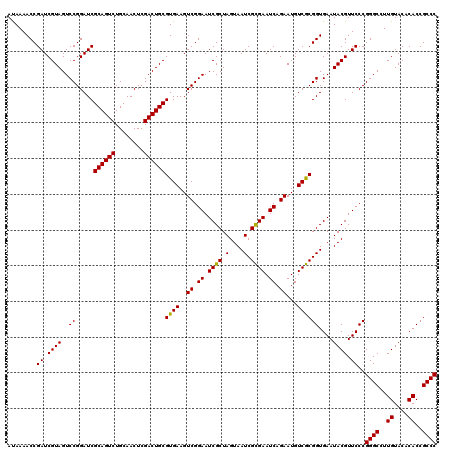
| Location | 6,043,386 – 6,043,506 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.22 |
| Mean single sequence MFE | -38.23 |
| Consensus MFE | -38.08 |
| Energy contribution | -37.63 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.01 |
| Structure conservation index | 1.00 |
| SVM decision value | 1.35 |
| SVM RNA-class probability | 0.945231 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043386 120 - 6264404 GGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGUGAAUCAGAAU (((.....(((..((...(((....)))...))....))).....)))(((.....(((((..(((((((........))))))).....))))).((((.(....).)))))))..... ( -37.20) >pau.3340 6316305 120 - 6537648 GGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGUGAAUCAGAAU (((.....(((..((...(((....)))...))....))).....)))(((.....(((((..(((((((........))))))).....))))).((((.(....).)))))))..... ( -37.20) >psp.1833 3635333 120 - 5928787 GGUACAGAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCUCACAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGAAUCAGAAU (((...((((...((...(((....)))...))...)))).....)))(((.....(((((..(((((((........))))))).....))))).((((.(....).)))))))..... ( -38.40) >pst.2031 3873321 120 - 6397126 GGUACAGAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCUCACAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGAAUCAGAAU (((...((((...((...(((....)))...))...)))).....)))(((.....(((((..(((((((........))))))).....))))).((((.(....).)))))))..... ( -38.40) >pfo.3002 5730201 120 - 6438405 GGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGAAUCAGAAU (((.....(((..((...(((....)))...))....))).....)))(((.....(((((..(((((((........))))))).....))))).((((.(....).)))))))..... ( -39.10) >pf5.3378 6386592 120 - 7074893 GGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGAAUCAGAAU (((.....(((..((...(((....)))...))....))).....)))(((.....(((((..(((((((........))))))).....))))).((((.(....).)))))))..... ( -39.10) >consensus GGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGACUGCGUGAAGUCGGAAUCGCUAGUAAUCGCGAAUCAGAAU (((.....(((((((...(((....)))...)).)))))......)))(((.....(((((..(((((((........))))))).....))))).((((.(....).)))))))..... (-38.08 = -37.63 + -0.44)

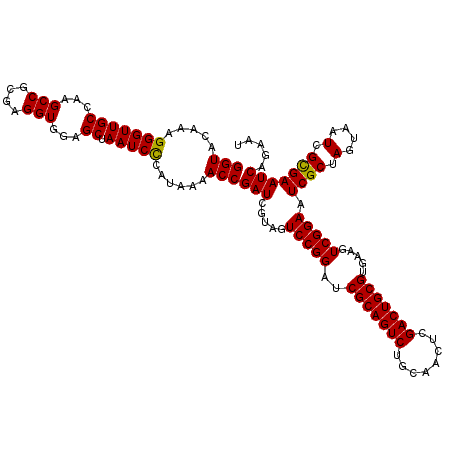
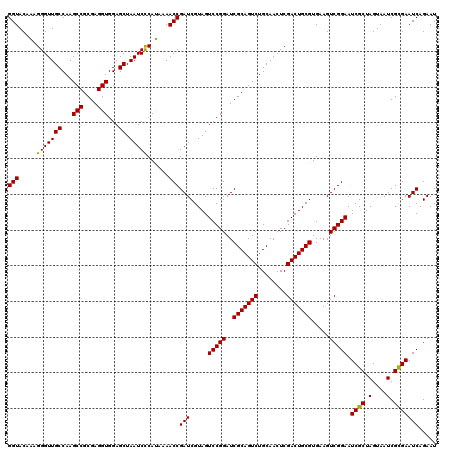
| Location | 6,043,426 – 6,043,546 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.22 |
| Mean single sequence MFE | -42.60 |
| Consensus MFE | -41.92 |
| Energy contribution | -41.70 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.08 |
| Structure conservation index | 0.98 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948688 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043426 120 - 6264404 UGGCCCUUACGGCCAGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGA (((((.....)))))(((((((....(.....)...(((((((.....(((..((...(((....)))...))....))).....))))))))))))))...(((.(((.....)))))) ( -43.70) >pau.3340 6316345 120 - 6537648 UGGCCCUUACGGCCAGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGA (((((.....)))))(((((((....(.....)...(((((((.....(((..((...(((....)))...))....))).....))))))))))))))...(((.(((.....)))))) ( -43.70) >psp.1833 3635373 120 - 5928787 UGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAGAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCUCACAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGA .((((.....))))((((((((....(.....)...(((((((...((((...((...(((....)))...))...)))).....)))))))))))))))..(((.(((.....)))))) ( -41.70) >pst.2031 3873361 120 - 6397126 UGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAGAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCUCACAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGA .((((.....))))((((((((....(.....)...(((((((...((((...((...(((....)))...))...)))).....)))))))))))))))..(((.(((.....)))))) ( -41.70) >pfo.3002 5730241 120 - 6438405 UGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGA .((((.....))))((((((((....(.....)...(((((((.....(((..((...(((....)))...))....))).....)))))))))))))))..(((.(((.....)))))) ( -42.40) >pf5.3378 6386632 120 - 7074893 UGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGA .((((.....))))((((((((....(.....)...(((((((.....(((..((...(((....)))...))....))).....)))))))))))))))..(((.(((.....)))))) ( -42.40) >consensus UGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUCGGUACAAAGGGUUGCCAAGCCGCGAGGUGGAGCUAAUCCCAUAAAACCGAUCGUAGUCCGGAUCGCAGUCUGCAACUCGA .((((.....)))).(((((((....(.....)...(((((((.....(((((((...(((....)))...)).)))))......))))))))))))))...(((.(((.....)))))) (-41.92 = -41.70 + -0.22)

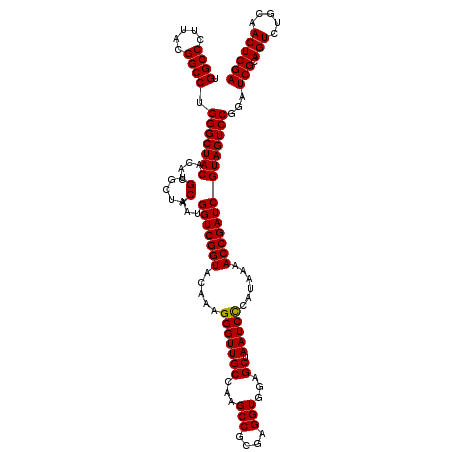
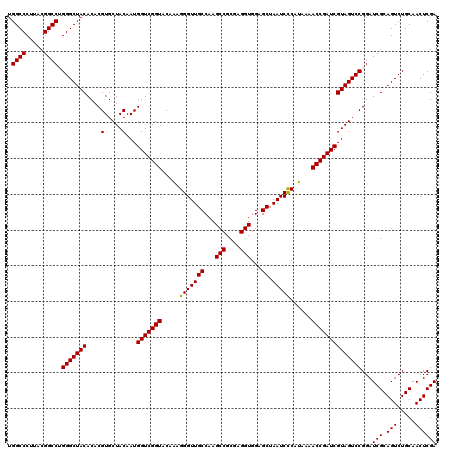
| Location | 6,043,506 – 6,043,625 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.44 |
| Mean single sequence MFE | -41.13 |
| Consensus MFE | -39.90 |
| Energy contribution | -40.23 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.12 |
| Structure conservation index | 0.97 |
| SVM decision value | 1.32 |
| SVM RNA-class probability | 0.941528 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043506 119 + 6264404 GACCAUUGUAGCACGUGUGUAGCCCUGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACC-CGAGGUGCUGGUA .((((.....(((((((...((((.(((((.....))))).((((((.....)))))).............))))...))).((((.(((....))).))))....-....)))))))). ( -39.80) >pau.3340 6316425 119 + 6537648 GACCAUUGUAGCACGUGUGUAGCCCUGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACC-CGAGGUGCUGGUA .((((.....(((((((...((((.(((((.....))))).((((((.....)))))).............))))...))).((((.(((....))).))))....-....)))))))). ( -39.80) >pss.1919 3851437 120 + 6093698 GACCAUUGUAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUGACGUGCUGGUA .((((.....(((((((((.((((..((((.....))))..((((((.....)))))).............))))...))).((((.(((....))).)))).......)))))))))). ( -41.90) >pst.2031 3873441 120 + 6397126 GACCAUUGUAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCUUAACGUGCUGGUA .((((.....(((((((((.((((..((((.....))))..((((((.....)))))).............))))...))).((((.(((....))).)))).......)))))))))). ( -41.80) >pfo.3002 5730321 120 + 6438405 GACCAUUGUAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUUACGUGCUGGUA .((((.....(((((((((.((((..((((.....))))..((((((.....)))))).............))))...))).((((.(((....))).)))).......)))))))))). ( -41.70) >pf5.3378 6386712 120 + 7074893 GACCAUUGUAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUA .((((.....(((((((((.((((..((((.....))))..((((((.....)))))).............))))...))).((((.(((....))).)))).......)))))))))). ( -41.80) >consensus GACCAUUGUAGCACGUGUGUAGCCCAGGCCGUAAGGGCCAUGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUGACGUGCUGGUA .((((.....(((((((((.((((..((((.....))))..((((((.....)))))).............))))...))).((((.(((....))).)))).......)))))))))). (-39.90 = -40.23 + 0.33)
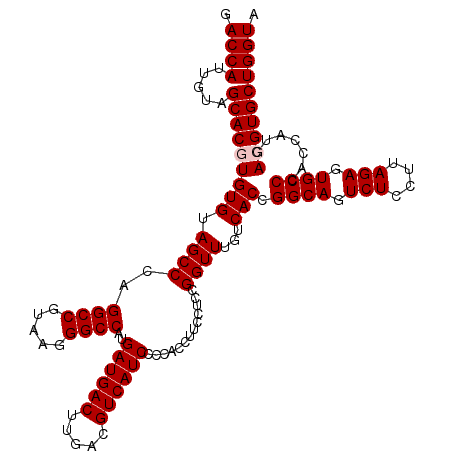
| Location | 6,043,506 – 6,043,625 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.44 |
| Mean single sequence MFE | -45.93 |
| Consensus MFE | -45.23 |
| Energy contribution | -45.23 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.64 |
| Structure conservation index | 0.98 |
| SVM decision value | 3.04 |
| SVM RNA-class probability | 0.998228 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043506 119 - 6264404 UACCAGCACCUCG-GGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCAGGGCUACACACGUGCUACAAUGGUC .((((...((((.-(((.((((.(((....))).))))(....)..))).))))...((((.((((((.....)))))).(((((((.......)))))))........))))..)))). ( -47.80) >pau.3340 6316425 119 - 6537648 UACCAGCACCUCG-GGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCAGGGCUACACACGUGCUACAAUGGUC .((((...((((.-(((.((((.(((....))).))))(....)..))).))))...((((.((((((.....)))))).(((((((.......)))))))........))))..)))). ( -47.80) >pss.1919 3851437 120 - 6093698 UACCAGCACGUCAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUC .((((.(((......)))((((((((.((....)).(((((.....))))).)))..(((..((((((.....)))))).((((((.........)))))))))..)))))....)))). ( -44.90) >pst.2031 3873441 120 - 6397126 UACCAGCACGUUAAGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUC .((((.(((......)))((((((((.((....)).(((((.....))))).)))..(((..((((((.....)))))).((((((.........)))))))))..)))))....)))). ( -44.90) >pfo.3002 5730321 120 - 6438405 UACCAGCACGUAAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUC .((((((((((..((((.((((.(((....))).))))(....)..)))).......(((..((((((.....)))))).((((((.........))))))))))))))).....)))). ( -45.30) >pf5.3378 6386712 120 - 7074893 UACCAGCACGUUAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUC .((((.(((......)))((((((((.((....)).(((((.....))))).)))..(((..((((((.....)))))).((((((.........)))))))))..)))))....)))). ( -44.90) >consensus UACCAGCACGUCAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUACGGCCUGGGCUACACACGUGCUACAAUGGUC .((((.(((......)))((((((((.((....)).(((((.....))))).)))..(((..((((((.....)))))).((((((.........)))))))))..)))))....)))). (-45.23 = -45.23 + -0.00)

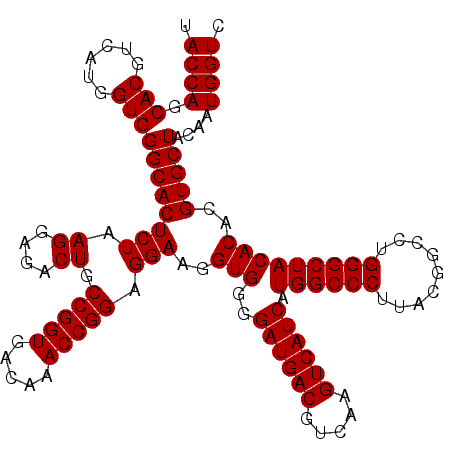
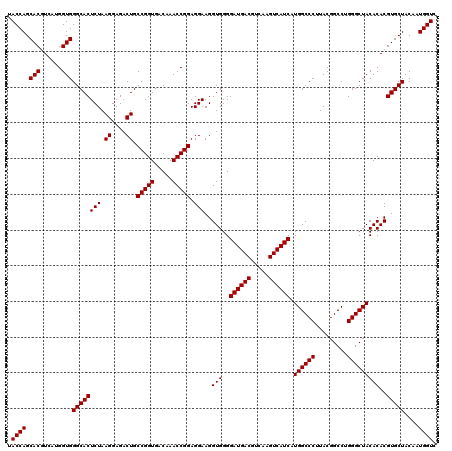
| Location | 6,043,546 – 6,043,665 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.89 |
| Mean single sequence MFE | -40.32 |
| Consensus MFE | -39.77 |
| Energy contribution | -39.77 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.99 |
| SVM decision value | 3.27 |
| SVM RNA-class probability | 0.998900 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043546 119 + 6264404 UGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACC-CGAGGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCA .((((((.....)))))).........(((((((.....))))).))..(((((((..((((((((-...))))..)))).)))))))...((((((.(((((...)))).).)))))). ( -41.20) >pau.3340 6316465 119 + 6537648 UGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACC-CGAGGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCA .((((((.....)))))).........(((((((.....))))).))..(((((((..((((((((-...))))..)))).)))))))...((((((.(((((...)))).).)))))). ( -41.20) >pss.1919 3851477 120 + 6093698 UGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUGACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCA .((((((.....))))))....((((...(((((.(((((..((((.(((....))).)))).....))))).))))).....))))....((((((.(((((...)))).).)))))). ( -42.20) >pst.2031 3873481 120 + 6397126 UGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCUUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCA .((((((.....)))))).........(((((((.....))))).))..(((((((..(((((((......)))..)))).)))))))...((((((.(((((...)))).).)))))). ( -39.10) >pfo.3002 5730361 120 + 6438405 UGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUUACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCA .((((((.....)))))).........(((((((.....))))).))..(((((((..(((((((......)))..)))).)))))))...((((((.(((((...)))).).)))))). ( -39.10) >pf5.3378 6386752 120 + 7074893 UGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCA .((((((.....)))))).........(((((((.....))))).))..(((((((..(((((((......)))..)))).)))))))...((((((.(((((...)))).).)))))). ( -39.10) >consensus UGAUGACUUGACGUCAUCCCCACCUUCCUCCGGUUUGUCACCGGCAGUCUCCUUAGAGUGCCCACCAUGACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCA .((((((.....)))))).........(((((((.....))))).))..(((((((..(((((((......)))..)))).)))))))...((((((.(((((...)))).).)))))). (-39.77 = -39.77 + 0.00)

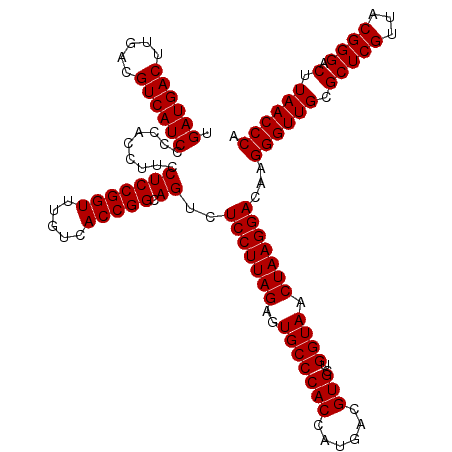
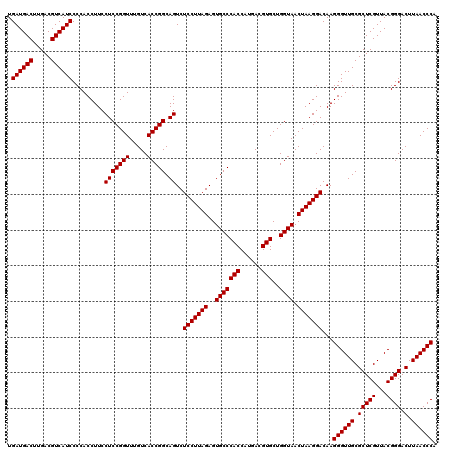
| Location | 6,043,546 – 6,043,665 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.89 |
| Mean single sequence MFE | -42.67 |
| Consensus MFE | -42.63 |
| Energy contribution | -42.63 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.30 |
| Structure conservation index | 1.00 |
| SVM decision value | 2.32 |
| SVM RNA-class probability | 0.992267 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043546 119 - 6264404 UGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACCUCG-GGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCA .(((......))).............(((...(((((.((((((..((((...-))))((((.(((....))).))))))))))......)))))....)))((((((.....)))))). ( -43.60) >pau.3340 6316465 119 - 6537648 UGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACCUCG-GGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCA .(((......))).............(((...(((((.((((((..((((...-))))((((.(((....))).))))))))))......)))))....)))((((((.....)))))). ( -43.60) >pss.1919 3851477 120 - 6093698 UGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUCAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCA .(((......))).............(((...(((((.((((((..(((......)))((((.(((....))).))))))))))......)))))....)))((((((.....)))))). ( -42.20) >pst.2031 3873481 120 - 6397126 UGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUUAAGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCA .(((......))).............(((...(((((.((((((..(((......)))((((.(((....))).))))))))))......)))))....)))((((((.....)))))). ( -42.20) >pfo.3002 5730361 120 - 6438405 UGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUAAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCA .(((......))).............(((...(((((.((((((..(((......)))((((.(((....))).))))))))))......)))))....)))((((((.....)))))). ( -42.20) >pf5.3378 6386752 120 - 7074893 UGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUUAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCA .(((......))).............(((...(((((.((((((..(((......)))((((.(((....))).))))))))))......)))))....)))((((((.....)))))). ( -42.20) >consensus UGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGUUACCAGCACGUCAUGGUGGGCACUCUAAGGAGACUGCCGGUGACAAACCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCA .(((......))).............(((...(((((.((((((..(((......)))((((.(((....))).))))))))))......)))))....)))((((((.....)))))). (-42.63 = -42.63 + -0.00)

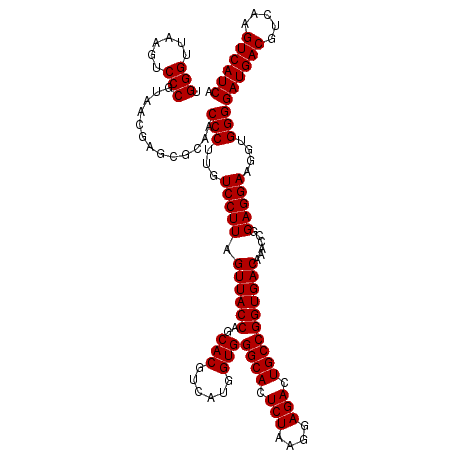
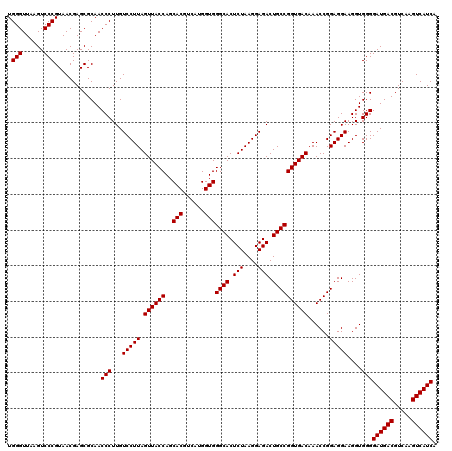
| Location | 6,043,586 – 6,043,705 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.89 |
| Mean single sequence MFE | -37.67 |
| Consensus MFE | -37.17 |
| Energy contribution | -37.17 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -0.87 |
| Structure conservation index | 0.99 |
| SVM decision value | 0.80 |
| SVM RNA-class probability | 0.854680 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043586 119 + 6264404 CCGGCAGUCUCCUUAGAGUGCCCACC-CGAGGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCU ..((((.(((....))).))))....-..((((((((((((((.........))))))((((((...(((......)))....((....)).))))))..............)))))))) ( -40.00) >pau.3340 6316505 119 + 6537648 CCGGCAGUCUCCUUAGAGUGCCCACC-CGAGGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCU ..((((.(((....))).))))....-..((((((((((((((.........))))))((((((...(((......)))....((....)).))))))..............)))))))) ( -40.00) >pss.1919 3851517 120 + 6093698 CCGGCAGUCUCCUUAGAGUGCCCACCAUGACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCU ..(((.((((((((((..(((((((......)))..)))).))))))).....((((.((((((...(((......)))....((....)).))))))))))))).)))........... ( -36.50) >pst.2031 3873521 120 + 6397126 CCGGCAGUCUCCUUAGAGUGCCCACCUUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCU ..(((.((((((((((..(((((((......)))..)))).))))))).....((((.((((((...(((......)))....((....)).))))))))))))).)))........... ( -36.50) >pfo.3002 5730401 120 + 6438405 CCGGCAGUCUCCUUAGAGUGCCCACCAUUACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCU ..(((.((((((((((..(((((((......)))..)))).))))))).....((((.((((((...(((......)))....((....)).))))))))))))).)))........... ( -36.50) >pf5.3378 6386792 120 + 7074893 CCGGCAGUCUCCUUAGAGUGCCCACCAUAACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCU ..(((.((((((((((..(((((((......)))..)))).))))))).....((((.((((((...(((......)))....((....)).))))))))))))).)))........... ( -36.50) >consensus CCGGCAGUCUCCUUAGAGUGCCCACCAUGACGUGCUGGUAACUAAGGACAAGGGUUGCGCUCGUUACGGGACUUAACCCAACAUCUCACGACACGAGCUGACGACAGCCAUGCAGCACCU ..(((.((((((((((..(((((((......)))..)))).))))))).....((((.((((((...(((......)))....((....)).))))))))))))).)))........... (-37.17 = -37.17 + -0.00)

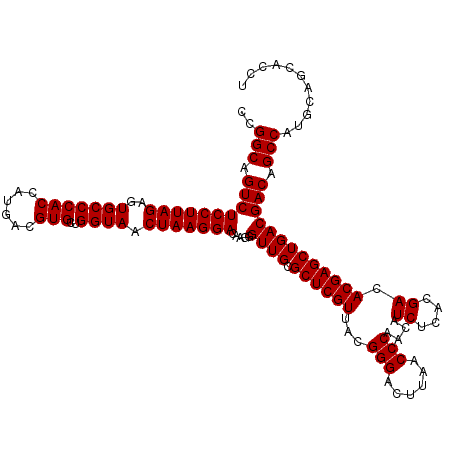
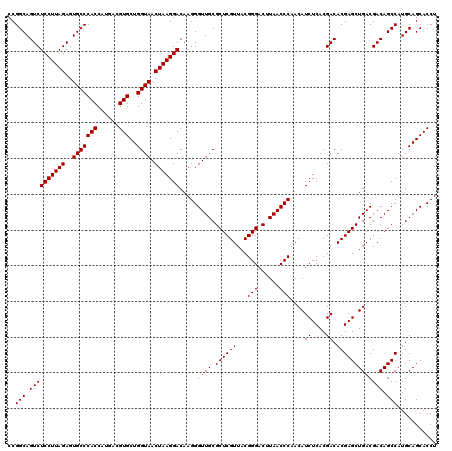
| Location | 6,043,625 – 6,043,745 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.94 |
| Mean single sequence MFE | -42.95 |
| Consensus MFE | -38.30 |
| Energy contribution | -37.30 |
| Covariance contribution | -1.00 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.15 |
| Structure conservation index | 0.89 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.541157 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.3201 6043625 120 - 6264404 CUUUCCAGAGAUGGAUUGGUGCCUUCGGGAACUCAGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGU (..((((....))))..)((.((....)).))...((((.(((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).))))))))...... ( -41.80) >pau.3340 6316544 120 - 6537648 CUUUCCAGAGAUGGAUUGGUGCCUUCGGGAACUCAGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGU (..((((....))))..)((.((....)).))...((((.(((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).))))))))...... ( -41.80) >ppk.94 6003943 120 + 6181863 CUUUCCAGAGAUGGAUUGGUGCCUUCGGGAACUCUGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGU (..((((....))))..)((.((....)).))...((((.(((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).))))))))...... ( -41.60) >pst.2031 3873561 120 - 6397126 UCCUUUAGAGAUAGAGGAGUGCCUUCGGGAGCAUUGAGACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGU (((((((....)))))))((.((....)).))(((((((((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).))))).)))))) ( -46.70) >pfo.3002 5730441 120 - 6438405 CUUUCCAGAGAUGGAUUGGUGCCUUCGGGAACAUUGAGACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGU (..((((....))))..)((.((....)).))(((((((((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).))))).)))))) ( -43.90) >pf5.3378 6386832 120 - 7074893 CUUUCUAGAGAUAGAUUGGUGCCUUCGGGAACAUUGAGACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGU (..((((....))))..)((.((....)).))(((((((((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).))))).)))))) ( -41.90) >consensus CUUUCCAGAGAUGGAUUGGUGCCUUCGGGAACACUGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUUGGGUUAAGUCCCGUAACGAGCGCAACCCUUGUCCUUAGU (..((((....))))..)((.((....)).))......(((((.(.(((.((((....))))(((((((((....)))...(((......)))...))))))))).).)))))....... (-38.30 = -37.30 + -1.00)

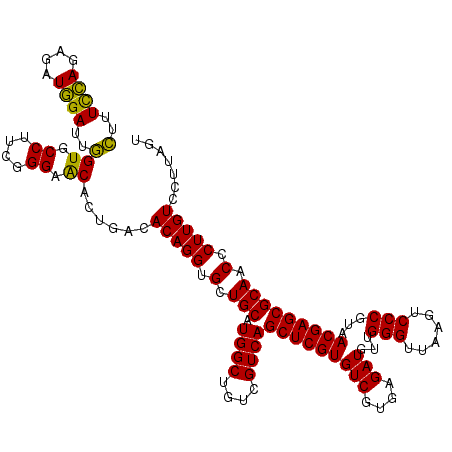
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:04:48 2007