
| Sequence ID | pao.1872 |
|---|---|
| Location | 3,524,006 – 3,524,125 |
| Length | 119 |
| Max. P | 0.982397 |

| Location | 3,524,006 – 3,524,115 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.77 |
| Mean single sequence MFE | -40.68 |
| Consensus MFE | -36.66 |
| Energy contribution | -33.95 |
| Covariance contribution | -2.71 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.90 |
| SVM decision value | 1.17 |
| SVM RNA-class probability | 0.925775 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1872 3524006 109 + 6264404 C-GCUGUUCCGCGAUAGCUCAGUCGGUAGAGCAAAUGACUGUUAAUCAUUGGGUCCCUGGUUCGAGUCCAGGUCGCGGAGCCAAAU---UCA------AAGCCCUCGGCACAUGCCG-GG .-((((((((((((..(((((((.(((..((((......)))).)))))))))).(((((.......)))))))))))))).....---...------.))).((((((....))))-)) ( -41.40) >pel.3003 5540594 116 + 5888780 C-CCUGUGCCGGUAUAGCUCAGAUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGAUUCCUGGUGCCGGCACCAUACGUAGUA-CUCAAGAGGC-UCACCGAAAGGUG-GG .-((((((((((((..((((........))))....((((((.......))))))(((((.......))))))))))))))..(((...)))-......)))(-(((((....))))-)) ( -43.40) >ppk.159 5846811 117 - 6181863 C-CCAUUGCCGGUAUAGCUCAGAUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGAUUCCUGGUGCCGGCACCAUACGCAGUAUCAAAAAAGGC-UCACCGAAAGGUG-GG .-((..((((((((..((((........))))....((((((.......))))))(((((.......)))))))))))))..((((...)))).......))(-(((((....))))-)) ( -40.50) >pf5.198 6700389 114 - 7074893 CUGACACGCCGGUAUAGCUCAGUUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGACUCCUGGUGCCGGCACCAUACAAAACA------AAGCCCUCGCAGCGAUGCGAGG .......((((((((((.(((((((......))))))).)))).)))....((..(((((.......)))))..))))).............------....(((((((....))))))) ( -41.90) >pfo.169 6085294 112 - 6438405 C--ACACGCCGGUAUAGCUCAGAUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGAUUCCUGGUGCCGGCACCAUACAAAACA------AAGCCCUUGCAGAAAUGCAGGG .--....(((((((..((((........))))....((((((.......))))))(((((.......)))))))))))).............------....(((((((....))))))) ( -40.50) >pst.3197 6097135 111 + 6397126 C-AUUACGCCGGUAUAGCUCAGUUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGACUCCUGGUGCCGGCACCAUAUAAAUCA------AGGGC-UUGCAGCGAUGCA-GG .-.....((((((((((.(((((((......))))))).)))).)))....((..(((((.......)))))..))))).............------....(-(((((....))))-)) ( -36.40) >consensus C_CCUACGCCGGUAUAGCUCAGAUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGAUUCCUGGUGCCGGCACCAUACAAAACA______AAGCC_UCGCAGAAAGGCG_GG .......(((((((..((((........)))).(((((.......))))).....(((((.......))))))))))))..........................((((....))))... (-36.66 = -33.95 + -2.71)

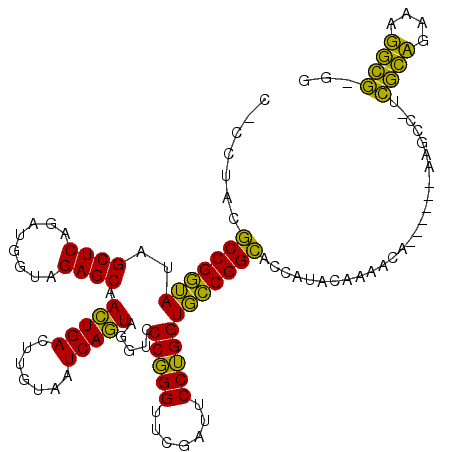
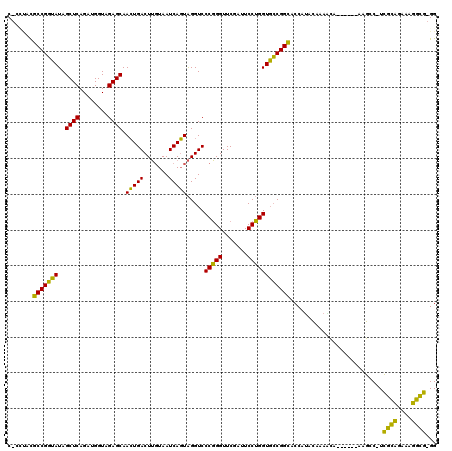
| Location | 3,524,006 – 3,524,115 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 77.77 |
| Mean single sequence MFE | -36.65 |
| Consensus MFE | -30.14 |
| Energy contribution | -27.65 |
| Covariance contribution | -2.49 |
| Combinations/Pair | 1.36 |
| Mean z-score | -0.84 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.584790 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1872 3524006 109 - 6264404 CC-CGGCAUGUGCCGAGGGCUU------UGA---AUUUGGCUCCGCGACCUGGACUCGAACCAGGGACCCAAUGAUUAACAGUCAUUUGCUCUACCGACUGAGCUAUCGCGGAACAGC-G ((-((((....)))).))(((.------...---....)))((((((((((((.......))))).....((((((.....)))))).((((........))))..))))))).....-. ( -39.40) >pel.3003 5540594 116 - 5888780 CC-CACCUUUCGGUGA-GCCUCUUGAG-UACUACGUAUGGUGCCGGCACCAGGAAUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAUCUGAGCUAUACCGGCACAGG-G ..-((((....)))).-.(((.....(-(((...)))).(((((((....(((..(((....)))..)))((((((.....)))))).((((........))))....))))))))))-. ( -36.30) >ppk.159 5846811 117 + 6181863 CC-CACCUUUCGGUGA-GCCUUUUUUGAUACUGCGUAUGGUGCCGGCACCAGGAAUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAUCUGAGCUAUACCGGCAAUGG-G ((-((...((((((..-.(((......((((...))))((((....))))))).)))))).((((.....((((((.....)))))).((((........))))....))))...)))-) ( -35.70) >pf5.198 6700389 114 + 7074893 CCUCGCAUCGCUGCGAGGGCUU------UGUUUUGUAUGGUGCCGGCACCAGGAGUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAACUGAGCUAUACCGGCGUGUCAG .((((((....))))))(((((------(........(((((....)))))))))))((((((((.....((((((.....)))))).((((........))))....)))).)).)).. ( -39.00) >pfo.169 6085294 112 + 6438405 CCCUGCAUUUCUGCAAGGGCUU------UGUUUUGUAUGGUGCCGGCACCAGGAAUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAUCUGAGCUAUACCGGCGUGU--G (((((((....)))).(((.((------((.(((...(((((....))))).))).))))))))))..........((((.(((.((.((((........))))...)).))).)))--) ( -34.50) >pst.3197 6097135 111 - 6397126 CC-UGCAUCGCUGCAA-GCCCU------UGAUUUAUAUGGUGCCGGCACCAGGAGUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAACUGAGCUAUACCGGCGUAAU-G ..-((((....)))).-....(------((((((...(((((....))))).)))))))((((((.....((((((.....)))))).((((........))))....)))).))...-. ( -35.00) >consensus CC_CGCAUUGCGGCGA_GCCUU______UGAUUUGUAUGGUGCCGGCACCAGGAAUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAACUGAGCUAUACCGGCAUAGC_G ...((((....))))........................(((((((..((.((.......)).)).....((((((.....)))))).((((........))))....)))))))..... (-30.14 = -27.65 + -2.49)

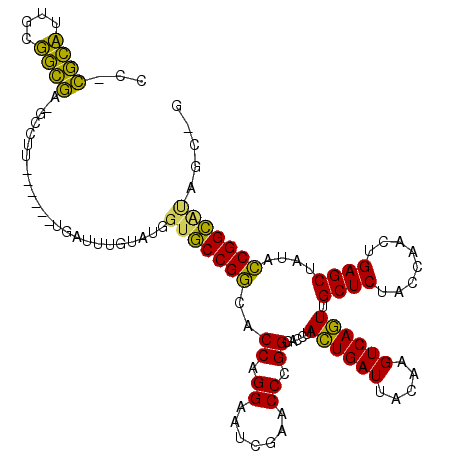
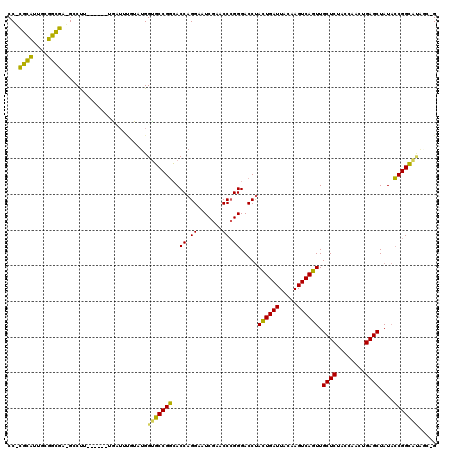
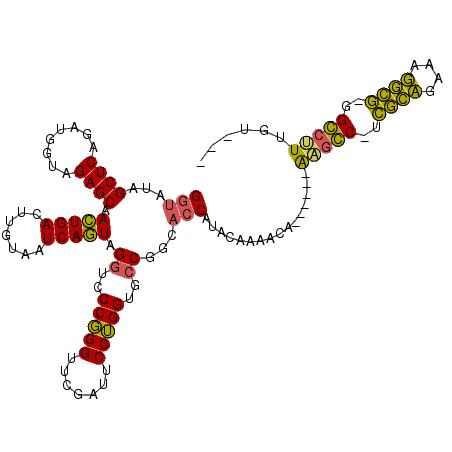
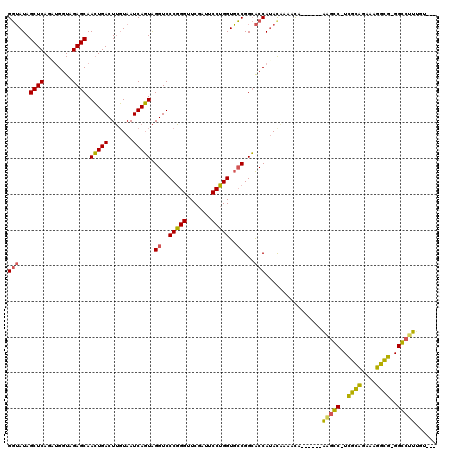
| Location | 3,524,015 – 3,524,125 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.33 |
| Mean single sequence MFE | -48.85 |
| Consensus MFE | -34.88 |
| Energy contribution | -32.97 |
| Covariance contribution | -1.91 |
| Combinations/Pair | 1.32 |
| Mean z-score | -4.18 |
| Structure conservation index | 0.71 |
| SVM decision value | 1.91 |
| SVM RNA-class probability | 0.982397 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1872 3524015 110 + 6264404 GCGAUAGCUCAGUCGGUAGAGCAAAUGACUGUUAAUCAUUGGGUCCCUGGUUCGAGUCCAGGUCGCGGAGCCAAAU---UCA------AAGCCCUCGGCACAUGCCG-GGGGCUUUUUCG ((((..(((((((.(((..((((......)))).)))))))))).(((((.......)))))))))(((......)---))(------(((((((((((....))))-)))))))).... ( -44.60) >pel.3003 5540603 114 + 5888780 GGUAUAGCUCAGAUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGAUUCCUGGUGCCGGCACCAUACGUAGUA-CUCAAGAGGC-UCACCGAAAGGUG-GGCCUUUUU--- (((((.((....(((((...((.(((((.......))))).((..(((((.......)))))..)).)))))))..)).)))-)).(((((((-(((((....))))-)))))))).--- ( -48.90) >ppk.159 5846820 115 - 6181863 GGUAUAGCUCAGAUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGAUUCCUGGUGCCGGCACCAUACGCAGUAUCAAAAAAGGC-UCACCGAAAGGUG-GGCCUUUUU--- (((((.((....(((((...((.(((((.......))))).((..(((((.......)))))..)).)))))))..)).))))).((((((((-(((((....))))-)))))))))--- ( -51.80) >pf5.198 6700399 111 - 7074893 GGUAUAGCUCAGUUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGACUCCUGGUGCCGGCACCAUACAAAACA------AAGCCCUCGCAGCGAUGCGAGGGCUUUGU--- (((((((.(((((((......))))))).)))).....((.((..(((((.......)))))..)).))))).......(((------(((((((((((....))))))))))))))--- ( -52.70) >pfo.169 6085302 111 - 6438405 GGUAUAGCUCAGAUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGAUUCCUGGUGCCGGCACCAUACAAAACA------AAGCCCUUGCAGAAAUGCAGGGGCUUUGU--- (((...((((........)))).(((((.......))))).((..(((((.......)))))..))...))).......(((------(((((((((((....))))))))))))))--- ( -50.50) >pst.3197 6097144 109 + 6397126 GGUAUAGCUCAGUUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGACUCCUGGUGCCGGCACCAUAUAAAUCA------AGGGC-UUGCAGCGAUGCA-GGCCCUCGU--- (((((((.(((((((......))))))).)))).....((.((..(((((.......)))))..)).)))))..........------(((((-(((((....))))-))))))...--- ( -44.60) >consensus GGUAUAGCUCAGAUGGUAGAGCAACUGACUUGUAAUCAGUAGGUCCCGGGUUCGAUUCCUGGUGCCGGCACCAUACAAAACA______AAGCC_UCGCAGAAAGGCG_GGCCUUUGU___ (((...((((........)))).(((((.......))))).((..(((((.......)))))..))...)))................(((((..((((....))))..)))))...... (-34.88 = -32.97 + -1.91)

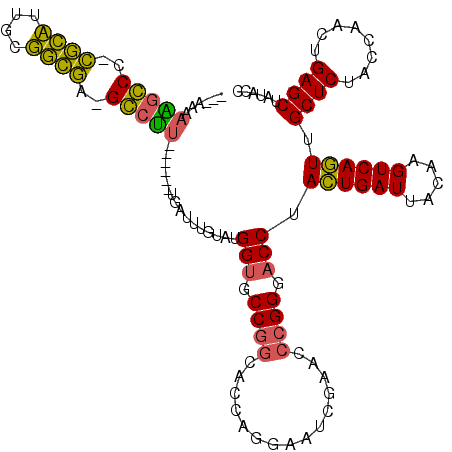
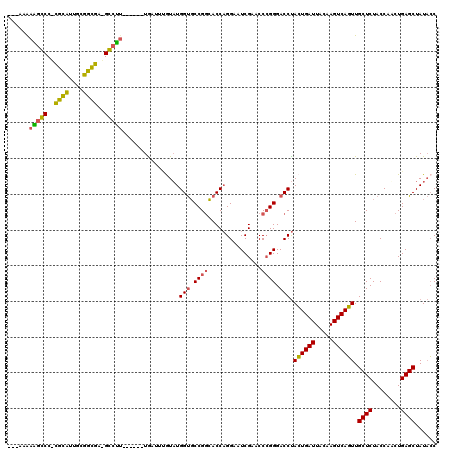
| Location | 3,524,015 – 3,524,125 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.33 |
| Mean single sequence MFE | -39.93 |
| Consensus MFE | -29.76 |
| Energy contribution | -27.79 |
| Covariance contribution | -1.97 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.74 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.81 |
| SVM RNA-class probability | 0.978379 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1872 3524015 110 - 6264404 CGAAAAAGCCCC-CGGCAUGUGCCGAGGGCUU------UGA---AUUUGGCUCCGCGACCUGGACUCGAACCAGGGACCCAAUGAUUAACAGUCAUUUGCUCUACCGACUGAGCUAUCGC (((.(((((((.-((((....)))).))))))------)..---...((((((.(((.(((((.......)))))((.(.((((((.....)))))).).))...)).).))))))))). ( -42.40) >pel.3003 5540603 114 - 5888780 ---AAAAAGGCC-CACCUUUCGGUGA-GCCUCUUGAG-UACUACGUAUGGUGCCGGCACCAGGAAUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAUCUGAGCUAUACC ---....((((.-((((....)))).-))))......-......((((((((....))))(((..(((....)))..)))((((((.....)))))).((((........)))).)))). ( -33.90) >ppk.159 5846820 115 + 6181863 ---AAAAAGGCC-CACCUUUCGGUGA-GCCUUUUUUGAUACUGCGUAUGGUGCCGGCACCAGGAAUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAUCUGAGCUAUACC ---((((((((.-((((....)))).-)))))))).........((((((((....))))(((..(((....)))..)))((((((.....)))))).((((........)))).)))). ( -37.20) >pf5.198 6700399 111 + 7074893 ---ACAAAGCCCUCGCAUCGCUGCGAGGGCUU------UGUUUUGUAUGGUGCCGGCACCAGGAGUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAACUGAGCUAUACC ---((((((((((((((....)))))))))))------)))...(((((((.((((.....(....)....)))).))).((((((.....)))))).((((........)))).)))). ( -46.70) >pfo.169 6085302 111 + 6438405 ---ACAAAGCCCCUGCAUUUCUGCAAGGGCUU------UGUUUUGUAUGGUGCCGGCACCAGGAAUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAUCUGAGCUAUACC ---(((((((((.((((....)))).))))))------)))...((((((((....))))(((..(((....)))..)))((((((.....)))))).((((........)))).)))). ( -40.80) >pst.3197 6097144 109 - 6397126 ---ACGAGGGCC-UGCAUCGCUGCAA-GCCCU------UGAUUUAUAUGGUGCCGGCACCAGGAGUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAACUGAGCUAUACC ---.(((((((.-((((....)))).-)))))------))........(((.((((.....(....)....)))).))).((((((.....)))))).((((........))))...... ( -38.60) >consensus ___AAAAAGCCC_CGCAUUGCGGCGA_GCCUU______UGAUUUGUAUGGUGCCGGCACCAGGAAUCGAACCCGGGACCUACUGAUUACAAGUCAGUUGCUCUACCAACUGAGCUAUACC ......(((((..((((....))))..)))))................(((.((((...............)))).))).((((((.....)))))).((((........))))...... (-29.76 = -27.79 + -1.97)

Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:13:14 2007