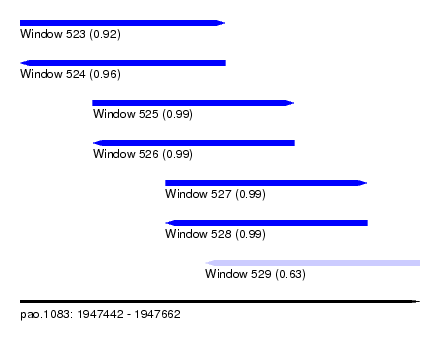
| Sequence ID | pao.1083 |
|---|---|
| Location | 1,947,442 – 1,947,662 |
| Length | 220 |
| Max. P | 0.993560 |

| Location | 1,947,442 – 1,947,555 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.91 |
| Mean single sequence MFE | -47.90 |
| Consensus MFE | -42.61 |
| Energy contribution | -41.17 |
| Covariance contribution | -1.44 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.16 |
| SVM RNA-class probability | 0.924314 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
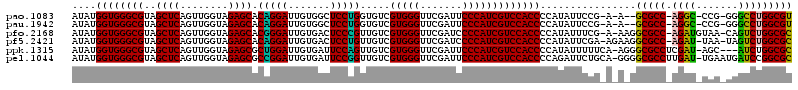
>pao.1083 1947442 113 + 6264404 AUAUGGUGGGCGUAGCUCAGUUGGUAGAGCACAGGAUUGUGGCUCCUGGUGUCGUGGGUUCGAUUCCCAUCGUCCACCCCAUAUUCCG-A-A--GCGCC-AGGC-CCG-GGGCCUGGCGU ....((((((((..((((........)))).(((((.......))))).....(((((.......)))))))))))))..........-.-.--(((((-((((-(..-.)))))))))) ( -51.10) >pau.1942 2850415 113 - 6537648 AUAUGGUGGGCGUAGCUCAGUUGGUAGAGCACAGGAUUGUGGCUCCUGGUGUCGUGGGUUCGAUUCCCAUCGUCCACCCCAUAUUCCG-A-A--GCGCC-AGGC-CCG-GGGCCUGGCGU ....((((((((..((((........)))).(((((.......))))).....(((((.......)))))))))))))..........-.-.--(((((-((((-(..-.)))))))))) ( -51.10) >pfo.2168 2249823 116 - 6438405 AUAUGGUGGGCGUAGCUCAGUUGGUAGAGCACGGGAUUGUGACUCCCGUUGUCGUGGGUUCGAUCCCCAUCGUCCACCCCAUAUUUCG-A-AAGGCGCC-AGAUGUAA-CAGUCUGGCGC ....((((((((..((((........))))..(((((((.(((((.((....)).))))))))))))...))))))))........(.-.-..)(((((-((((....-..))))))))) ( -53.30) >pf5.2421 2466463 116 - 7074893 AUAUGGUGGGCGUAGCUCAGUUGGUAGAGCACAGGAUUGUGACUCCUGUUGUCGUGGGUUCGAUCCCCAUCGUCCACCCCAUAUUCGA-AGAAGGCGCC-AGAU-UAA-UAGUCUGGCGC ....((((((((..((((........))))...((((((.(((((.((....)).)))))))))))....))))))))..........-.....(((((-((((-...-..))))))))) ( -46.90) >ppk.1315 2585873 115 + 6181863 AUAUGGUGGGCGUAGCUCAGUUGGUAGAGCGCUGGAUUGUGAUUCCAGUUGUCGUGGGUUCGAUUCCCAUCGUCCACCCCAUAUUUUUCA-AGGGCGCCUCGAU-AGC---AUCUGGCGC ....((((((((..((((........))))((((((.......))))))....(((((.......)))))))))))))............-...(((((..(((-...---))).))))) ( -40.70) >pel.1044 1952749 118 + 5888780 AUAUGGUGGGCGUAGCUCAGUUGGUAGAGCGCCGGAUUGUGAUUCCGGUUGUCGUGGGUUCGAUUCCCAUCGUCCACCCCAGAUUCUGCA-GGGGCGCCUUGAU-UGAAUGAUCCGGCGC ....((((((((..((((........))))((((((.......))))))....(((((.......)))))))))))))............-...(((((..(((-(....)))).))))) ( -44.30) >consensus AUAUGGUGGGCGUAGCUCAGUUGGUAGAGCACAGGAUUGUGACUCCUGUUGUCGUGGGUUCGAUUCCCAUCGUCCACCCCAUAUUCCG_A_A_GGCGCC_AGAU_UAA_GGGUCUGGCGC ....((((((((..((((........)))).(((((.......))))).....(((((.......)))))))))))))................(((((.((((.......))))))))) (-42.61 = -41.17 + -1.44)
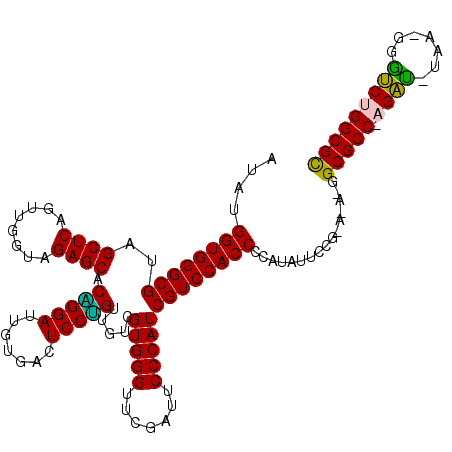
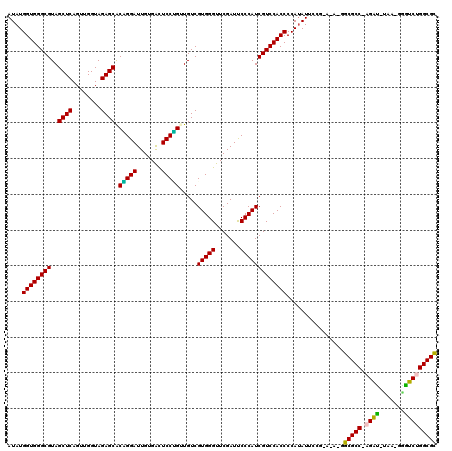
| Location | 1,947,442 – 1,947,555 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.91 |
| Mean single sequence MFE | -43.83 |
| Consensus MFE | -37.63 |
| Energy contribution | -37.47 |
| Covariance contribution | -0.17 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.80 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.963646 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1083 1947442 113 - 6264404 ACGCCAGGCCC-CGG-GCCU-GGCGC--U-U-CGGAAUAUGGGGUGGACGAUGGGAAUCGAACCCACGACACCAGGAGCCACAAUCCUGUGCUCUACCAACUGAGCUACGCCCACCAUAU .(((((((((.-..)-))))-))))(--.-.-..).((((((((((..((.((((.......)))))).)))).((.((.(((....)))((((........))))...)))).)))))) ( -47.70) >pau.1942 2850415 113 + 6537648 ACGCCAGGCCC-CGG-GCCU-GGCGC--U-U-CGGAAUAUGGGGUGGACGAUGGGAAUCGAACCCACGACACCAGGAGCCACAAUCCUGUGCUCUACCAACUGAGCUACGCCCACCAUAU .(((((((((.-..)-))))-))))(--.-.-..).((((((((((..((.((((.......)))))).)))).((.((.(((....)))((((........))))...)))).)))))) ( -47.70) >pfo.2168 2249823 116 + 6438405 GCGCCAGACUG-UUACAUCU-GGCGCCUU-U-CGAAAUAUGGGGUGGACGAUGGGGAUCGAACCCACGACAACGGGAGUCACAAUCCCGUGCUCUACCAACUGAGCUACGCCCACCAUAU ((((((((...-.....)))-)))))...-.-....((((((((((..((.((((.......))))))...((((((.......))))))((((........))))..))))..)))))) ( -46.70) >pf5.2421 2466463 116 + 7074893 GCGCCAGACUA-UUA-AUCU-GGCGCCUUCU-UCGAAUAUGGGGUGGACGAUGGGGAUCGAACCCACGACAACAGGAGUCACAAUCCUGUGCUCUACCAACUGAGCUACGCCCACCAUAU ((((((((...-...-.)))-))))).....-....((((((((((..((.((((.......))))))...((((((.......))))))((((........))))..))))..)))))) ( -44.90) >ppk.1315 2585873 115 - 6181863 GCGCCAGAU---GCU-AUCGAGGCGCCCU-UGAAAAAUAUGGGGUGGACGAUGGGAAUCGAACCCACGACAACUGGAAUCACAAUCCAGCGCUCUACCAACUGAGCUACGCCCACCAUAU (((((.(((---...-)))..)))))...-......((((((((((..((.((((.......))))))....(((((.......))))).((((........))))..))))..)))))) ( -36.80) >pel.1044 1952749 118 - 5888780 GCGCCGGAUCAUUCA-AUCAAGGCGCCCC-UGCAGAAUCUGGGGUGGACGAUGGGAAUCGAACCCACGACAACCGGAAUCACAAUCCGGCGCUCUACCAACUGAGCUACGCCCACCAUAU (((((((((.((((.-.....((((((((-..........))))))..((.((((.......))))))....)).))))....)))))))((((........))))...))......... ( -39.20) >consensus GCGCCAGACCA_UCA_AUCU_GGCGCC_U_U_CGGAAUAUGGGGUGGACGAUGGGAAUCGAACCCACGACAACAGGAGUCACAAUCCUGUGCUCUACCAACUGAGCUACGCCCACCAUAU (((((((((.......)))).)))))..........((((((((((..((.((((.......))))))....(((((.......))))).((((........))))..))))..)))))) (-37.63 = -37.47 + -0.17)
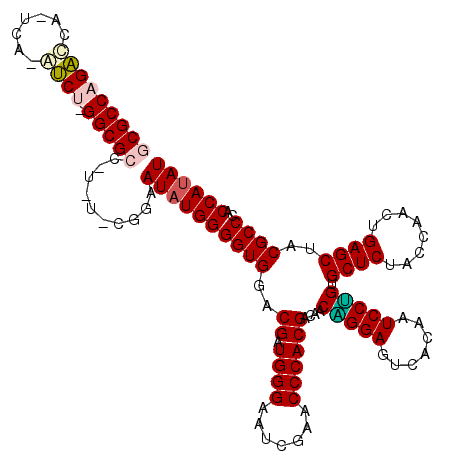
| Location | 1,947,482 – 1,947,593 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.07 |
| Mean single sequence MFE | -44.00 |
| Consensus MFE | -30.36 |
| Energy contribution | -29.62 |
| Covariance contribution | -0.74 |
| Combinations/Pair | 1.38 |
| Mean z-score | -2.67 |
| Structure conservation index | 0.69 |
| SVM decision value | 2.24 |
| SVM RNA-class probability | 0.990973 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1083 1947482 111 + 6264404 GGCUCCUGGUGUCGUGGGUUCGAUUCCCAUCGUCCACCCCAUAUUCCG-A-A--GCGCC-AGGC-CCG-GGGCCUGGCGUUUUCA-UUUC-CAAGCAGUGUCCCGCGGACGUGGUGGAAU ((.....((((.((((((.......)))).))..)))).......))(-(-(--(((((-((((-(..-.)))))))))))))..-.(((-((..((.(((((...))))))).))))). ( -45.80) >pfo.2168 2249863 115 - 6438405 GACUCCCGUUGUCGUGGGUUCGAUCCCCAUCGUCCACCCCAUAUUUCG-A-AAGGCGCC-AGAUGUAA-CAGUCUGGCGCCUUUG-CUUUAAAAAGCUUCAUCUGCGGAUGUGGUGGAAU (((.......)))(((((.......)))))..((((((.((...((((-(-((((((((-((((....-..)))))))))))))(-(((....))))........)))))).)))))).. ( -46.60) >pf5.2421 2466503 114 - 7074893 GACUCCUGUUGUCGUGGGUUCGAUCCCCAUCGUCCACCCCAUAUUCGA-AGAAGGCGCC-AGAU-UAA-UAGUCUGGCGCCUUUG-CUUU-AAAAGCUUGUUACGCGGACGUGGUGGAAU (((((.((....)).)))))...((((((.(((((.............-.(((((((((-((((-...-..)))))))))))))(-((..-...))).........)))))))).))).. ( -45.20) >ppk.1315 2585913 109 + 6181863 GAUUCCAGUUGUCGUGGGUUCGAUUCCCAUCGUCCACCCCAUAUUUUUCA-AGGGCGCCUCGAU-AGC---AUCUGGCGCCUUUG-UUUU-AAGCGUU----ACGCGGACGUGGUGAAAU ((..(((.......)))..))..((((((.(((((.............((-((((((((..(((-...---))).))))))))))-....-..(((..----.))))))))))).))).. ( -38.40) >pel.1044 1952789 113 + 5888780 GAUUCCGGUUGUCGUGGGUUCGAUUCCCAUCGUCCACCCCAGAUUCUGCA-GGGGCGCCUUGAU-UGAAUGAUCCGGCGCCUUUGUUUUU-GAGUUUC----AUGCGGACGUGGUGAAAU ((..(((.......)))..))..((((((.(((((.(...((((((.(((-((((((((..(((-(....)))).)))))))))))....-)))))).----..).)))))))).))).. ( -43.30) >pss.1031 1981523 113 + 6093698 GACUCCUGCGGUCGUGGGUUCGAACCCCAUCGUCCACCCCAUAUUAAG-A-AGGGCGCC-AGAU-UCA-UAGUCUGGCGCCUUUG-CUUU-AACAGUUUGAAAUGCGGAUGUGGUGGAAU .........((.((((((.......)))).)).)).(((((((((...-(-((((((((-((((-...-..)))))))))))))(-((((-((....)))))..)).))))))).))... ( -44.70) >consensus GACUCCUGUUGUCGUGGGUUCGAUCCCCAUCGUCCACCCCAUAUUCCG_A_AGGGCGCC_AGAU_UAA_UAGUCUGGCGCCUUUG_CUUU_AAAAGUUUG__ACGCGGACGUGGUGGAAU ((..((.........))..))..((((((.(((((................((((((((.((((.......))))))))))))............((.......)))))))))).))).. (-30.36 = -29.62 + -0.74)

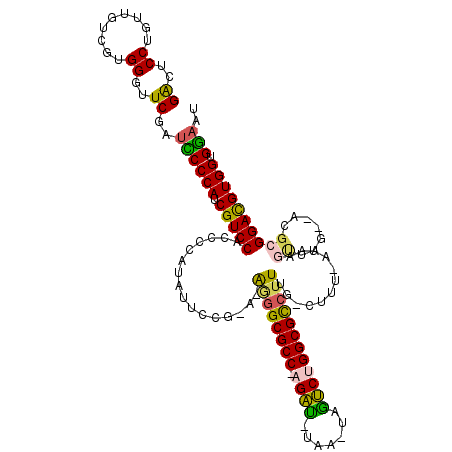
| Location | 1,947,482 – 1,947,593 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.07 |
| Mean single sequence MFE | -41.25 |
| Consensus MFE | -26.93 |
| Energy contribution | -27.43 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.75 |
| Structure conservation index | 0.65 |
| SVM decision value | 2.33 |
| SVM RNA-class probability | 0.992426 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1083 1947482 111 - 6264404 AUUCCACCACGUCCGCGGGACACUGCUUG-GAAA-UGAAAACGCCAGGCCC-CGG-GCCU-GGCGC--U-U-CGGAAUAUGGGGUGGACGAUGGGAAUCGAACCCACGACACCAGGAGCC .((((.(((..(((((((....))))...-....-.(((..(((((((((.-..)-))))-)))).--)-)-))))...)))((((..((.((((.......)))))).)))).)))).. ( -49.30) >pfo.2168 2249863 115 + 6438405 AUUCCACCACAUCCGCAGAUGAAGCUUUUUAAAG-CAAAGGCGCCAGACUG-UUACAUCU-GGCGCCUU-U-CGAAAUAUGGGGUGGACGAUGGGGAUCGAACCCACGACAACGGGAGUC (((((.((((.((((........((((....)))-)((((((((((((...-.....)))-))))))))-)-.......)))))))).((.((((.......))))))......))))). ( -45.10) >pf5.2421 2466503 114 + 7074893 AUUCCACCACGUCCGCGUAACAAGCUUUU-AAAG-CAAAGGCGCCAGACUA-UUA-AUCU-GGCGCCUUCU-UCGAAUAUGGGGUGGACGAUGGGGAUCGAACCCACGACAACAGGAGUC (((((....(((((((.......(((...-..))-).(((((((((((...-...-.)))-))))))))..-...........))))))).((((.......))))........))))). ( -44.60) >ppk.1315 2585913 109 - 6181863 AUUUCACCACGUCCGCGU----AACGCUU-AAAA-CAAAGGCGCCAGAU---GCU-AUCGAGGCGCCCU-UGAAAAAUAUGGGGUGGACGAUGGGAAUCGAACCCACGACAACUGGAAUC ((((((((((.((((((.----..)))..-....-(((.((((((.(((---...-)))..)))))).)-))........))))))).((.((((.......)))))).....)))))). ( -34.90) >pel.1044 1952789 113 - 5888780 AUUUCACCACGUCCGCAU----GAAACUC-AAAAACAAAGGCGCCGGAUCAUUCA-AUCAAGGCGCCCC-UGCAGAAUCUGGGGUGGACGAUGGGAAUCGAACCCACGACAACCGGAAUC (((((.((((((((((..----.......-.........((((((.(((......-)))..))))))((-(.........)))))))))).)))...(((......))).....))))). ( -34.20) >pss.1031 1981523 113 - 6093698 AUUCCACCACAUCCGCAUUUCAAACUGUU-AAAG-CAAAGGCGCCAGACUA-UGA-AUCU-GGCGCCCU-U-CUUAAUAUGGGGUGGACGAUGGGGUUCGAACCCACGACCGCAGGAGUC (((((.((.(((..(((........))).-.(((-.((.(((((((((...-...-.)))-)))))).)-)-)))...)))))((((.((.((((.......)))))).)))).))))). ( -39.40) >consensus AUUCCACCACGUCCGCAU__CAAACUUUU_AAAA_CAAAGGCGCCAGACUA_UCA_AUCU_GGCGCCCU_U_CAGAAUAUGGGGUGGACGAUGGGAAUCGAACCCACGACAACAGGAGUC (((((....(((((((.......................(((((((((.........))).)))))).....(........).))))))).((((.......))))........))))). (-26.93 = -27.43 + 0.50)
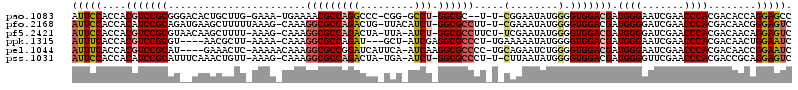
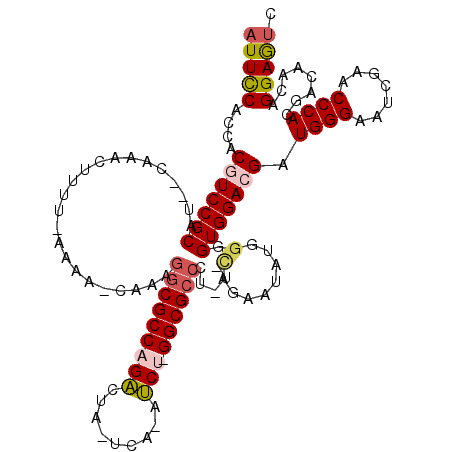
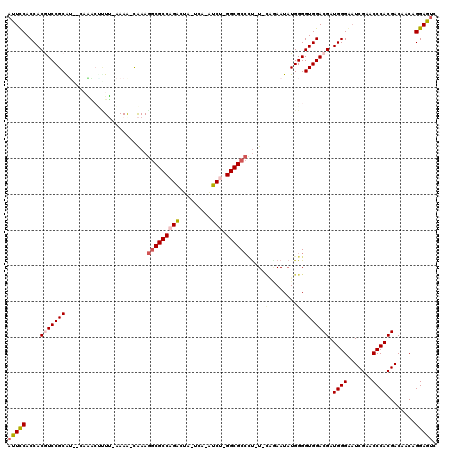
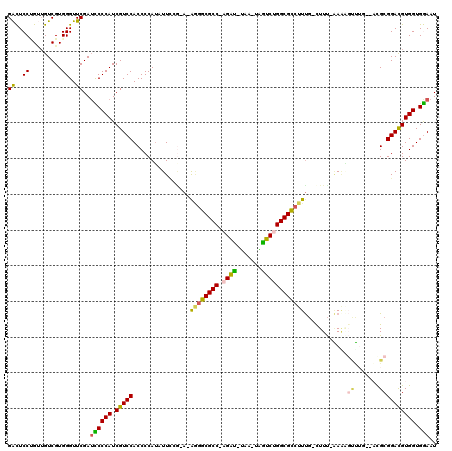
| Location | 1,947,522 – 1,947,633 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 75.81 |
| Mean single sequence MFE | -44.75 |
| Consensus MFE | -31.14 |
| Energy contribution | -29.87 |
| Covariance contribution | -1.27 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.61 |
| Structure conservation index | 0.70 |
| SVM decision value | 2.24 |
| SVM RNA-class probability | 0.990984 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1083 1947522 111 + 6264404 AUAUUCCG-A-A--GCGCC-AGGC-CCG-GGGCCUGGCGUUUUCA-UUUC-CAAGCAGUGUCCCGCGGACGUGGUGGAAUUGGUAGACACACUGGAUUUAGGUUCCAGCGCCGCAAGGCG .......(-(-(--(((((-((((-(..-.)))))))))))))((-.(((-((..((.(((((...))))))).))))).)).........(((((.......)))))((((....)))) ( -50.40) >pau.1942 2850495 111 - 6537648 AUAUUCCG-A-A--GCGCC-AGGC-CCG-GGGCCUGGCGUUUUCA-UUUC-CAAGCAGUGUCCCGCGGACGUGGUGGAAUUGGUAGACACACUGGAUUUAGGUUCCAGCGCCGCAAGGCG .......(-(-(--(((((-((((-(..-.)))))))))))))((-.(((-((..((.(((((...))))))).))))).)).........(((((.......)))))((((....)))) ( -50.40) >pfo.2168 2249903 115 - 6438405 AUAUUUCG-A-AAGGCGCC-AGAUGUAA-CAGUCUGGCGCCUUUG-CUUUAAAAAGCUUCAUCUGCGGAUGUGGUGGAAUUGGUAGACACACUGGAUUUAGGUUCCAGCGCCGCGAGGCG ........-(-((((((((-((((....-..)))))))))))))(-(((....))))....(((((.(((........))).)))))....(((((.......)))))((((....)))) ( -47.90) >pf5.2421 2466543 114 - 7074893 AUAUUCGA-AGAAGGCGCC-AGAU-UAA-UAGUCUGGCGCCUUUG-CUUU-AAAAGCUUGUUACGCGGACGUGGUGGAAUUGGUAGACACACUGGAUUUAGGUUCCAGCGCCGCAAGGUG ........-.(((((((((-((((-...-..)))))))))))))(-((..-...))).......((((.(((..(((((((...(..(.....)..)...))))))))))))))...... ( -43.10) >ppk.1315 2585953 109 + 6181863 AUAUUUUUCA-AGGGCGCCUCGAU-AGC---AUCUGGCGCCUUUG-UUUU-AAGCGUU----ACGCGGACGUGGUGAAAUUGGUAGACACACUGGAUUUAGGUUCCAGCGGCGCAAGCUG ........((-((((((((..(((-...---))).))))))))))-((((-(.(((((----.....)))))..)))))............(((((.......)))))((((....)))) ( -36.40) >pel.1044 1952829 113 + 5888780 AGAUUCUGCA-GGGGCGCCUUGAU-UGAAUGAUCCGGCGCCUUUGUUUUU-GAGUUUC----AUGCGGACGUGGUGAAAUUGGUAGACACACUGGAUUUAGGUUCCAGCGGCGCAAGCUG ....((((((-((((((((..(((-(....)))).)))))))))......-.((((((----((((....)).)))))))).)))))....(((((.......)))))((((....)))) ( -40.30) >consensus AUAUUCCG_A_A_GGCGCC_AGAU_UAA_GGGUCUGGCGCCUUUG_UUUU_AAAAGAUUGU_ACGCGGACGUGGUGGAAUUGGUAGACACACUGGAUUUAGGUUCCAGCGCCGCAAGGCG ...........((((((((.((((.......))))))))))))....................(((....)))(((...........))).(((((.......)))))((((....)))) (-31.14 = -29.87 + -1.27)

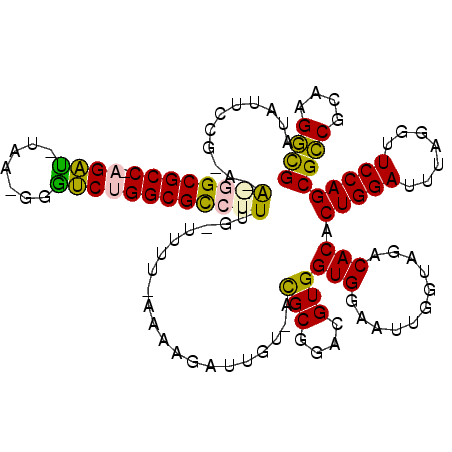
| Location | 1,947,522 – 1,947,633 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 75.81 |
| Mean single sequence MFE | -37.93 |
| Consensus MFE | -23.56 |
| Energy contribution | -24.56 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.77 |
| Structure conservation index | 0.62 |
| SVM decision value | 2.41 |
| SVM RNA-class probability | 0.993560 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1083 1947522 111 - 6264404 CGCCUUGCGGCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUCCACCACGUCCGCGGGACACUGCUUG-GAAA-UGAAAACGCCAGGCCC-CGG-GCCU-GGCGC--U-U-CGGAAUAU (((...)))((((((((.......))))))))........(((((..((..(((((((....))))..)-))..-))....(((((((((.-..)-))))-)))).--.-.-.))))).. ( -45.60) >pau.1942 2850495 111 + 6537648 CGCCUUGCGGCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUCCACCACGUCCGCGGGACACUGCUUG-GAAA-UGAAAACGCCAGGCCC-CGG-GCCU-GGCGC--U-U-CGGAAUAU (((...)))((((((((.......))))))))........(((((..((..(((((((....))))..)-))..-))....(((((((((.-..)-))))-)))).--.-.-.))))).. ( -45.60) >pfo.2168 2249903 115 + 6438405 CGCCUCGCGGCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUCCACCACAUCCGCAGAUGAAGCUUUUUAAAG-CAAAGGCGCCAGACUG-UUACAUCU-GGCGCCUU-U-CGAAAUAU (((...)))((((((((.......)))))))).................((((....))))..((((....)))-)((((((((((((...-.....)))-))))))))-)-........ ( -39.00) >pf5.2421 2466543 114 + 7074893 CACCUUGCGGCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUCCACCACGUCCGCGUAACAAGCUUUU-AAAG-CAAAGGCGCCAGACUA-UUA-AUCU-GGCGCCUUCU-UCGAAUAU ......(((((((((((.......))))))(((..............)))).)))).......(((...-..))-).(((((((((((...-...-.)))-))))))))..-........ ( -36.84) >ppk.1315 2585953 109 - 6181863 CAGCUUGCGCCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUUCACCACGUCCGCGU----AACGCUU-AAAA-CAAAGGCGCCAGAU---GCU-AUCGAGGCGCCCU-UGAAAAAUAU .(((((((((.((((((.......))))))(((..............)))....))))----)).))).-....-(((.((((((.(((---...-)))..)))))).)-))........ ( -33.44) >pel.1044 1952829 113 - 5888780 CAGCUUGCGCCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUUCACCACGUCCGCAU----GAAACUC-AAAAACAAAGGCGCCGGAUCAUUCA-AUCAAGGCGCCCC-UGCAGAAUCU ....(((((((((((((.......))))))).))........................----.......-.........((((((.(((......-)))..))))))..-.))))..... ( -27.10) >consensus CACCUUGCGGCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUCCACCACGUCCGCGG_ACAAACCUUU_AAAA_CAAAGGCGCCAGACCA_UCA_AUCU_GGCGCC_U_U_CGGAAUAU ......((((.((((((.......))))))(((..............)))..)))).......................((((((((((.......)))).))))))............. (-23.56 = -24.56 + 1.00)

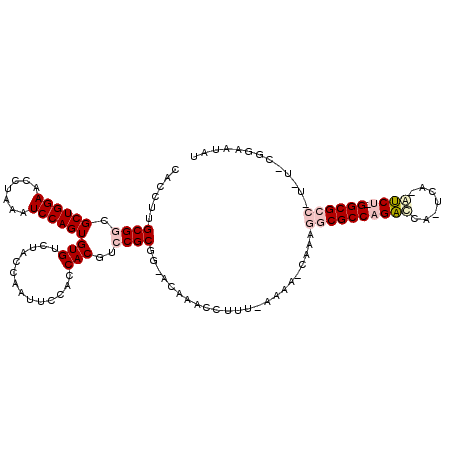
| Location | 1,947,544 – 1,947,662 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.56 |
| Mean single sequence MFE | -37.77 |
| Consensus MFE | -31.35 |
| Energy contribution | -30.05 |
| Covariance contribution | -1.30 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.627427 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1083 1947544 118 - 6264404 UGGUGCGGACGGAGAGACUCGAACUCUCACGCCUUGCGGCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUCCACCACGUCCGCGGGACACUGCUUG-GAAA-UGAAAACGCCAGGCCC ...(((((((((((((.......))))).(((...)))((((((((.......)))))))).................)))))))).......(((((-(...-(....)..)))))).. ( -41.60) >pau.1942 2850517 118 + 6537648 UGGUGCGGACGGAGAGACUCGAACUCUCACGCCUUGCGGCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUCCACCACGUCCGCGGGACACUGCUUG-GAAA-UGAAAACGCCAGGCCC ...(((((((((((((.......))))).(((...)))((((((((.......)))))))).................)))))))).......(((((-(...-(....)..)))))).. ( -41.60) >pfo.2168 2249928 119 + 6438405 UGGUGCGGAUGAAGAGACUCGAACUCUUACGCCUCGCGGCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUCCACCACAUCCGCAGAUGAAGCUUUUUAAAG-CAAAGGCGCCAGACUG ((((((((((((((((.......))))).(((...)))((((((((.......)))))))).................))))))).......((((((.....-.))))))))))..... ( -38.10) >pf5.2421 2466568 118 + 7074893 UGGUGCGGACGAAGAGACUCGAACUCUUACACCUUGCGGCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUCCACCACGUCCGCGUAACAAGCUUUU-AAAG-CAAAGGCGCCAGACUA ((((((((((((((((.......)))))..........((((((((.......)))))))).................))))))).......((((((-....-.))))))))))..... ( -38.00) >ppk.1315 2585979 112 - 6181863 UGGUGCGGACGAAGAGACUCGAACUCUUACAGCUUGCGCCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUUCACCACGUCCGCGU----AACGCUU-AAAA-CAAAGGCGCCAGAU-- ((((((((((((((((.......))))).......((((.((((((.......))))))))))...............)))))))..----...((((-....-...))))))))...-- ( -35.50) >pel.1044 1952856 115 - 5888780 UGGUGCGGACGAAGAGACUCGAACUCUUACAGCUUGCGCCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUUCACCACGUCCGCAU----GAAACUC-AAAAACAAAGGCGCCGGAUCA .(((((((((((((((.......))))).......((((.((((((.......))))))))))...............)))))...(----(.....)-).........)))))...... ( -31.80) >consensus UGGUGCGGACGAAGAGACUCGAACUCUUACACCUUGCGGCGCUGGAACCUAAAUCCAGUGUGUCUACCAAUUCCACCACGUCCGCGG_ACAAACCUUU_AAAA_CAAAGGCGCCAGACCA ((((((((((((((((.......))))).......(((.(((((((.......))))))))))...............)))))))...................(....).))))..... (-31.35 = -30.05 + -1.30)


Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:12:53 2007