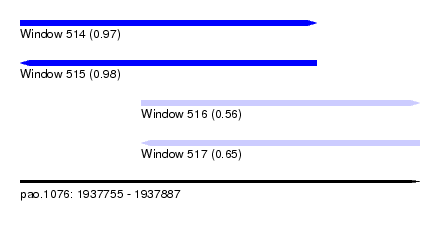
| Sequence ID | pao.1076 |
|---|---|
| Location | 1,937,755 – 1,937,887 |
| Length | 132 |
| Max. P | 0.978928 |

| Location | 1,937,755 – 1,937,853 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 69.99 |
| Mean single sequence MFE | -45.72 |
| Consensus MFE | -18.39 |
| Energy contribution | -17.97 |
| Covariance contribution | -0.41 |
| Combinations/Pair | 1.44 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.40 |
| SVM decision value | 1.69 |
| SVM RNA-class probability | 0.972537 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1076 1937755 98 + 6264404 UUCCAGACAGAAGCUGUUUUCAAAGUUUUACGCCGGCGGGCUGGGGUG-------GGGCUGGCGA--GACGCUU--------GAGGGAGGCGCC--GUCGCCAGCGUACCUGCCGGA- .........((((((........))))))...((((((((((.....)-------).((((((((--(.(((((--------.....))))).)--.))))))))...)))))))).- ( -41.50) >ppk.1364 2672426 111 + 6181863 AUCCUUACAGAAGCUGCUUUCAAAGUUUUACGCCUGCAGAACGGG-GACUGAUGAGGGCUGGCGC--GGGCAUG-CGAGGG--AUGCAUGGCCCGGGCCAACGCCGUUC-UACCGGAU ((((....((((((.(((((((.((((.....((((.....))))-))))..)))))))((((.(--(((((((-((....--.))))).))))).))))..))..)))-)...)))) ( -42.30) >pst.2228 2157993 110 - 6397126 UUCCUAACAGAAGCUGUUUUCAAAGUUUUACGCCUGCAGAACGGGGGG--AGGGACGGUCGACAC--GGCCAUU-CUUGAUGAGGGAAUGGCCC--GUCGACGCCGCAA-UGCGGGGU (((((..(.((((((........))))))...((((.....)))))..--)))))..((((((..--(((((((-(((.....)))))))))).--)))))).((((..-.))))... ( -44.00) >pfo.1992 2567069 113 - 6438405 UUCCUUACAGAAGCUGCUUUCAAAGUUUUACGCCUGCAGAACGGGGAGCUGAUGAGGGUUGGCGUUGGGCUUUUCCCCGCUGGAAAAACCCUAC--GCCAACACCGCUC-UGAAGG-- ..((((.((((.((.(((..(...(((((........))))).)..)))......((((((((((.(((.((((((.....)))))).))).))--)))))).))))))-))))))-- ( -48.80) >pss.1020 1961627 110 + 6093698 UUCCUAACAGAAGCUGUUUUCAAAGUUUUACGCCUGCAGGGCGGGGGG--AGUGGCGGUCGACAC--GGCCAUU-CGUGAUGAGGGAAUGGCCU--GUCGACGCCGCAC-UGCGGGAU .((((....((((((........)))))).((((.....)))).(.((--.((((((.((((((.--(((((((-(.(.....).)))))))))--))))))))))).)-).))))). ( -52.70) >psp.2080 1835236 110 - 5928787 UUCCUAACAGAAGCUGUUUUCAAAGUUUUACGCCUGCAGAACGGGGGA--AGUGACGGUCGACAC--GGCCAUU-CUUGAUGAGGGAAUGGCCU--GUCGACGCCGCAA-UGCGGGGU .........((((((........))))))...((((((...((((...--.....).(((((((.--(((((((-(((.....)))))))))))--)))))).)))...-)))))).. ( -45.00) >consensus UUCCUAACAGAAGCUGUUUUCAAAGUUUUACGCCUGCAGAACGGGGGG__AGUGACGGUCGACAC__GGCCAUU_CGUGAUGAAGGAAUGGCCC__GUCGACGCCGCAC_UGCGGGAU .........((((((........))))))...((((((...(((.............((((((....((.((((............)))).))...)))))).)))....)))))).. (-18.39 = -17.97 + -0.41)

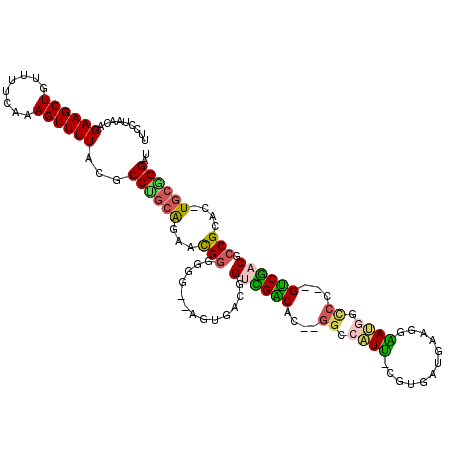
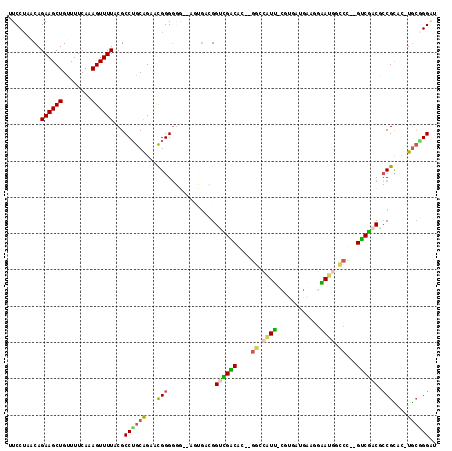
| Location | 1,937,755 – 1,937,853 |
|---|---|
| Length | 98 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 69.99 |
| Mean single sequence MFE | -41.25 |
| Consensus MFE | -11.09 |
| Energy contribution | -11.79 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.79 |
| Structure conservation index | 0.27 |
| SVM decision value | 1.83 |
| SVM RNA-class probability | 0.978928 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1076 1937755 98 - 6264404 -UCCGGCAGGUACGCUGGCGAC--GGCGCCUCCCUC--------AAGCGUC--UCGCCAGCCC-------CACCCCAGCCCGCCGGCGUAAAACUUUGAAAACAGCUUCUGUCUGGAA -(((((((((..(((((((((.--(((((.......--------..)))))--))))))))..-------.......(((....))).................)..)))).))))). ( -38.10) >ppk.1364 2672426 111 - 6181863 AUCCGGUA-GAACGGCGUUGGCCCGGGCCAUGCAU--CCCUCG-CAUGCCC--GCGCCAGCCCUCAUCAGUC-CCCGUUCUGCAGGCGUAAAACUUUGAAAGCAGCUUCUGUAAGGAU ((((.(((-((((((.((((((.(((((.((((..--.....)-)))))))--).)))))).((....))..-.)))))))))..................((((...))))..)))) ( -42.60) >pst.2228 2157993 110 + 6397126 ACCCCGCA-UUGCGGCGUCGAC--GGGCCAUUCCCUCAUCAAG-AAUGGCC--GUGUCGACCGUCCCU--CCCCCCGUUCUGCAGGCGUAAAACUUUGAAAACAGCUUCUGUUAGGAA ((((.(((-..((((.((((((--(((((((((.........)-)))))))--.))))))).......--....))))..))).)).))...........(((((...)))))..... ( -35.70) >pfo.1992 2567069 113 + 6438405 --CCUUCA-GAGCGGUGUUGGC--GUAGGGUUUUUCCAGCGGGGAAAAGCCCAACGCCAACCCUCAUCAGCUCCCCGUUCUGCAGGCGUAAAACUUUGAAAGCAGCUUCUGUAAGGAA --((((((-((((((.((((((--((.((((((((((.....)))))))))).))))))))))......(((..(.(((.(((....))).)))...)..))).)).)))).)))).. ( -49.30) >pss.1020 1961627 110 - 6093698 AUCCCGCA-GUGCGGCGUCGAC--AGGCCAUUCCCUCAUCACG-AAUGGCC--GUGUCGACCGCCACU--CCCCCCGCCCUGCAGGCGUAAAACUUUGAAAACAGCUUCUGUUAGGAA .(((.(((-(.(((((((((((--(((((((((.........)-)))))))--.)))))).))))...--.....((((.....))))................))..))))..))). ( -44.90) >psp.2080 1835236 110 + 5928787 ACCCCGCA-UUGCGGCGUCGAC--AGGCCAUUCCCUCAUCAAG-AAUGGCC--GUGUCGACCGUCACU--UCCCCCGUUCUGCAGGCGUAAAACUUUGAAAACAGCUUCUGUUAGGAA .((.(((.-(((((((((((((--(((((((((.........)-)))))))--.)))))).))))((.--......))...)))))))............(((((...))))).)).. ( -36.90) >consensus AUCCCGCA_GUGCGGCGUCGAC__GGGCCAUUCCCUCAUCAAG_AAUGGCC__GCGCCAACCCUCACU__CCCCCCGUUCUGCAGGCGUAAAACUUUGAAAACAGCUUCUGUUAGGAA ..((.(((...((((.((((((...((.((((............)))).))....)))))).............))))..))).))...............((((...))))...... (-11.09 = -11.79 + 0.70)
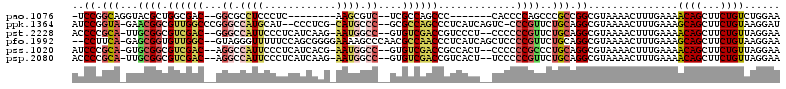
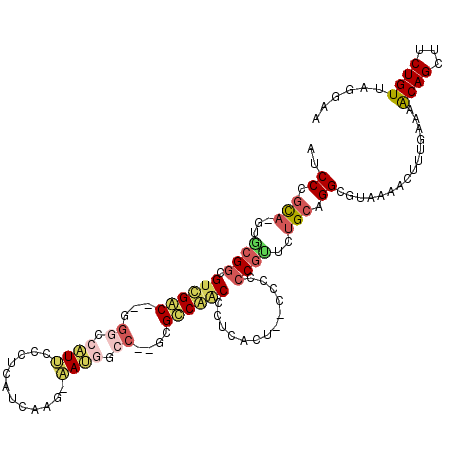
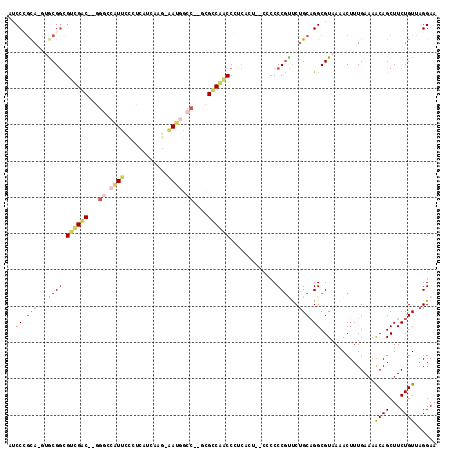
| Location | 1,937,795 – 1,937,887 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 66.92 |
| Mean single sequence MFE | -44.63 |
| Consensus MFE | -11.81 |
| Energy contribution | -11.26 |
| Covariance contribution | -0.55 |
| Combinations/Pair | 1.60 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.26 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.559636 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1076 1937795 92 + 6264404 CUGGGGUG-------GGGCUGGCGA--GACGCUU--------GAGGGAGGCGCC--GUCGCCAGCGUACCUGCCGGA--AAC-----UGUGCUCGUGACGCUUUGAAAACAGCUUCUA ....((..-------((((((((((--(.(((((--------.....))))).)--.))))))))...))..))(((--(.(-----(((..(((........)))..)))).)))). ( -39.00) >ppk.1364 2672466 106 + 6181863 ACGGG-GACUGAUGAGGGCUGGCGC--GGGCAUG-CGAGGG--AUGCAUGGCCCGGGCCAACGCCGUUC-UACCGGAUCAAC-----UGUGCUCGUGACGCUUUGAAAACAGCUUCCA (((((-.((.(.(((.(((((((.(--(((((((-((....--.))))).))))).))))..)))..((-.....))))).)-----.)).)))))((.(((.(....).))).)).. ( -41.20) >pf5.1596 2914685 110 + 7074893 ACGGGGAGCUGAUGAGGGUUGGCGUUGGGUUUUUCCCCGCUGGAAAAACCCCUC--GCCAACACCGCUC-UGAAGG---AAC--GACUGUGCUCGUGACGCUUUGAAAACAGCUUCAA ....(((((((....(((((((((..((((((((((.....))))))))))..)--)))))).))....-....(.---.((--((......))))..)..........))))))).. ( -47.90) >pst.2228 2158033 110 - 6397126 ACGGGGGG--AGGGACGGUCGACAC--GGCCAUU-CUUGAUGAGGGAAUGGCCC--GUCGACGCCGCAA-UGCGGGGUUAACCUGACUGUGCUCGUGACGCUUUGAAAACAGCUUCCA .....(((--(((((((((((((..--(((((((-(((.....)))))))))).--)))(((.((((..-.)))).))).....)))))).))).....(((.(....).))))))). ( -45.50) >pfo.1992 2567109 110 - 6438405 ACGGGGAGCUGAUGAGGGUUGGCGUUGGGCUUUUCCCCGCUGGAAAAACCCUAC--GCCAACACCGCUC-UGAAGG---AAC--GACUGUGCUCGUGACGCUUUGAAAACAGCUUCUA ...((((((((....((((((((((.(((.((((((.....)))))).))).))--)))))).))....-....(.---.((--((......))))..)..........)))))))). ( -46.00) >pss.1020 1961667 110 + 6093698 GCGGGGGG--AGUGGCGGUCGACAC--GGCCAUU-CGUGAUGAGGGAAUGGCCU--GUCGACGCCGCAC-UGCGGGAUUAACUUGACUGUGCUCGUGACGCUUUGAAAACAGCUUCCA .....(((--(((((((.((((((.--(((((((-(.(.....).)))))))))--)))))))))((((-..((((.....))))...))))...................)))))). ( -48.20) >consensus ACGGGGGG__GAUGAGGGUUGGCGC__GGCCAUU_CCCGAUGGAGGAAUGCCCC__GCCAACACCGCAC_UGCAGGA__AAC__GACUGUGCUCGUGACGCUUUGAAAACAGCUUCCA (((((............((((((......(((((........))))).........)))))).((((....)).))...............)))))((.(((........))).)).. (-11.81 = -11.26 + -0.55)

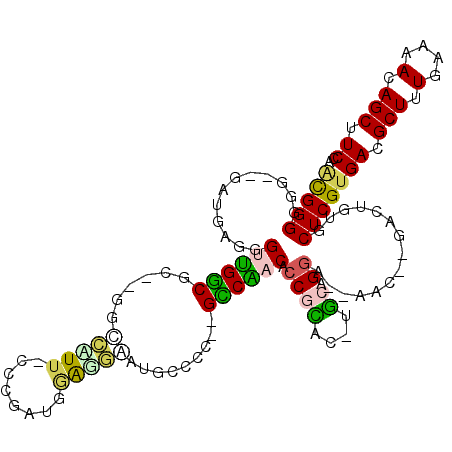
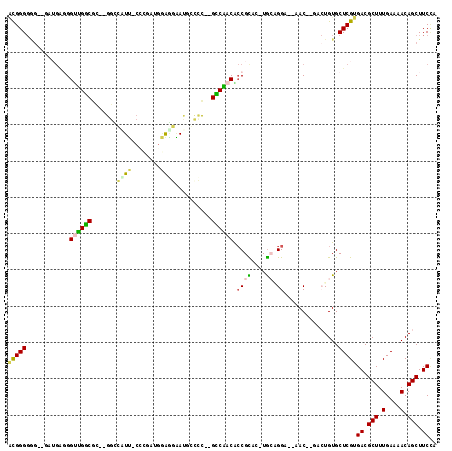
| Location | 1,937,795 – 1,937,887 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 66.92 |
| Mean single sequence MFE | -41.12 |
| Consensus MFE | -7.77 |
| Energy contribution | -7.49 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.38 |
| Structure conservation index | 0.19 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.654356 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao.1076 1937795 92 - 6264404 UAGAAGCUGUUUUCAAAGCGUCACGAGCACA-----GUU--UCCGGCAGGUACGCUGGCGAC--GGCGCCUCCCUC--------AAGCGUC--UCGCCAGCCC-------CACCCCAG ..((((((((.(((..........))).)))-----)))--)).((..(((..((((((((.--(((((.......--------..)))))--))))))))..-------.))))).. ( -36.90) >ppk.1364 2672466 106 - 6181863 UGGAAGCUGUUUUCAAAGCGUCACGAGCACA-----GUUGAUCCGGUA-GAACGGCGUUGGCCCGGGCCAUGCAU--CCCUCG-CAUGCCC--GCGCCAGCCCUCAUCAGUC-CCCGU .(((.(((((((((...(.(((((.......-----).)))).).).)-)))))))((((((.(((((.((((..--.....)-)))))))--).)))))).........))-).... ( -39.20) >pf5.1596 2914685 110 - 7074893 UUGAAGCUGUUUUCAAAGCGUCACGAGCACAGUC--GUU---CCUUCA-GAGCGGUGUUGGC--GAGGGGUUUUUCCAGCGGGGAAAAACCCAACGCCAACCCUCAUCAGCUCCCCGU .(((.(((........))).))).((((......--(((---(.....-))))((.((((((--(..((((((((((.....))))))))))..)))))))))......))))..... ( -46.60) >pst.2228 2158033 110 + 6397126 UGGAAGCUGUUUUCAAAGCGUCACGAGCACAGUCAGGUUAACCCCGCA-UUGCGGCGUCGAC--GGGCCAUUCCCUCAUCAAG-AAUGGCC--GUGUCGACCGUCCCU--CCCCCCGU ..((.(((........))).))(((.(((..((..((....))..)).-.)))(((((((((--(((((((((.........)-)))))))--.)))))).))))...--.....))) ( -36.90) >pfo.1992 2567109 110 + 6438405 UAGAAGCUGUUUUCAAAGCGUCACGAGCACAGUC--GUU---CCUUCA-GAGCGGUGUUGGC--GUAGGGUUUUUCCAGCGGGGAAAAGCCCAACGCCAACCCUCAUCAGCUCCCCGU ..((.(((........))).))..((((......--(((---(.....-))))((.((((((--((.((((((((((.....)))))))))).))))))))))......))))..... ( -47.30) >pss.1020 1961667 110 - 6093698 UGGAAGCUGUUUUCAAAGCGUCACGAGCACAGUCAAGUUAAUCCCGCA-GUGCGGCGUCGAC--AGGCCAUUCCCUCAUCACG-AAUGGCC--GUGUCGACCGCCACU--CCCCCCGC .(((.(((........))).......((((.((............)).-))))(((((((((--(((((((((.........)-)))))))--.)))))).))))..)--))...... ( -39.80) >consensus UGGAAGCUGUUUUCAAAGCGUCACGAGCACAGUC__GUU__UCCCGCA_GAGCGGCGUCGAC__GGGCCAUUCCUCCAGCACG_AAUGGCC__GCGCCAACCCUCAUC__CCCCCCGU ..((.(((........))).))..................................((((((.................................))))))................. ( -7.77 = -7.49 + -0.28)

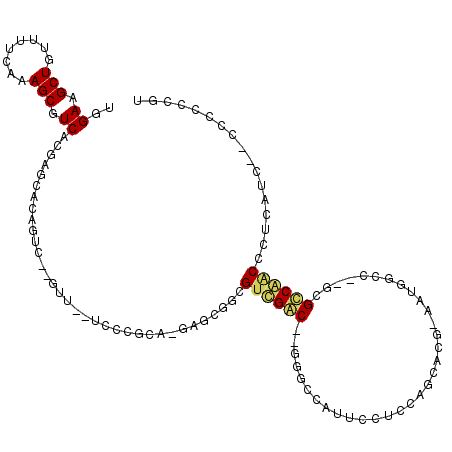
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 21:12:40 2007