
| Sequence ID | pao_1 |
|---|---|
| Location | 5,332,038 – 5,332,175 |
| Length | 137 |
| Max. P | 0.999800 |

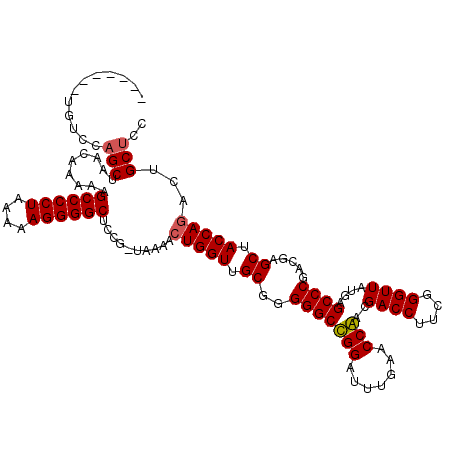
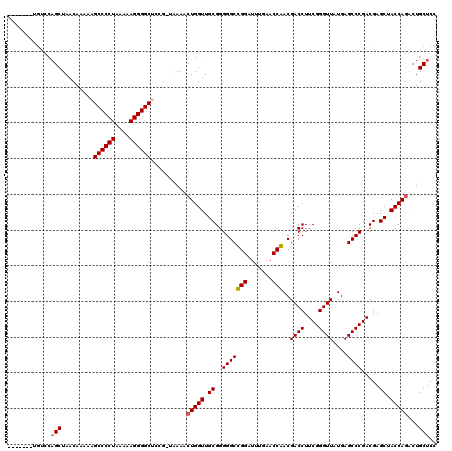
| Location | 5,332,038 – 5,332,157 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.52 |
| Mean single sequence MFE | -43.83 |
| Consensus MFE | -38.55 |
| Energy contribution | -38.88 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.89 |
| SVM RNA-class probability | 0.981441 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 5332038 119 + 1 CAUGGACUGUCCAGCUAACAAAAAGCCCCUAAAAAGGGGCCCUG-AAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCC ..(((...((((((((........((((((....))))))((((-...((....)).)))))))))))).....))).......((((((.....)))))).((((.........)))). ( -44.50) >pau_1 1 119 + 1 CAUGGACUGUCCAGCUAACAAAAAGCCCCUAAAAAGGGGCCCUG-AAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCC ..(((...((((((((........((((((....))))))((((-...((....)).)))))))))))).....))).......((((((.....)))))).((((.........)))). ( -44.50) >pel_1 1 107 + 1 -------------GCUAGCAAAAAGCCCCUGAAAAGGGGCUCUGCUGAAGCUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAAGCUGCUCC -------------(((((((...(((((((....))))))).))))).(((((((.((..(((((((.......))...((((....))))...))))).....)).))).))))))... ( -47.30) >pf5_1 1 113 + 1 -------UGUCCAGCUAACAAAAAGCCCCUGAAAAGGGGCUCCGCUAAAACUGGUUGCGGGGGCCGGAUUUGAACCGACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCC -------.(((............(((((((....)))))))((((...........))))(((((((.......)))..((((....))))....)))))))((((.........)))). ( -44.50) >pfo_1 1 111 + 1 -------UGUCCAGCUAACAAAAAGCCCCUUAAAAGGGGCUCC--UUAAACUGGUUGCGGGGGCCGGAUUUGAACCGACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCC -------.....(((........(((((((....)))))))..--.....(((((.((..(((((((.......)))..((((....))))....)))).....)).)))))...))).. ( -40.30) >pfs_1 1 113 + 1 -------UGUCCAGCUAACAAAAAGCCCCUUAAAAGGGGCUCCGCUAAAACUGGUUGCGGGGGCCGGAUUUGAACCGACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCC -------.(((............(((((((....)))))))((((...........))))(((((((.......)))..((((....))))....)))))))((((.........)))). ( -41.90) >consensus _______UGUCCAGCUAACAAAAAGCCCCUAAAAAGGGGCUCCG_UAAAACUGGUUGCGGGGGCCGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCC ............(((.........((((((....))))))..........(((((.((((..(.(((.......))).)..)).((((((.....))))))...)).)))))...))).. (-38.55 = -38.88 + 0.33)

| Location | 5,332,038 – 5,332,157 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.52 |
| Mean single sequence MFE | -46.23 |
| Consensus MFE | -43.81 |
| Energy contribution | -43.20 |
| Covariance contribution | -0.61 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.53 |
| Structure conservation index | 0.95 |
| SVM decision value | 3.34 |
| SVM RNA-class probability | 0.999043 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 5332038 119 - 1 GGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUU-CAGGGCCCCUUUUUAGGGGCUUUUUGUUAGCUGGACAGUCCAUG (((.(.(((((((.((...(((((.......))))).((..(((((.......)))))..)))).)))((((...-.(((((((((....))))))))).....)))))))).))))... ( -45.60) >pau_1 1 119 - 1 GGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUU-CAGGGCCCCUUUUUAGGGGCUUUUUGUUAGCUGGACAGUCCAUG (((.(.(((((((.((...(((((.......))))).((..(((((.......)))))..)))).)))((((...-.(((((((((....))))))))).....)))))))).))))... ( -45.60) >pel_1 1 107 - 1 GGAGCAGCUUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGCUUCAGCAGAGCCCCUUUUCAGGGGCUUUUUGCUAGC------------- (((((....((((.((...(((((.......))))).((..(((((.......)))))..)))).)))))))))((((((((((((....))))))))..))))...------------- ( -46.40) >pf5_1 1 113 - 1 GGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUCGGUUCAAAUCCGGCCCCCGCAACCAGUUUUAGCGGAGCCCCUUUUCAGGGGCUUUUUGUUAGCUGGACA------- ....(((((((((.((...(((((.......))))).((..(((((.......)))))..)))).)))))...(((((((((((((....))))))))..)))))))))....------- ( -46.20) >pfo_1 1 111 - 1 GGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUCGGUUCAAAUCCGGCCCCCGCAACCAGUUUAA--GGAGCCCCUUUUAAGGGGCUUUUUGUUAGCUGGACA------- ....(((((((((.((...(((((.......))))).((..(((((.......)))))..)))).)))))..(((--(((((((((....))))))))))))...))))....------- ( -47.20) >pfs_1 1 113 - 1 GGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUCGGUUCAAAUCCGGCCCCCGCAACCAGUUUUAGCGGAGCCCCUUUUAAGGGGCUUUUUGUUAGCUGGACA------- ....(((((((((.((...(((((.......))))).((..(((((.......)))))..)))).)))))...(((((((((((((....))))))))..)))))))))....------- ( -46.40) >consensus GGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUCGGUUCAAAUCCAGCCCCCGCAACCAGUUUUA_CAGAGCCCCUUUUAAGGGGCUUUUUGUUAGCUGGACA_______ .((((.........)))).(((((.......))))).((..(((((.......)))))..))....((((((.....(((((((((....))))))))).....)))))).......... (-43.81 = -43.20 + -0.61)

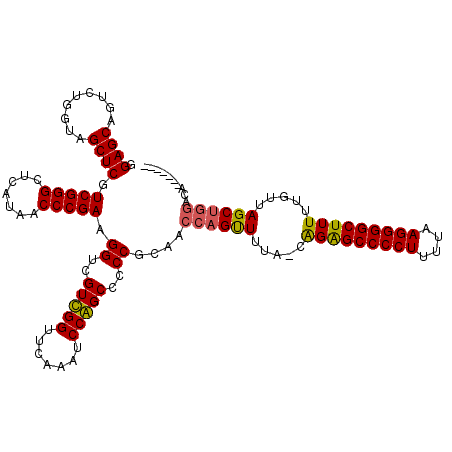
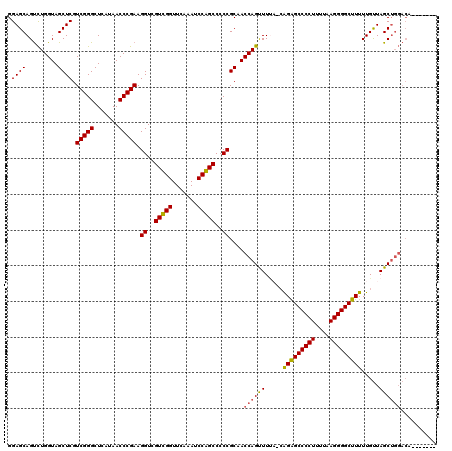
| Location | 5,332,056 – 5,332,175 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.05 |
| Mean single sequence MFE | -47.27 |
| Consensus MFE | -45.03 |
| Energy contribution | -45.42 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.95 |
| SVM decision value | 4.11 |
| SVM RNA-class probability | 0.999800 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
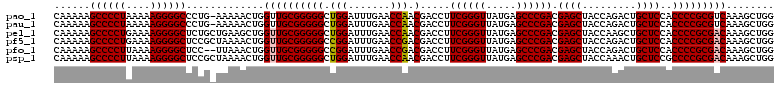
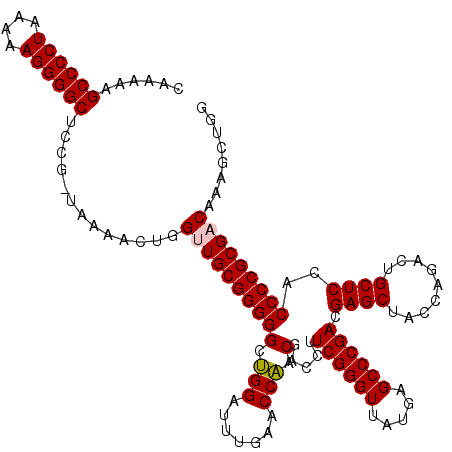
>pao_1 5332056 119 + 1 CAAAAAGCCCCUAAAAAGGGGCCCUG-AAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCCACCCCGCGUCAAAGCUGG ......((((((....))))))....-.....(..((((((((((.(((.......))).).....((((((.....)))))).((((.........))))..))))))).....))..) ( -42.30) >pau_1 19 119 + 1 CAAAAAGCCCCUAAAAAGGGGCCCUG-AAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCCACCCCGCGUCAAAGCUGG ......((((((....))))))....-.....(..((((((((((.(((.......))).).....((((((.....)))))).((((.........))))..))))))).....))..) ( -42.30) >pel_1 6 120 + 1 CAAAAAGCCCCUGAAAAGGGGCUCUGCUGAAGCUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAAGCUGCUCCACCCCGCGACAAAGCUGG .....(((((((....))))))).......((((.(((((((((..((((..............(.((((((.....)))))).)((((....))))..)))))))))))))..)))).. ( -53.30) >pf5_1 12 120 + 1 CAAAAAGCCCCUGAAAAGGGGCUCCGCUAAAACUGGUUGCGGGGGCCGGAUUUGAACCGACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCCACCCCGCGACAAAGCUGG .....(((((((....)))))))(((((.......(((((((((..(((.......))).......((((((.....)))))).((((.........))))..)))))))))..))).)) ( -51.10) >pfo_1 12 118 + 1 CAAAAAGCCCCUUAAAAGGGGCUCC--UUAAACUGGUUGCGGGGGCCGGAUUUGAACCGACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCCACCCCGCGACAAAGCUGG .....(((((((....)))))))..--........(((((((((..(((.......))).......((((((.....)))))).((((.........))))..)))))))))........ ( -47.20) >psp_1 19 120 + 1 CAAAAAGCCCCUUAAAAGGGGCUCCGCUAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAAACUGCUCCGCCCCGCGACAAAGCUGG .....(((((((....)))))))(((((.......(((((((((..(((.......))).((....((((((.....)))))).((((.........)))))))))))))))..))).)) ( -47.40) >consensus CAAAAAGCCCCUAAAAAGGGGCUCCG_UAAAACUGGUUGCGGGGGCUGGAUUUGAACCAACGACCUUCGGGUUAUGAGCCCGACGAGCUACCAGACUGCUCCACCCCGCGACAAAGCUGG ......((((((....)))))).............((((((((((.(((.......))).).....((((((.....)))))).((((.........))))..)))))))))........ (-45.03 = -45.42 + 0.39)
| Location | 5,332,056 – 5,332,175 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.05 |
| Mean single sequence MFE | -48.38 |
| Consensus MFE | -46.29 |
| Energy contribution | -45.57 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.96 |
| SVM decision value | 2.78 |
| SVM RNA-class probability | 0.996968 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
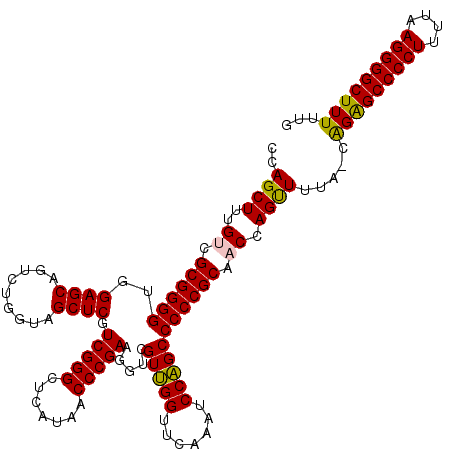
>pao_1 5332056 119 - 1 CCAGCUUUGACGCGGGGUGGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUU-CAGGGCCCCUUUUUAGGGGCUUUUUG ...........((((((((((.....(..(..(((..(((((.......))))).)))..)..)......))))..))))))...........-.(((((((((....)))))))))... ( -45.60) >pau_1 19 119 - 1 CCAGCUUUGACGCGGGGUGGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUU-CAGGGCCCCUUUUUAGGGGCUUUUUG ...........((((((((((.....(..(..(((..(((((.......))))).)))..)..)......))))..))))))...........-.(((((((((....)))))))))... ( -45.60) >pel_1 6 120 - 1 CCAGCUUUGUCGCGGGGUGGAGCAGCUUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGCUUCAGCAGAGCCCCUUUUCAGGGGCUUUUUG ...((((..((....))..))))((((.(((.((...(((((.......))))).((..(((((.......)))))..)))).))))))).(((.(((((((((....)))))))))))) ( -49.50) >pf5_1 12 120 - 1 CCAGCUUUGUCGCGGGGUGGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUCGGUUCAAAUCCGGCCCCCGCAACCAGUUUUAGCGGAGCCCCUUUUCAGGGGCUUUUUG ((.((((..((....))..))))...(((((.((...(((((.......))))).((..(((((.......)))))..)))).))))).......))(((((((....)))))))..... ( -49.40) >pfo_1 12 118 - 1 CCAGCUUUGUCGCGGGGUGGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUCGGUUCAAAUCCGGCCCCCGCAACCAGUUUAA--GGAGCCCCUUUUAAGGGGCUUUUUG ...((((..((....))..))))...(((((.((...(((((.......))))).((..(((((.......)))))..)))).)))))..(((--(((((((((....)))))))))))) ( -51.80) >psp_1 19 120 - 1 CCAGCUUUGUCGCGGGGCGGAGCAGUUUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUAGCGGAGCCCCUUUUAAGGGGCUUUUUG ((.((((((((....))))))))...(((((.((...(((((.......))))).((..(((((.......)))))..)))).))))).......))(((((((....)))))))..... ( -48.40) >consensus CCAGCUUUGUCGCGGGGUGGAGCAGUCUGGUAGCUCGUCGGGCUCAUAACCCGAAGGUCGUUGGUUCAAAUCCAGCCCCCGCAACCAGUUUUA_CAGAGCCCCUUUUAAGGGGCUUUUUG ..((((..((.((((((..((((.........)))).(((((.......))))).....(((((.......))))))))))).)).)))).....(((((((((....)))))))))... (-46.29 = -45.57 + -0.72)

Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 11:53:23 2007