
| Sequence ID | pao_1 |
|---|---|
| Location | 5,087,097 – 5,087,176 |
| Length | 79 |
| Max. P | 0.999991 |

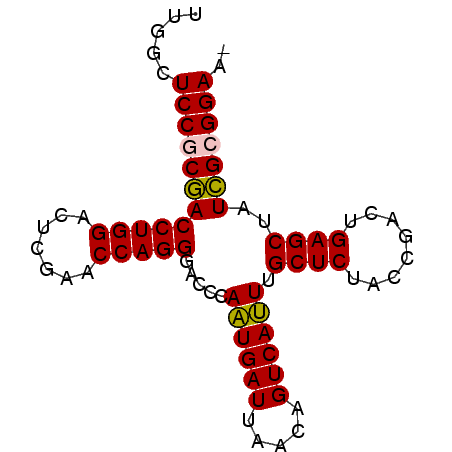
| Location | 5,087,097 – 5,087,176 |
|---|---|
| Length | 79 |
| Sequences | 6 |
| Columns | 79 |
| Reading direction | forward |
| Mean pairwise identity | 95.00 |
| Mean single sequence MFE | -26.77 |
| Consensus MFE | -27.02 |
| Energy contribution | -26.63 |
| Covariance contribution | -0.39 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.64 |
| Structure conservation index | 1.01 |
| SVM decision value | 5.62 |
| SVM RNA-class probability | 0.999991 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
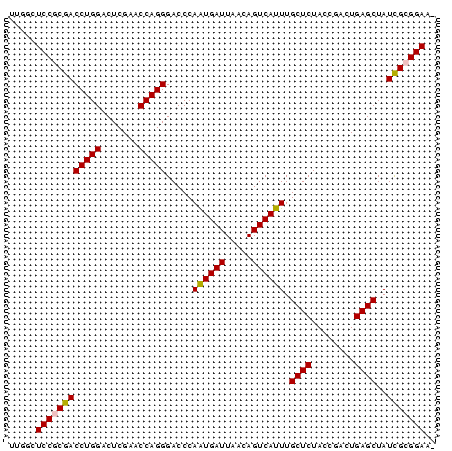
>pao_1 5087097 79 + 1 UUGGCUCCGCGACCUGGACUCGAACCAGGGACCCAAUGAUUAACAGUCAUUUGCUCUACCGACUGAGCUAUCGCGGAAC .....((((((((((((.......))))).....((((((.....)))))).((((........))))..))))))).. ( -28.10) >pau_1 1 79 + 1 UUGGCUCCGCGACCUGGACUCGAACCAGGGACCCAAUGAUUAACAGUCAUUUGCUCUACCGACUGAGCUAUCGCGGAAC .....((((((((((((.......))))).....((((((.....)))))).((((........))))..))))))).. ( -28.10) >pel_1 1 78 + 1 UUGGCUCCGCGACCUGGACUCGAACCAGGGACCCAAUGAUUAACAGUCAUUUGCUCUACCGACUGAGCUAUCGCGGAA- .....((((((((((((.......))))).....((((((.....)))))).((((........))))..))))))).- ( -28.10) >pf5_1 1 78 + 1 AUGGCUCCACGACCUGGACUCGAACCAGGGACCCAGUGAUUAACAGUCACUUGCUCUACCAACUGAGCUAUCGCGGAA- .....(((.((((((((.......))))).....((((((.....)))))).((((........))))..))).))).- ( -25.00) >pfo_1 1 78 + 1 UUGGCUCCACAACCUGGACUCGAACCAGGGACCCAGUGAUUAACAGUCACUUGCUCUACCAACUGAGCUAUUGCGGAA- .....(((.((((((((.......))))).....((((((.....)))))).((((........))))..))).))).- ( -23.20) >ppk_1 1 77 + 1 -UGGCUCCGCGACCUGGACUCGAACCAGGGACCCAAUGAUUAACAGUCAUUUGCUCUACCGACUGAGCUAUCGCGGAA- -....((((((((((((.......))))).....((((((.....)))))).((((........))))..))))))).- ( -28.10) >consensus UUGGCUCCGCGACCUGGACUCGAACCAGGGACCCAAUGAUUAACAGUCAUUUGCUCUACCGACUGAGCUAUCGCGGAA_ .....((((((((((((.......))))).....((((((.....)))))).((((........))))..))))))).. (-27.02 = -26.63 + -0.39)

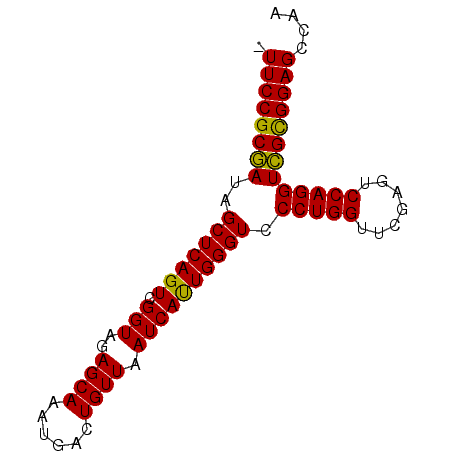
| Location | 5,087,097 – 5,087,176 |
|---|---|
| Length | 79 |
| Sequences | 6 |
| Columns | 79 |
| Reading direction | reverse |
| Mean pairwise identity | 95.00 |
| Mean single sequence MFE | -29.33 |
| Consensus MFE | -29.02 |
| Energy contribution | -28.30 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.99 |
| SVM decision value | 4.66 |
| SVM RNA-class probability | 0.999935 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
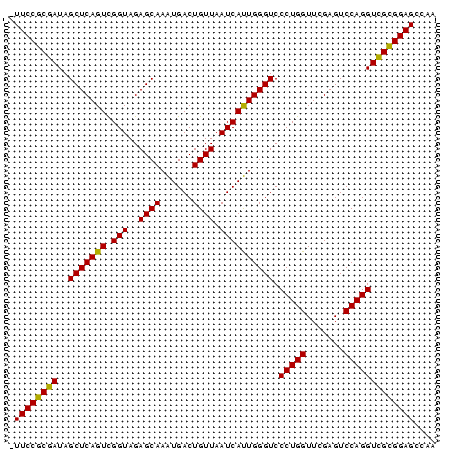
>pao_1 5087097 79 - 1 GUUCCGCGAUAGCUCAGUCGGUAGAGCAAAUGACUGUUAAUCAUUGGGUCCCUGGUUCGAGUCCAGGUCGCGGAGCCAA (((((((((..(((((((.(((..((((......)))).)))))))))).(((((.......))))))))))))))... ( -31.70) >pau_1 1 79 - 1 GUUCCGCGAUAGCUCAGUCGGUAGAGCAAAUGACUGUUAAUCAUUGGGUCCCUGGUUCGAGUCCAGGUCGCGGAGCCAA (((((((((..(((((((.(((..((((......)))).)))))))))).(((((.......))))))))))))))... ( -31.70) >pel_1 1 78 - 1 -UUCCGCGAUAGCUCAGUCGGUAGAGCAAAUGACUGUUAAUCAUUGGGUCCCUGGUUCGAGUCCAGGUCGCGGAGCCAA -((((((((..(((((((.(((..((((......)))).)))))))))).(((((.......))))))))))))).... ( -28.40) >pf5_1 1 78 - 1 -UUCCGCGAUAGCUCAGUUGGUAGAGCAAGUGACUGUUAAUCACUGGGUCCCUGGUUCGAGUCCAGGUCGUGGAGCCAU -((((((((..(((((((.(((..((((......)))).)))))))))).(((((.......))))))))))))).... ( -28.80) >pfo_1 1 78 - 1 -UUCCGCAAUAGCUCAGUUGGUAGAGCAAGUGACUGUUAAUCACUGGGUCCCUGGUUCGAGUCCAGGUUGUGGAGCCAA -((((((((..(((((((.(((..((((......)))).)))))))))).(((((.......))))))))))))).... ( -27.00) >ppk_1 1 77 - 1 -UUCCGCGAUAGCUCAGUCGGUAGAGCAAAUGACUGUUAAUCAUUGGGUCCCUGGUUCGAGUCCAGGUCGCGGAGCCA- -((((((((..(((((((.(((..((((......)))).)))))))))).(((((.......)))))))))))))...- ( -28.40) >consensus _UUCCGCGAUAGCUCAGUCGGUAGAGCAAAUGACUGUUAAUCAUUGGGUCCCUGGUUCGAGUCCAGGUCGCGGAGCCAA .((((((((..(((((((.(((..((((......)))).)))))))))).(((((.......))))))))))))).... (-29.02 = -28.30 + -0.72)

Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 11:51:11 2007