
| Sequence ID | pao_1 |
|---|---|
| Location | 4,785,556 – 4,785,677 |
| Length | 121 |
| Max. P | 0.999965 |

| Location | 4,785,556 – 4,785,676 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.52 |
| Mean single sequence MFE | -47.57 |
| Consensus MFE | -46.95 |
| Energy contribution | -46.73 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.06 |
| Mean z-score | -5.96 |
| Structure conservation index | 0.99 |
| SVM decision value | 3.96 |
| SVM RNA-class probability | 0.999729 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 4785556 120 + 1 AACAAGGGCAGAUAUUUUCAUAUCUGCCCCUGGAUAAAUGGAGCUCAUGAGCGGAUUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAUGAGCGAAACA ..((.((((((((((....)))))))))).))..........((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))...... ( -50.00) >pst_1 1 118 + 1 AACAAAGGCAGAUAUUUUCAUAUCUGCCUUUAG-CA-AUGGAGCUCUUGAGCGGAUUCGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAAGAGCAAAACA ...((((((((((((....))))))))))))..-..-.....((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))...... ( -48.10) >pau_1 1 120 + 1 AACAAGGGCAGAUAUUUUCAUAUCUGCCCCUGGAUAAAUGGAGCUCAUGAGCGGAUUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAUGAGCGAAACA ..((.((((((((((....)))))))))).))..........((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))...... ( -50.00) >ppk_1 1 119 + 1 AACAAAGGCAGAUAUUUUCAUAUCUGCCUUGUU-UAUAUGGAGCUCUUGAGCGGACUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAAGAGCGAAACA (((((.(((((((((....))))))))))))))-........((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))...... ( -47.70) >psp_1 1 118 + 1 AACAAAGGCAGAUAUUUUCAUAUCUUCCUUUAG-CA-AUGGAGCUCUUGAGCGGAUUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAAGAGCAAAACA ...(((((.((((((....)))))).)))))..-..-.....((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))...... ( -41.50) >pss_1 1 118 + 1 AACAAAGGCAGAUAUUUUCAUAUCUGCCUUUAU-CA-AUGGAGCUCUUGAGCGGAUUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAAGAGCAAAACA ...((((((((((((....))))))))))))..-..-.....((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))...... ( -48.10) >consensus AACAAAGGCAGAUAUUUUCAUAUCUGCCUUUAG_CA_AUGGAGCUCUUGAGCGGAUUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAAGAGCAAAACA ...((((((((((((....))))))))))))...........((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))...... (-46.95 = -46.73 + -0.22)

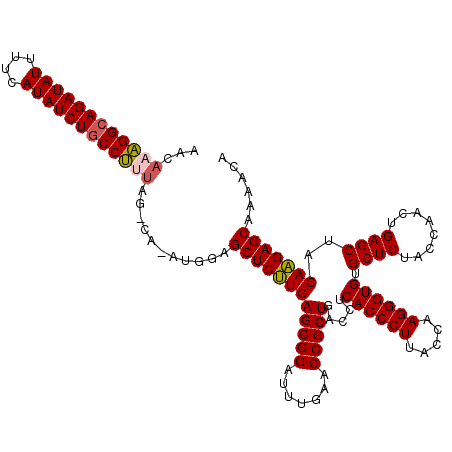
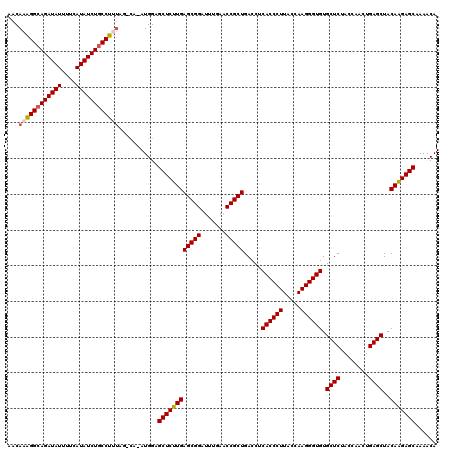
| Location | 4,785,556 – 4,785,676 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.52 |
| Mean single sequence MFE | -51.43 |
| Consensus MFE | -51.78 |
| Energy contribution | -51.00 |
| Covariance contribution | -0.78 |
| Combinations/Pair | 1.11 |
| Mean z-score | -5.54 |
| Structure conservation index | 1.01 |
| SVM decision value | 4.51 |
| SVM RNA-class probability | 0.999912 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 4785556 120 - 1 UGUUUCGCUCAUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAAUCCGCUCAUGAGCUCCAUUUAUCCAGGGGCAGAUAUGAAAAUAUCUGCCCUUGUU ......(((((((..((((........)))).((((((.....)))))).....(((((.......))))))))))))..........(((((((((((((....))))))))))))).. ( -55.20) >pst_1 1 118 - 1 UGUUUUGCUCUUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCGAAUCCGCUCAAGAGCUCCAU-UG-CUAAAGGCAGAUAUGAAAAUAUCUGCCUUUGUU ......(((((((..((((........)))).((((((.....)))))).....(((((.......)))))))))))).....-..-.(((((((((((((....))))))))))))).. ( -50.80) >pau_1 1 120 - 1 UGUUUCGCUCAUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAAUCCGCUCAUGAGCUCCAUUUAUCCAGGGGCAGAUAUGAAAAUAUCUGCCCUUGUU ......(((((((..((((........)))).((((((.....)))))).....(((((.......))))))))))))..........(((((((((((((....))))))))))))).. ( -55.20) >ppk_1 1 119 - 1 UGUUUCGCUCUUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAGUCCGCUCAAGAGCUCCAUAUA-AACAAGGCAGAUAUGAAAAUAUCUGCCUUUGUU ......(((((((..((((........)))).((((((.....)))))).....(((((.......))))))))))))........-((((((((((((((....))))))))).))))) ( -50.20) >psp_1 1 118 - 1 UGUUUUGCUCUUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAAUCCGCUCAAGAGCUCCAU-UG-CUAAAGGAAGAUAUGAAAAUAUCUGCCUUUGUU ......(((((((..((((........)))).((((((.....)))))).....(((((.......)))))))))))).....-..-.((((((.((((((....)))))).)))))).. ( -44.20) >pss_1 1 118 - 1 UGUUUUGCUCUUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAAUCCGCUCAAGAGCUCCAU-UG-AUAAAGGCAGAUAUGAAAAUAUCUGCCUUUGUU ......(((((((..((((........)))).((((((.....)))))).....(((((.......)))))))))))).....-.(-((((((((((((((....))))))))))))))) ( -53.00) >consensus UGUUUCGCUCUUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAAUCCGCUCAAGAGCUCCAU_UA_CUAAAGGCAGAUAUGAAAAUAUCUGCCUUUGUU ......(((((((..((((........)))).((((((.....)))))).....(((((.......))))))))))))..........(((((((((((((....))))))))))))).. (-51.78 = -51.00 + -0.78)

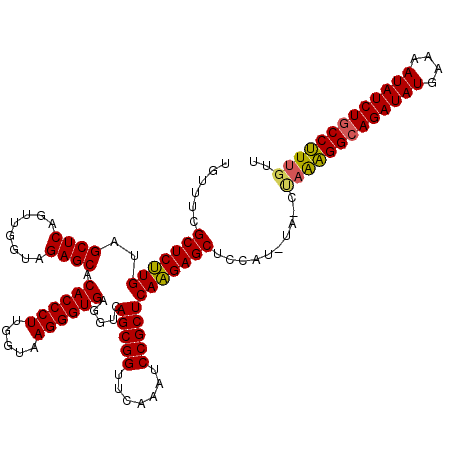
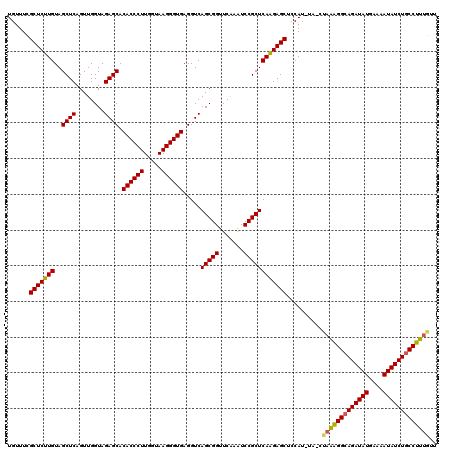
| Location | 4,785,557 – 4,785,677 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.52 |
| Mean single sequence MFE | -47.48 |
| Consensus MFE | -46.95 |
| Energy contribution | -46.73 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.06 |
| Mean z-score | -5.82 |
| Structure conservation index | 0.99 |
| SVM decision value | 4.13 |
| SVM RNA-class probability | 0.999810 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 4785557 120 + 1 ACAAGGGCAGAUAUUUUCAUAUCUGCCCCUGGAUAAAUGGAGCUCAUGAGCGGAUUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAUGAGCGAAACAC .((.((((((((((....)))))))))).))..........((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))....... ( -50.00) >pst_1 2 118 + 1 ACAAAGGCAGAUAUUUUCAUAUCUGCCUUUAG-CA-AUGGAGCUCUUGAGCGGAUUCGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAAGAGCAAAACAC ..((((((((((((....))))))))))))..-..-.....((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))....... ( -48.10) >pau_1 2 120 + 1 ACAAGGGCAGAUAUUUUCAUAUCUGCCCCUGGAUAAAUGGAGCUCAUGAGCGGAUUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAUGAGCGAAACAC .((.((((((((((....)))))))))).))..........((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))....... ( -50.00) >ppk_1 2 119 + 1 ACAAAGGCAGAUAUUUUCAUAUCUGCCUUGUU-UAUAUGGAGCUCUUGAGCGGACUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAAGAGCGAAACAC ...(((((((((((....)))))))))))...-........((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))....... ( -47.20) >psp_1 2 118 + 1 ACAAAGGCAGAUAUUUUCAUAUCUUCCUUUAG-CA-AUGGAGCUCUUGAGCGGAUUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAAGAGCAAAACAC ..(((((.((((((....)))))).)))))..-..-.....((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))....... ( -41.50) >pss_1 2 118 + 1 ACAAAGGCAGAUAUUUUCAUAUCUGCCUUUAU-CA-AUGGAGCUCUUGAGCGGAUUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAAGAGCAAAACAC ..((((((((((((....))))))))))))..-..-.....((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))....... ( -48.10) >consensus ACAAAGGCAGAUAUUUUCAUAUCUGCCUUUAG_CA_AUGGAGCUCUUGAGCGGAUUUGAACCGCUGACCUCACCCUUACCAAGGGUGUGCUCUACCAACUGAGCUACAAGAGCAAAACAC ..((((((((((((....))))))))))))...........((((((((((((.......))))).....((((((.....)))))).((((........))))..)))))))....... (-46.95 = -46.73 + -0.22)

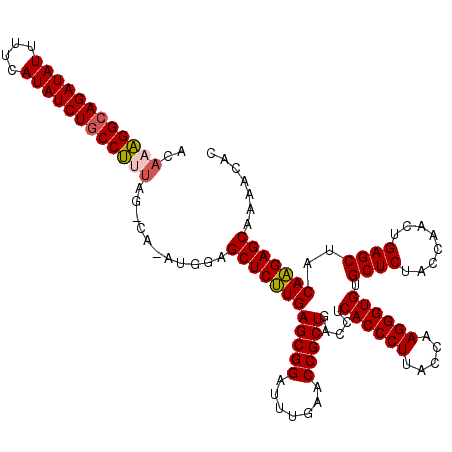
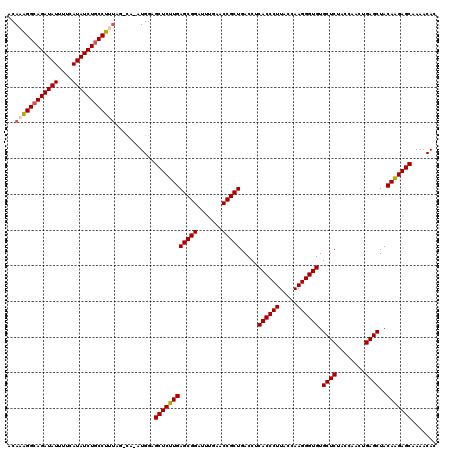
| Location | 4,785,557 – 4,785,677 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.52 |
| Mean single sequence MFE | -51.10 |
| Consensus MFE | -51.78 |
| Energy contribution | -51.00 |
| Covariance contribution | -0.78 |
| Combinations/Pair | 1.11 |
| Mean z-score | -5.20 |
| Structure conservation index | 1.01 |
| SVM decision value | 4.96 |
| SVM RNA-class probability | 0.999965 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 4785557 120 - 1 GUGUUUCGCUCAUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAAUCCGCUCAUGAGCUCCAUUUAUCCAGGGGCAGAUAUGAAAAUAUCUGCCCUUGU .......(((((((..((((........)))).((((((.....)))))).....(((((.......))))))))))))..........(((((((((((((....))))))))))))). ( -55.20) >pst_1 2 118 - 1 GUGUUUUGCUCUUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCGAAUCCGCUCAAGAGCUCCAU-UG-CUAAAGGCAGAUAUGAAAAUAUCUGCCUUUGU .......(((((((..((((........)))).((((((.....)))))).....(((((.......)))))))))))).....-..-.(((((((((((((....))))))))))))). ( -50.80) >pau_1 2 120 - 1 GUGUUUCGCUCAUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAAUCCGCUCAUGAGCUCCAUUUAUCCAGGGGCAGAUAUGAAAAUAUCUGCCCUUGU .......(((((((..((((........)))).((((((.....)))))).....(((((.......))))))))))))..........(((((((((((((....))))))))))))). ( -55.20) >ppk_1 2 119 - 1 GUGUUUCGCUCUUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAGUCCGCUCAAGAGCUCCAUAUA-AACAAGGCAGAUAUGAAAAUAUCUGCCUUUGU .......(((((((..((((........)))).((((((.....)))))).....(((((.......))))))))))))........-.(((((((((((((....))))))))).)))) ( -49.30) >psp_1 2 118 - 1 GUGUUUUGCUCUUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAAUCCGCUCAAGAGCUCCAU-UG-CUAAAGGAAGAUAUGAAAAUAUCUGCCUUUGU .......(((((((..((((........)))).((((((.....)))))).....(((((.......)))))))))))).....-..-.((((((.((((((....)))))).)))))). ( -44.20) >pss_1 2 118 - 1 GUGUUUUGCUCUUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAAUCCGCUCAAGAGCUCCAU-UG-AUAAAGGCAGAUAUGAAAAUAUCUGCCUUUGU .......(((((((..((((........)))).((((((.....)))))).....(((((.......)))))))))))).....-..-((((((((((((((....)))))))))))))) ( -51.90) >consensus GUGUUUCGCUCUUGUAGCUCAGUUGGUAGAGCACACCCUUGGUAAGGGUGAGGUCAGCGGUUCAAAUCCGCUCAAGAGCUCCAU_UA_CUAAAGGCAGAUAUGAAAAUAUCUGCCUUUGU .......(((((((..((((........)))).((((((.....)))))).....(((((.......))))))))))))..........(((((((((((((....))))))))))))). (-51.78 = -51.00 + -0.78)

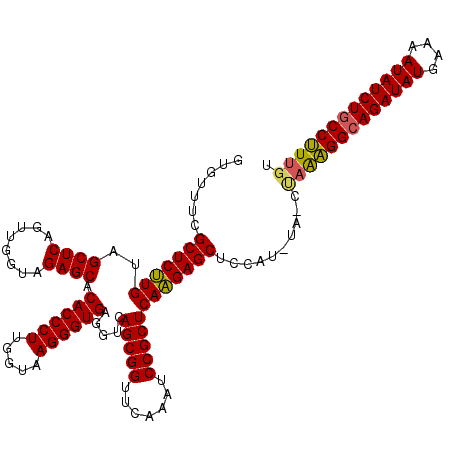
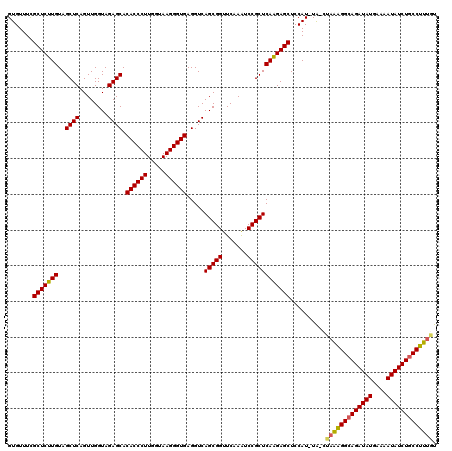
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 11:48:52 2007