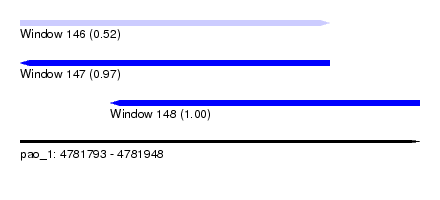
| Sequence ID | pao_1 |
|---|---|
| Location | 4,781,793 – 4,781,948 |
| Length | 155 |
| Max. P | 0.999696 |

| Location | 4,781,793 – 4,781,913 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.08 |
| Mean single sequence MFE | -46.35 |
| Consensus MFE | -38.05 |
| Energy contribution | -38.47 |
| Covariance contribution | 0.42 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.82 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.517912 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 4781793 120 + 1 ACUCCUGGAUGUUACCGUUUCGUCCAGCCGGGGCCGGACGUCGUUUUGGUGUCUGAUUCGGUAACCGAAUCGGGAGCACCAUCUGCGUAGGCUUGUGGUUUAAGGCUUGCGCCACCUACG ......((((.....((...(((((.((....)).))))).))....((((((((((((((...)))))))))..)))))))))((((((((((........))))))))))........ ( -48.00) >pau_1 1 120 + 1 ACUCCUGGAUGUUACCGUUUCGUCCAGCCGGGGCCGGACGUCGUUUUGGUGUCUGAUUCGGUAACCGAAUCGGGAGCACCAUCUGCGUAGGCUUGUGGUUUAAGGCUUGCGCCACCUACG ......((((.....((...(((((.((....)).))))).))....((((((((((((((...)))))))))..)))))))))((((((((((........))))))))))........ ( -48.00) >pel_1 1 119 + 1 ACUCCUGGUUUCUACCGUUUCAUCUGGCCGGAGCCGGAUGUCGUUUUGGUGUCUGAUUCGCGAAC-GAAUCGGGAGCACCAUCUGCGUAGGCUUGAGGUUUAAGGCUUGCGCCGCCUACG ......(((......((...((((((((....)))))))).))...((((((((((((((....)-)))))))..))))))...((((((((((........)))))))))).))).... ( -47.20) >pf5_1 1 119 + 1 ACUCCUGCUUGUUACCGUUUCAUCCAACCGGAGCUGGAUGUCGUUUUGGUGUCUGAUUCGGUAAG-GAACCGGGAGCACCAUCUGCGUAGGCUUGAGGUUUAAGACUUGCGUCGCCUACG .((((((.((.((((((..(((..((.(((((((........)))))))))..)))..)))))).-))..)))))).........(((((((.(((((((...))))).))..))))))) ( -43.20) >pfs_1 1 119 + 1 ACUCCUGCUUGUUACCGUUUCAUCUGGCCGGAGCCGGAUGUCGUUUUGGUGUCUGAUUCGGUAAG-GAACCGGGAGCACCAUCUGCGUAGGCUUGUGGUUUAAGACUUACGUCGCCUACG .((((((.((.((((((..(((....((((((((........))))))))...)))..)))))).-))..)))))).........(((((((.(((((((...))).))))..))))))) ( -41.40) >ppk_1 1 119 + 1 ACUCCUGGUUUUUACCGUUUCAUCUGGCCGAGGCCGGAUGUCGUUUUGGUGUCUGAUUCGGUAAC-GAAUCGGGAGCACCAUCUGCGUGGGCUGAUGUUUUAAGGCUUGCGCCGCCUACG ......(((....)))....((((((((....))))))))..((..((((((((((((((....)-)))))))..))))))...))(((((((((....))).(((....))))))))). ( -50.30) >consensus ACUCCUGGUUGUUACCGUUUCAUCCAGCCGGAGCCGGAUGUCGUUUUGGUGUCUGAUUCGGUAAC_GAAUCGGGAGCACCAUCUGCGUAGGCUUGUGGUUUAAGGCUUGCGCCGCCUACG .(((((((((.((((((..(((....((((((((........))))))))...)))..))))))...)))))))))........((((((((((........))))))))))........ (-38.05 = -38.47 + 0.42)

| Location | 4,781,793 – 4,781,913 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.08 |
| Mean single sequence MFE | -43.03 |
| Consensus MFE | -36.32 |
| Energy contribution | -34.63 |
| Covariance contribution | -1.69 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.76 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.58 |
| SVM RNA-class probability | 0.965326 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 4781793 120 - 1 CGUAGGUGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUCGGUUACCGAAUCAGACACCAAAACGACGUCCGGCCCCGGCUGGACGAAACGGUAACAUCCAGGAGU ((((((((((....)))...........)))))))....(((((.((..(((((((...))))))).)))))))......((((((((....))))))))....((......))...... ( -48.70) >pau_1 1 120 - 1 CGUAGGUGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUCGGUUACCGAAUCAGACACCAAAACGACGUCCGGCCCCGGCUGGACGAAACGGUAACAUCCAGGAGU ((((((((((....)))...........)))))))....(((((.((..(((((((...))))))).)))))))......((((((((....))))))))....((......))...... ( -48.70) >pel_1 1 119 - 1 CGUAGGCGGCGCAAGCCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGAUUC-GUUCGCGAAUCAGACACCAAAACGACAUCCGGCUCCGGCCAGAUGAAACGGUAGAAACCAGGAGU ((((((((((....)))...........)))))))....(((((.((..(((((-(....)))))).)))))))......((((.(((....))).))))....(((....)))...... ( -43.70) >pf5_1 1 119 - 1 CGUAGGCGACGCAAGUCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGGUUC-CUUACCGAAUCAGACACCAAAACGACAUCCAGCUCCGGUUGGAUGAAACGGUAACAAGCAGGAGU ((((((((((....)))...........)))))))........(((((..(((.-.((((((.....(....).......((((((((....))))))))...))))))..))).))))) ( -40.60) >pfs_1 1 119 - 1 CGUAGGCGACGUAAGUCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGGUUC-CUUACCGAAUCAGACACCAAAACGACAUCCGGCUCCGGCCAGAUGAAACGGUAACAAGCAGGAGU ((((((((((....)))...........)))))))........(((((..(((.-.((((((.....(....).......((((.(((....))).))))...))))))..))).))))) ( -37.40) >ppk_1 1 119 - 1 CGUAGGCGGCGCAAGCCUUAAAACAUCAGCCCACGCAGAUGGUGCUCCCGAUUC-GUUACCGAAUCAGACACCAAAACGACAUCCGGCCUCGGCCAGAUGAAACGGUAAAAACCAGGAGU (((.((((((....)))...........))).)))....(((((.((..(((((-(....)))))).)))))))......((((.(((....))).))))....(((....)))...... ( -39.10) >consensus CGUAGGCGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUC_GUUACCGAAUCAGACACCAAAACGACAUCCGGCCCCGGCCAGAUGAAACGGUAACAACCAGGAGU ((((((((((....)))...........)))))))....(((((.((..(((((.......))))).)))))))......((((.(((....))).)))).................... (-36.32 = -34.63 + -1.69)
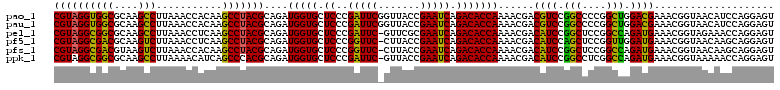
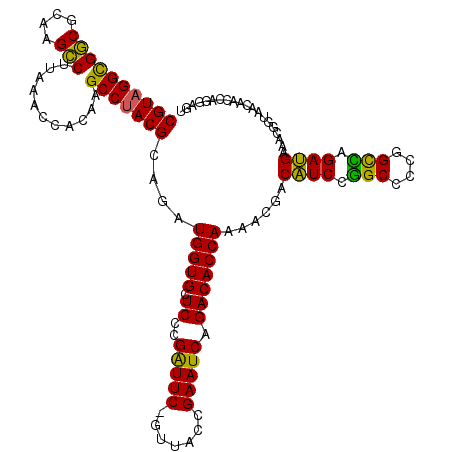
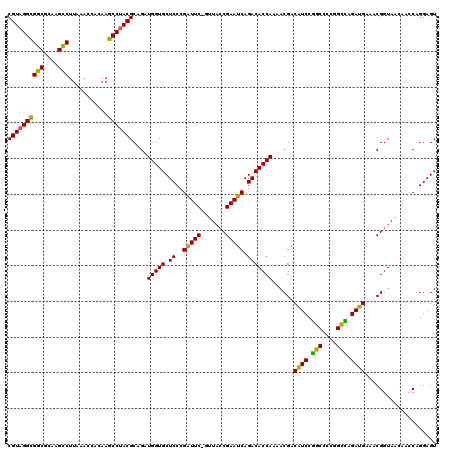
| Location | 4,781,828 – 4,781,948 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.65 |
| Mean single sequence MFE | -46.78 |
| Consensus MFE | -43.60 |
| Energy contribution | -42.97 |
| Covariance contribution | -0.64 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.66 |
| Structure conservation index | 0.93 |
| SVM decision value | 3.90 |
| SVM RNA-class probability | 0.999696 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 4781828 120 - 1 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGUGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUCGGUUACCGAAUCAGACACCAAAACGACGUCC .(((((((((((((...)))))))))..))))(((((((((((((....)))...........)))))))....(((((.((..(((((((...))))))).))))))).......))). ( -51.10) >pau_1 36 120 - 1 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGUGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUCGGUUACCGAAUCAGACACCAAAACGACGUCC .(((((((((((((...)))))))))..))))(((((((((((((....)))...........)))))))....(((((.((..(((((((...))))))).))))))).......))). ( -51.10) >pel_1 36 119 - 1 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGCCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGAUUC-GUUCGCGAAUCAGACACCAAAACGACAUCC .(((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((.((..(((((-(....)))))).)))))))........... ( -49.40) >pfo_1 1 115 - 1 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGUCUUAAACCUCAAGCCUACGCAGAUGGUGCUCCCGGUUC-CUUACCGAAUCAGACACCAAAACGAC---- .(((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((((..((((..-...))))....)).))))).......---- ( -42.90) >pfs_1 36 119 - 1 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGACGUAAGUCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGGUUC-CUUACCGAAUCAGACACCAAAACGACAUCC .(((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((((..((((..-...))))....)).)))))........... ( -42.50) >ppk_1 36 119 - 1 AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGCCUUAAAACAUCAGCCCACGCAGAUGGUGCUCCCGAUUC-GUUACCGAAUCAGACACCAAAACGACAUCC .(((((((((((((...)))))))))..))))...(((.((((((....)))...........))).)))....(((((.((..(((((-(....)))))).)))))))........... ( -43.70) >consensus AUUGGGGUCCCUGCCUGGCGGGGGCUAUCCAAGACCGUAGGCGGCGCAAGCCUUAAACCACAAGCCUACGCAGAUGGUGCUCCCGAUUC_GUUACCGAAUCAGACACCAAAACGACAUCC .(((((((((((((...)))))))))..))))...((((((((((....)))...........)))))))....(((((.((..(((((.......))))).)))))))........... (-43.60 = -42.97 + -0.64)

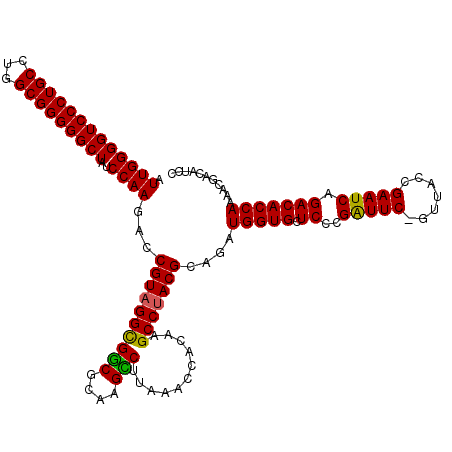
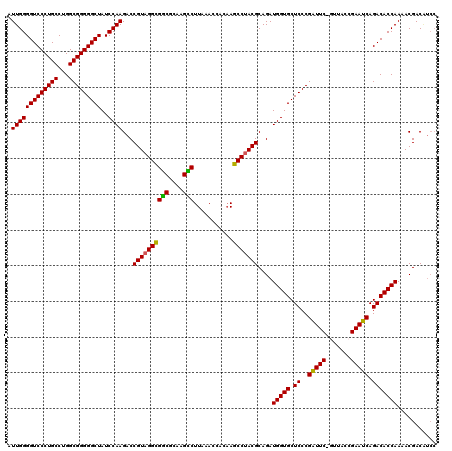
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 11:48:42 2007