
| Sequence ID | pao_1 |
|---|---|
| Location | 630,406 – 630,515 |
| Length | 109 |
| Max. P | 0.999973 |

| Location | 630,406 – 630,515 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 90.55 |
| Mean single sequence MFE | -45.18 |
| Consensus MFE | -42.77 |
| Energy contribution | -41.83 |
| Covariance contribution | -0.94 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.07 |
| Structure conservation index | 0.95 |
| SVM decision value | 4.18 |
| SVM RNA-class probability | 0.999828 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 630406 109 + 1 AAAGCCGCGCAGUGCGCGGCUUU-GAAGGUGGUGGGCCCACACGGACUCGAACCGUGGACCAAAGGAUUAUGAGUCCUCUGCUCUAACCGACUGAGCUAUAGGCCCCCAG ((((((((((...))))))))))-.....(((.(((((.....(((((((((((..........)).)).)))))))...((((.........))))....)))))))). ( -47.60) >pau_1 1 109 + 1 AAAGCCGCGCAAUGCGCGGCUUU-GAAGGUGGUGGGCCCACACGGACUCGAACCGUGGACCAAAGGAUUAUGAGUCCUCUGCUCUAACCGACUGAGCUAUAGGCCCCCAG ((((((((((...))))))))))-.....(((.(((((.....(((((((((((..........)).)).)))))))...((((.........))))....)))))))). ( -47.60) >pel_1 1 109 + 1 AAAGCCGCGCAAUGCGCGGCUUC-GAAGAUGGUGGGCCCACACGGACUCGAACCGUGGACCAAAGGAUUAUGAGUCCUCUGCUCUAACCGACUGAGCUAUAGGCCCUCAG .(((((((((...))))))))).-.....(((.(((((.....(((((((((((..........)).)).)))))))...((((.........))))....)))))))). ( -43.50) >pf5_1 1 106 + 1 AAAGCCGCGCAUUGCGCGGCUUUCUUGUUUGGUGGGCCCACACGGACUUGAACCGUGGACCAAAGGAUUAUGAGUCCUCUGCUCUAACCAACUGAGCUAUAGGCCC---- ((((((((((...))))))))))..........(((((..(((((.......)))))......(((((.....)))))..((((.........))))....)))))---- ( -41.20) >pfs_1 1 105 + 1 AAAGCCGCGCAAUGCGCGGCUUU-CAAGAUGGUGGGCCCAGUAGGACUCGAACCUACGACCAAGGGAUUAUGAGUCCCCUGCUCUAACCAACUGAGCUAUAGGCCC---- ((((((((((...))))))))))-.........(((((..(((((.......)))))......(((((.....)))))..((((.........))))....)))))---- ( -42.50) >pss_1 1 110 + 1 AAAGCCGCGCAAUGCGCGGCUUUCAAAGGUGGUGGGCCCACACGGACUCGAACCGUGGACCAAAGGAUUAUGAGUCCUCUGCUCUAACCAACUGAGCUAUGGGCCCUCAG ((((((((((...))))))))))......(((.(((((((...(((((((((((..........)).)).)))))))...((((.........))))..)))))))))). ( -48.70) >consensus AAAGCCGCGCAAUGCGCGGCUUU_GAAGGUGGUGGGCCCACACGGACUCGAACCGUGGACCAAAGGAUUAUGAGUCCUCUGCUCUAACCAACUGAGCUAUAGGCCC_CAG ((((((((((...))))))))))..........(((((..(((((.......)))))......(((((.....)))))..((((.........))))....))))).... (-42.77 = -41.83 + -0.94)

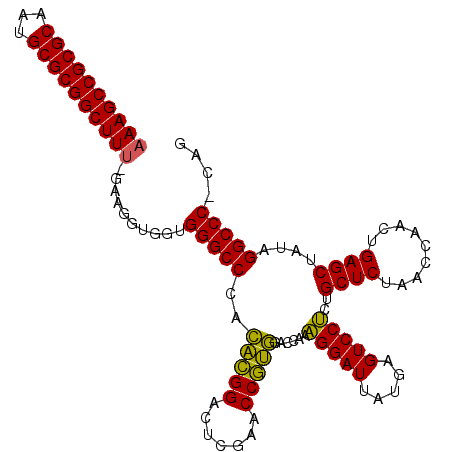
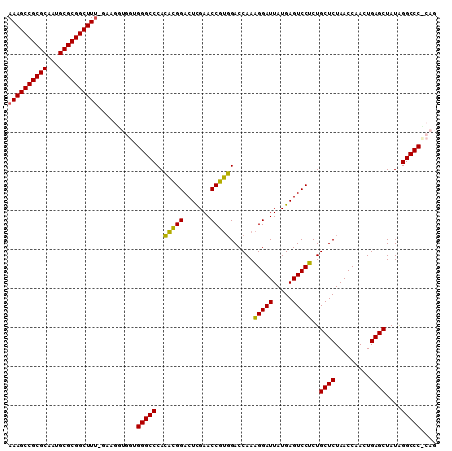
| Location | 630,406 – 630,515 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 90.55 |
| Mean single sequence MFE | -51.05 |
| Consensus MFE | -50.27 |
| Energy contribution | -48.88 |
| Covariance contribution | -1.38 |
| Combinations/Pair | 1.17 |
| Mean z-score | -4.14 |
| Structure conservation index | 0.98 |
| SVM decision value | 5.10 |
| SVM RNA-class probability | 0.999973 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>pao_1 630406 109 - 1 CUGGGGGCCUAUAGCUCAGUCGGUUAGAGCAGAGGACUCAUAAUCCUUUGGUCCACGGUUCGAGUCCGUGUGGGCCCACCACCUUC-AAAGCCGCGCACUGCGCGGCUUU .(((((((((((((((((..((....((.(((((((.......))))))).))..))..).))))...))))))))).))).....-((((((((((...)))))))))) ( -53.20) >pau_1 1 109 - 1 CUGGGGGCCUAUAGCUCAGUCGGUUAGAGCAGAGGACUCAUAAUCCUUUGGUCCACGGUUCGAGUCCGUGUGGGCCCACCACCUUC-AAAGCCGCGCAUUGCGCGGCUUU .(((((((((((((((((..((....((.(((((((.......))))))).))..))..).))))...))))))))).))).....-((((((((((...)))))))))) ( -53.20) >pel_1 1 109 - 1 CUGAGGGCCUAUAGCUCAGUCGGUUAGAGCAGAGGACUCAUAAUCCUUUGGUCCACGGUUCGAGUCCGUGUGGGCCCACCAUCUUC-GAAGCCGCGCAUUGCGCGGCUUU ....((((((((((((((..((....((.(((((((.......))))))).))..))..).))))...))))))))).........-((((((((((...)))))))))) ( -50.50) >pf5_1 1 106 - 1 ----GGGCCUAUAGCUCAGUUGGUUAGAGCAGAGGACUCAUAAUCCUUUGGUCCACGGUUCAAGUCCGUGUGGGCCCACCAAACAAGAAAGCCGCGCAAUGCGCGGCUUU ----((((((((((((.....)))))((.(((((((.......))))))).))(((((.......))))))))))))..........((((((((((...)))))))))) ( -48.20) >pfs_1 1 105 - 1 ----GGGCCUAUAGCUCAGUUGGUUAGAGCAGGGGACUCAUAAUCCCUUGGUCGUAGGUUCGAGUCCUACUGGGCCCACCAUCUUG-AAAGCCGCGCAUUGCGCGGCUUU ----((((((((((((.....)))))((.(((((((.......))))))).))(((((.......)))))))))))).........-((((((((((...)))))))))) ( -50.50) >pss_1 1 110 - 1 CUGAGGGCCCAUAGCUCAGUUGGUUAGAGCAGAGGACUCAUAAUCCUUUGGUCCACGGUUCGAGUCCGUGUGGGCCCACCACCUUUGAAAGCCGCGCAUUGCGCGGCUUU ....((((((((((((.....)))))((.(((((((.......))))))).))(((((.......))))))))))))..........((((((((((...)))))))))) ( -50.70) >consensus CUG_GGGCCUAUAGCUCAGUCGGUUAGAGCAGAGGACUCAUAAUCCUUUGGUCCACGGUUCGAGUCCGUGUGGGCCCACCACCUUC_AAAGCCGCGCAUUGCGCGGCUUU ....((((((((((((.....)))))((.(((((((.......))))))).))(((((.......))))))))))))..........((((((((((...)))))))))) (-50.27 = -48.88 + -1.38)

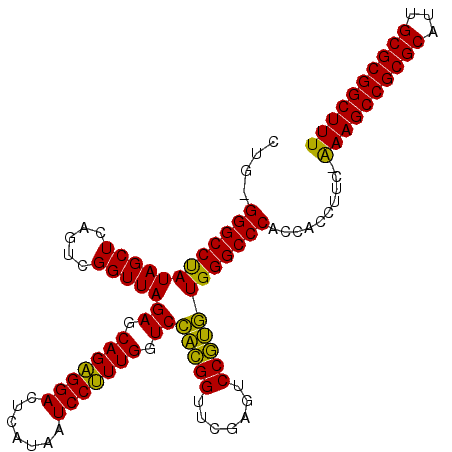
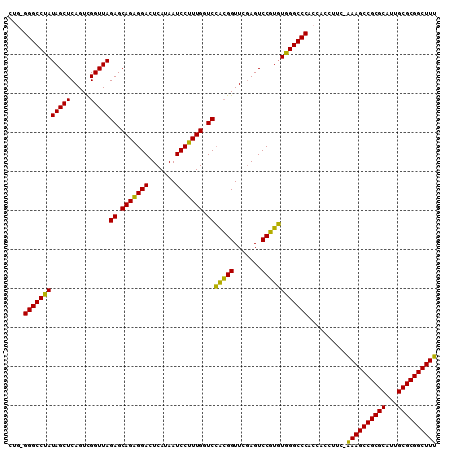
Generated by rnazCluster.pl (part of RNAz 1.0) on Thu Jan 18 11:45:31 2007