
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 19,653,244 – 19,653,352 |
| Length | 108 |
| Max. P | 0.533874 |

| Location | 19,653,244 – 19,653,352 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.30 |
| Mean single sequence MFE | -30.88 |
| Consensus MFE | -24.39 |
| Energy contribution | -24.37 |
| Covariance contribution | -0.03 |
| Combinations/Pair | 1.07 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.79 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.533874 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 19653244 108 + 27905053 UGUU-------GAUGACGACGUCAAAGUCGAAGAUGGGCAUGUGAUAAAAUAAAUUACGGGCAGAUUAGUGCGAAUUUAAUUAUCGUUUGUUCCUUUGAUGUCG-----CUAAGUAAAUA ....-------.....(((((((((((........(((((..((((((..((((((.((.((......)).)))))))).))))))..))))))))))))))))-----........... ( -28.80) >DroPse_CAF1 53662 113 + 1 UGCU-------GAUGACGACGUCAAAGUCGAAGAUGGGCAUGUGAUAAAAUAAAUUACGGCCAGAUUAGUGCGAAUUUAAUUAUCAUUUGUUCCUUUGAUGUCGCCGCGCAGAGUAAAUA (((.-------(.((.(((((((((((....((((((((.((((((.......))))))))).((((((.......))))))..)))))....))))))))))).))))))......... ( -31.50) >DroWil_CAF1 71511 110 + 1 UGUU-------GAUGACGACGUCAAAGCCGAAGAUGGGCAUGUGAUAAAAUAAAUUACGGCCAGAUUAGUGCGAAUUUAAUUAUCAUUUGUUCCUUUGAUGUCG---GGCAAAAUAAAUA ((((-------..((.(((((((((((....((((((((.((((((.......))))))))).((((((.......))))))..)))))....)))))))))))---..)).)))).... ( -30.20) >DroMoj_CAF1 43410 110 + 1 UGCU-------GAUGAGGACGUCAAAGUCGAAGAUGGGCAUGUGAUAAAAUAAAUUACGGGCAGAUUAGUGCGAAUUUAAUUAUCAUUUGUUCCUUUGAUGUAG---CUCUGGGUAAAUA ((((-------.(.(((.(((((((((........(((((.(((((((..((((((.((.((......)).)))))))).))))))).))))))))))))))..---)))).)))).... ( -32.10) >DroAna_CAF1 39380 117 + 1 UGUUGCUCCCUGAUGACGACGUCAAAGUCGAAGAUGGGUAUGUGAUAAAAUAAAUUACGGGCAGAUUAGUGCGAAUUUAAUUAUCGUUUGUUCCUUUGAUGUCG---CGCUAAGUAAAUA ..(((((....(.((.(((((((((((........(..((..((((((..((((((.((.((......)).)))))))).))))))..))..))))))))))))---)))..)))))... ( -29.10) >DroPer_CAF1 53137 118 + 1 UGCUGA--GCUGAUGACGACGUCAAAGUCGAAGAUGGGCAUGUGAUAAAAUAAAUUACGGCCAGAUUAGUGCGAAUUUAAUUAUCAUUUGUUCCUUUGAUGUCGCCGCGCAGAGUAAAUA ((((..--((.(.((.(((((((((((....((((((((.((((((.......))))))))).((((((.......))))))..)))))....))))))))))).)))))..)))).... ( -33.60) >consensus UGCU_______GAUGACGACGUCAAAGUCGAAGAUGGGCAUGUGAUAAAAUAAAUUACGGCCAGAUUAGUGCGAAUUUAAUUAUCAUUUGUUCCUUUGAUGUCG___CGCAAAGUAAAUA ................(((((((((((........(..((..((((((..((((((.((..(......)..)))))))).))))))..))..))))))))))))................ (-24.39 = -24.37 + -0.03)

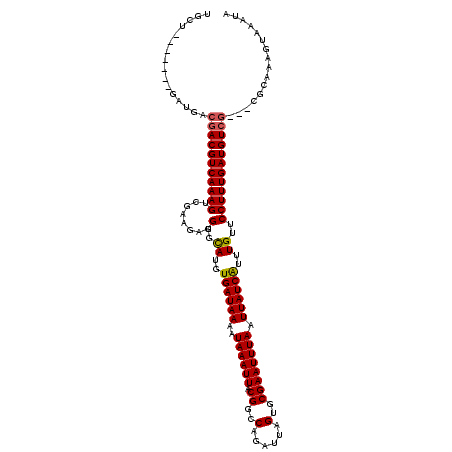
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:44:56 2006