| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 1,541,526 – 1,541,616 |
| Length | 90 |
| Max. P | 0.989640 |

| Location | 1,541,526 – 1,541,616 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 78.24 |
| Mean single sequence MFE | -50.55 |
| Consensus MFE | -32.10 |
| Energy contribution | -32.63 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.28 |
| Structure conservation index | 0.64 |
| SVM decision value | 2.17 |
| SVM RNA-class probability | 0.989640 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 1541526 90 + 27905053 GGACGCCCGACAACA---GCAGCUUCUCGGAGCUGGCGUUGGUGU-G--------------------GCCCGAGGAGCUGCAGCUGGUGAGGCUGGACAGCGGCUUAUCAUCGU ((....)).......---((((((((((((.((((((....)).)-)--------------------))))))))))))))....((((((((((.....)))))..))))).. ( -38.90) >DroVir_CAF1 52256 114 + 1 GUAUGCCGGACAACAGGAGCAGCUUCUCGGUGCUGGCGCUGGUGCUAGCAUCAUUGGUGCUGGCCAGGCCCGAGGAGCUGCAGCUGUUCAAGCUCGACAGCGGCUUGUCGUCCU .......((((.(((((.((((((((((((..(((((((....))((((((.....)))))))))))..)))))))))))).((((((.......))))))..))))).)))). ( -62.50) >DroPse_CAF1 42301 99 + 1 GCACGCCCGACAUCACCAGCAGCUUCUCGGAGCUGGCAUUGGUGC-U--------------GACCAGCCCCGAGGAGCUGCAGCUGUUCAGGCUGGACAGCGGCUUGUCAUCGU ..(((...((((...((.((((((((((((.(((((((.......-)--------------).))))).)))))))))))).(((((((.....)))))))))..))))..))) ( -52.60) >DroYak_CAF1 38499 90 + 1 GAACGCCCGACAGGA---GCAGCUUCUCGGAGCUGGCAUUGGUGU-G--------------------GCCCGAGGAGCUGCAGCUGUUGAGGCUGGACAGCGGCUUGUCAUCGU ((..(((((((((..---((((((((((((.((((.((....)))-)--------------------))))))))))))))..)))))).)))..(((((....))))).)).. ( -46.10) >DroAna_CAF1 40921 111 + 1 GCACGCCCGACAGCAGCAGCAGCUUCUCGGAACUGGCAUUGGUGU-GGG--UGCUGUUGCUUGCCACGCCCGAGGAGCUGCAGCUGUGGAGGCUAGACAACGGCUUGUCAUCGU ....((((.(((((....((((((((((((..(.......)((((-((.--.((....))...)))))))))))))))))).))))).).)))..(((((....)))))..... ( -50.60) >DroPer_CAF1 43815 99 + 1 GCACGCCCGACAUCACCAGCAGCUUCUCGGAGCUGGCAUUGGUGC-U--------------GACCAGCCCCGAGGAGCUGCAGCUGUUCAGGCUGGACAGCGGCUUGUCAUCGU ..(((...((((...((.((((((((((((.(((((((.......-)--------------).))))).)))))))))))).(((((((.....)))))))))..))))..))) ( -52.60) >consensus GCACGCCCGACAACA_CAGCAGCUUCUCGGAGCUGGCAUUGGUGC_G______________G_CCAGGCCCGAGGAGCUGCAGCUGUUCAGGCUGGACAGCGGCUUGUCAUCGU ....(((...........((((((((((((.((..(((....)))......................)))))))))))))).((((((.......))))))))).......... (-32.10 = -32.63 + 0.53)

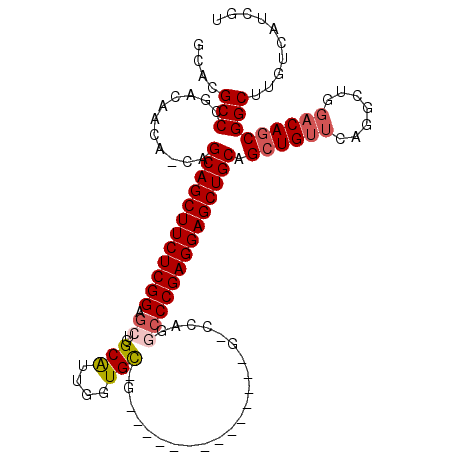
| Location | 1,541,526 – 1,541,616 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 78.24 |
| Mean single sequence MFE | -45.80 |
| Consensus MFE | -29.29 |
| Energy contribution | -30.96 |
| Covariance contribution | 1.67 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.19 |
| Structure conservation index | 0.64 |
| SVM decision value | 2.13 |
| SVM RNA-class probability | 0.988726 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 1541526 90 - 27905053 ACGAUGAUAAGCCGCUGUCCAGCCUCACCAGCUGCAGCUCCUCGGGC--------------------C-ACACCAACGCCAGCUCCGAGAAGCUGC---UGUUGUCGGGCGUCC ..(((((((......))))..((((...((((.((((((.(((((((--------------------.-............)).))))).))))))---.))))..))))))). ( -33.32) >DroVir_CAF1 52256 114 - 1 AGGACGACAAGCCGCUGUCGAGCUUGAACAGCUGCAGCUCCUCGGGCCUGGCCAGCACCAAUGAUGCUAGCACCAGCGCCAGCACCGAGAAGCUGCUCCUGUUGUCCGGCAUAC .((((..(((((((....)).)))))(((((..((((((.(((((..(((((.((((.......)))).((....)))))))..))))).))))))..)))))))))....... ( -54.30) >DroPse_CAF1 42301 99 - 1 ACGAUGACAAGCCGCUGUCCAGCCUGAACAGCUGCAGCUCCUCGGGGCUGGUC--------------A-GCACCAAUGCCAGCUCCGAGAAGCUGCUGGUGAUGUCGGGCGUGC (((.(((((.((((((((.(.....).))))).((((((.(((((((((((.(--------------(-.......))))))))))))).)))))).)))..)))))..))).. ( -53.30) >DroYak_CAF1 38499 90 - 1 ACGAUGACAAGCCGCUGUCCAGCCUCAACAGCUGCAGCUCCUCGGGC--------------------C-ACACCAAUGCCAGCUCCGAGAAGCUGC---UCCUGUCGGGCGUUC .....((((......))))..((((..((((..((((((.(((((((--------------------.-.((....))...)).))))).))))))---..)))).)))).... ( -35.10) >DroAna_CAF1 40921 111 - 1 ACGAUGACAAGCCGUUGUCUAGCCUCCACAGCUGCAGCUCCUCGGGCGUGGCAAGCAACAGCA--CCC-ACACCAAUGCCAGUUCCGAGAAGCUGCUGCUGCUGUCGGGCGUGC ..((..((.....))..))..((((.((((((.((((((.((((((..(((((..........--...-.......)))))..)))))).)))))).)))).))..)))).... ( -45.45) >DroPer_CAF1 43815 99 - 1 ACGAUGACAAGCCGCUGUCCAGCCUGAACAGCUGCAGCUCCUCGGGGCUGGUC--------------A-GCACCAAUGCCAGCUCCGAGAAGCUGCUGGUGAUGUCGGGCGUGC (((.(((((.((((((((.(.....).))))).((((((.(((((((((((.(--------------(-.......))))))))))))).)))))).)))..)))))..))).. ( -53.30) >consensus ACGAUGACAAGCCGCUGUCCAGCCUCAACAGCUGCAGCUCCUCGGGCCUGG_C______________C_ACACCAAUGCCAGCUCCGAGAAGCUGCUG_UGAUGUCGGGCGUGC .....((((......))))..((((...((((.((((((.((((((..((((.........................))))..)))))).))))))....))))..)))).... (-29.29 = -30.96 + 1.67)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:42:35 2006