
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
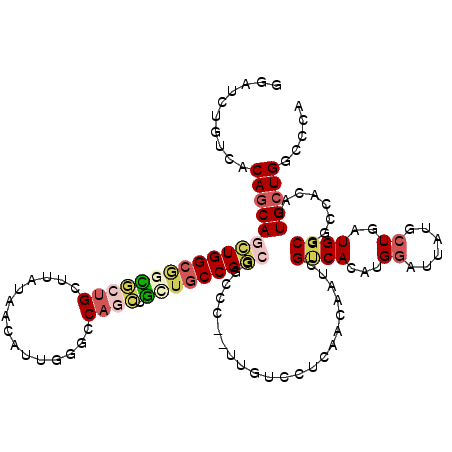
| Location | 2,908,027 – 2,908,138 |
| Length | 111 |
| Max. P | 0.875436 |

| Location | 2,908,027 – 2,908,138 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 74.48 |
| Mean single sequence MFE | -48.27 |
| Consensus MFE | -20.65 |
| Energy contribution | -21.79 |
| Covariance contribution | 1.14 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.89 |
| Structure conservation index | 0.43 |
| SVM decision value | 0.07 |
| SVM RNA-class probability | 0.567535 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
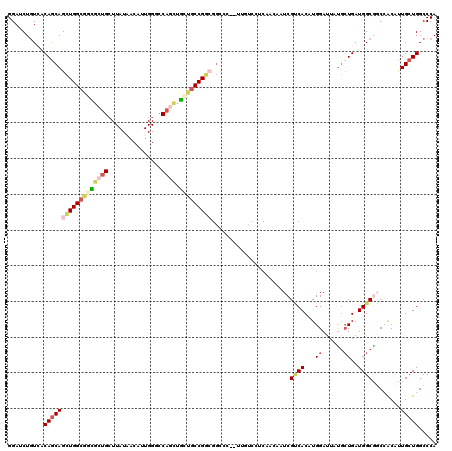
>3L_DroMel_CAF1 2908027 111 + 23771897 GGAUCUCUACCAGCAGCUGGCGGUGCUGCUUAUCACCUUGGGCCAGCUAAUCCCGCAGGCC---------UCGUUUCUCGUCACUUGGAUUACGGUGAUGGCGGCCACCUUGCUGGCCCA .........(((((((.....(((((((((.((((((..(((((.((.......)).))))---------).((.(((........)))..))))))))))))).))))))))))).... ( -41.50) >DroVir_CAF1 197044 120 + 1 GGAACUGUCACAGCAGCUGGCGGCGCUGCUUAUAACGGUGGCGCAGCUGCUGCCGGCGGCGCUGUUAUCCUCAACAAUCGCCACAUGGAUUAUGCUGAUGGCGUCGGCAUUGCUGGCCCA ((....(((((((((((((((((((((((((((....)))).))))).)))))))))((((.((((......))))..))))..........))))).))))((((((...)))))))). ( -56.40) >DroPse_CAF1 171886 120 + 1 GGACCUGUGGCAGCAGCUGGCUGCCCUGCUUAUAACAUUGGGCCAGGUGCUGCCGGCGGCCCAAUGGUCCUCAACAAUCGUCACAUGGACUAUGCUGAUGGCGGCCACCCUGCUGGCCCA ......(.((((((((.((((((((..((......(((((((((.((.....))...)))))))))((((................))))...))....))))))))..))))).)))). ( -52.99) >DroWil_CAF1 144513 120 + 1 GGAUCUGUCCCAACAGCUGGCAGUGGUGCUUAUAACAUUGGCCCAAAUGUUGCCGGCGGCCCACUUGUCCUCAACAAUCGUCACCUGGAUUAUGCUGAUGGCAGCCACAUUGGUGGCCCA ((.......(((.((((.((((((((((((..(((((((......)))))))..))))..)))).))))......((((........))))..)))).)))..(((((....))))))). ( -37.80) >DroMoj_CAF1 189822 120 + 1 GGAGCUGUCACAGCAGCUGGCGGCGGUGCUUAUAACAGUGGGCCAACUGUUGCCGGCGGCGCUGUUGUCGCCAACAAUCGGCACUUGGAUUAUGCUGAUGGCAUCGGCAUUGCUGGCCCA ((((((((....))))))(.((((((((((.((((((((......))))))((((((((((....)))))))....(((((((.........)))))))))))).)))))))))).))). ( -54.40) >DroAna_CAF1 138881 99 + 1 GGAUCUGUCGCAGCAGGUGGCGGCGCUGGUUAUCACCUUGGACCUGCUGGUGCCGC---------------------UCGCCACCUGGAUUAUCCUGAUGGCGGCCACCUUGCUGGCCCA ..........((((((((((((((((..((..((......))...))..)))))..---------------------.(((((.(.((.....)).).))))))))))).)))))..... ( -46.50) >consensus GGAUCUGUCACAGCAGCUGGCGGCGCUGCUUAUAACAUUGGGCCAGCUGCUGCCGGCGGCCC__UUGUCCUCAACAAUCGUCACAUGGAUUAUGCUGAUGGCGGCCACAUUGCUGGCCCA ..........((((((((((((((((((...............)))).)))))))))......................((((...((......))..))))........)))))..... (-20.65 = -21.79 + 1.14)

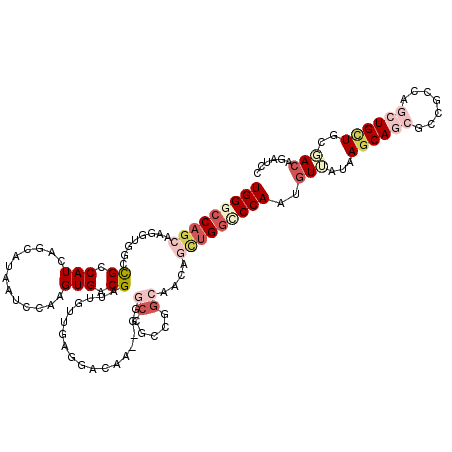
| Location | 2,908,027 – 2,908,138 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 74.48 |
| Mean single sequence MFE | -48.38 |
| Consensus MFE | -17.76 |
| Energy contribution | -20.07 |
| Covariance contribution | 2.31 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.36 |
| Structure conservation index | 0.37 |
| SVM decision value | 0.89 |
| SVM RNA-class probability | 0.875436 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
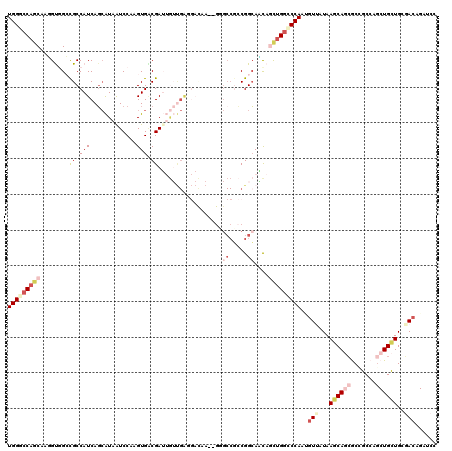
>3L_DroMel_CAF1 2908027 111 - 23771897 UGGGCCAGCAAGGUGGCCGCCAUCACCGUAAUCCAAGUGACGAGAAACGA---------GGCCUGCGGGAUUAGCUGGCCCAAGGUGAUAAGCAGCACCGCCAGCUGCUGGUAGAGAUCC (((((((((..(((((...))))).(((((..((..((........))..---------))..))))).....))))))))).((((........))))(((((...)))))........ ( -45.30) >DroVir_CAF1 197044 120 - 1 UGGGCCAGCAAUGCCGACGCCAUCAGCAUAAUCCAUGUGGCGAUUGUUGAGGAUAACAGCGCCGCCGGCAGCAGCUGCGCCACCGUUAUAAGCAGCGCCGCCAGCUGCUGUGACAGUUCC ..(((((((..(((((..((.....)).........((((((.((((((....)))))))))))))))))...)))).)))...((((((.(((((.......)))))))))))...... ( -50.40) >DroPse_CAF1 171886 120 - 1 UGGGCCAGCAGGGUGGCCGCCAUCAGCAUAGUCCAUGUGACGAUUGUUGAGGACCAUUGGGCCGCCGGCAGCACCUGGCCCAAUGUUAUAAGCAGGGCAGCCAGCUGCUGCCACAGGUCC .(((((.....(((((((.((.((((((..(((.....)))...)))))))).......)))))))(((((((.(((((((..(((.....))).))..))))).)))))))...))))) ( -58.30) >DroWil_CAF1 144513 120 - 1 UGGGCCACCAAUGUGGCUGCCAUCAGCAUAAUCCAGGUGACGAUUGUUGAGGACAAGUGGGCCGCCGGCAACAUUUGGGCCAAUGUUAUAAGCACCACUGCCAGCUGUUGGGACAGAUCC ..((((((....)))))).(((.((((((((((..(....)))))))...((...((((((((...)))((((((......)))))).......))))).)).)))).)))......... ( -39.40) >DroMoj_CAF1 189822 120 - 1 UGGGCCAGCAAUGCCGAUGCCAUCAGCAUAAUCCAAGUGCCGAUUGUUGGCGACAACAGCGCCGCCGGCAACAGUUGGCCCACUGUUAUAAGCACCGCCGCCAGCUGCUGUGACAGCUCC (((((((((..(((((((((.....)))).......(((.((.((((((....)))))))).))))))))...)))))))))((((((((.(((..((.....))))))))))))).... ( -51.30) >DroAna_CAF1 138881 99 - 1 UGGGCCAGCAAGGUGGCCGCCAUCAGGAUAAUCCAGGUGGCGA---------------------GCGGCACCAGCAGGUCCAAGGUGAUAACCAGCGCCGCCACCUGCUGCGACAGAUCC ((..((((((.(((((((((((((.((.....)).))))))).---------------------((((.(((....)))))..(((....))).))...))))))))))).).))..... ( -45.60) >consensus UGGGCCAGCAAGGUGGCCGCCAUCAGCAUAAUCCAAGUGACGAUUGUUGAGGACAA__GGGCCGCCGGCAACAGCUGGCCCAAUGUUAUAAGCAGCGCCGCCAGCUGCUGCGACAGAUCC (((((((((........((.(((.............))).))..................((.....))....)))))))))..(((...((((((.......))))))..)))...... (-17.76 = -20.07 + 2.31)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:55:52 2006