
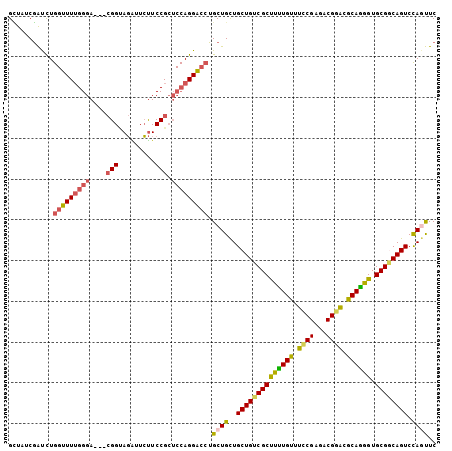
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 293,402 – 293,516 |
| Length | 114 |
| Max. P | 0.663042 |

| Location | 293,402 – 293,504 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 80.97 |
| Mean single sequence MFE | -38.17 |
| Consensus MFE | -30.83 |
| Energy contribution | -31.08 |
| Covariance contribution | 0.26 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.33 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.663042 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 293402 102 + 23771897 GCUUUCGAUCUGGUUUUGGGA---CGGUAGAUUCUUCCGCUCCAGGACCUGCUGUUGCUGUCGCUUUUGUUUCCGAGACGGACGCAGGGUGCGGCAGUCCAAUUC ...........((((((((..---(((.((...)).)))..)))))))).......((((((((((((((.((((...)))).)))))).))))))))....... ( -38.50) >DroSec_CAF1 3195 102 + 1 GCUUUCGAUUUGGUUUUGGGA---CGGUAGAUUCUUCCGCUCCAGGACCUGCUGCUGCUGUCGCUUUUGUUUUCGAGACGGACGCAGGGUGCGGCAGUCCAGUUC ...........((((((((..---(((.((...)).)))..)))))))).((((..((((((((((((((.((((...)))).)))))).)))))))).)))).. ( -39.30) >DroSim_CAF1 5221 102 + 1 GCUUUCGAUUUGGUUUUGGGA---CGGUAGAUUCUUCCGCUCCAGGACCUGCUGCUGCUGUCGCUUUUGUUUUCGAGACGGACGCAGGGUGCGGCAGUCCAGUUC ...........((((((((..---(((.((...)).)))..)))))))).((((..((((((((((((((.((((...)))).)))))).)))))))).)))).. ( -39.30) >DroEre_CAF1 3194 102 + 1 GUUAUCGAUCUGGUUUUGGGA---CGGUAGGUUCUUCCGCUCCAGGAGCUGCUGCUGCUGUCGCUUUUGUUUCCGGGACGGGCGCAAGGUGCGGCAGUCUAGUUC ......((.((((.....((.---((((((.((((........)))).))))))))((((((((((((((.((((...)))).)))))).)))))))))))).)) ( -35.80) >DroYak_CAF1 4193 105 + 1 GUUAUCGACCUGGUUUUGGGACGACGGCACGUUGCUCCGCUCCAGGACCUGCUGCUGCUGUCGCUUUUGUUUCCGGGACGGACGCAGGGUGCGACAGUCCAAUUC ........(((((...(((..(((((...)))))..)))..)))))..........((((((((((((((.((((...)))).)))))).))))))))....... ( -40.00) >DroPer_CAF1 4384 83 + 1 GACAUCUGUUUGGCUU----------------------GCUCCAGGGCGAGCUGCUGCUGGCGCCUCUGCUUGCGCGACGGACGCAGAGUGCGACAGUCCAGCUC ...........(((((----------------------(((....))))))))(((((((.(((((((((.(.((...)).).)))))).))).)))..)))).. ( -36.10) >consensus GCUAUCGAUCUGGUUUUGGGA___CGGUAGAUUCUUCCGCUCCAGGACCUGCUGCUGCUGUCGCUUUUGUUUCCGAGACGGACGCAGGGUGCGGCAGUCCAGUUC ...........(((((((((....(((.........))).))))))))).((((..((((((((((((((.((((...)))).)))))).)))))))).)))).. (-30.83 = -31.08 + 0.26)


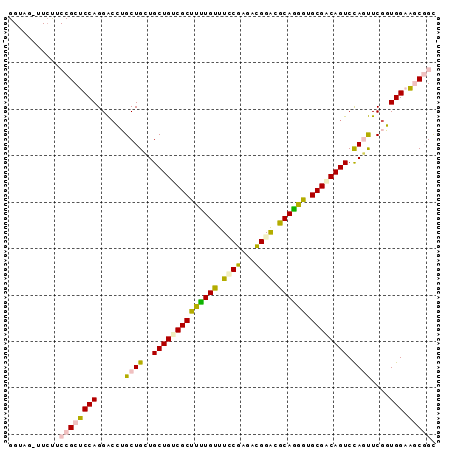
| Location | 293,424 – 293,516 |
|---|---|
| Length | 92 |
| Sequences | 6 |
| Columns | 92 |
| Reading direction | forward |
| Mean pairwise identity | 77.54 |
| Mean single sequence MFE | -36.60 |
| Consensus MFE | -29.70 |
| Energy contribution | -29.65 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.40 |
| Mean z-score | -0.83 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.589097 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 293424 92 + 23771897 GGUAGAUUCUUCCGCUCCAGGACCUGCUGUUGCUGUCGCUUUUGUUUCCGAGACGGACGCAGGGUGCGGCAGUCCAAUUCGGUGGAAGCGGC ........((((((((...((((..((....))((((((((((((.((((...)))).)))))).)))))))))).....)))))))).... ( -34.00) >DroPse_CAF1 4470 79 + 1 -------------GCUCCAGGGCGAGCUGCUGCUGGCGCCUCUGCUUGCGCGAUGGACGCAGAGUGCGACAGUCCAGCUCGUUGGCGACUGC -------------(((....(((((((((..((((.(((((((((.(.(.....).).)))))).))).)))).))))))))))))...... ( -36.20) >DroSim_CAF1 5243 92 + 1 GGUAGAUUCUUCCGCUCCAGGACCUGCUGCUGCUGUCGCUUUUGUUUUCGAGACGGACGCAGGGUGCGGCAGUCCAGUUCGGUGGAAGCGGC ...........(((((((...(((.((((..((((((((((((((.((((...)))).)))))).)))))))).))))..))))).))))). ( -35.40) >DroEre_CAF1 3216 92 + 1 GGUAGGUUCUUCCGCUCCAGGAGCUGCUGCUGCUGUCGCUUUUGUUUCCGGGACGGGCGCAAGGUGCGGCAGUCUAGUUCGGUGGAAGCGGU ...........((((((((.((((((..(((((((.((((((.((.((((...)))).))))))))))))))).))))))..))).))))). ( -39.10) >DroYak_CAF1 4218 92 + 1 GGCACGUUGCUCCGCUCCAGGACCUGCUGCUGCUGUCGCUUUUGUUUCCGGGACGGACGCAGGGUGCGACAGUCCAAUUCGGUGGAAGCGGC .....(((((((((((...((((..((....))((((((((((((.((((...)))).)))))).)))))))))).....))))).)))))) ( -37.50) >DroPer_CAF1 4400 79 + 1 -------------GCUCCAGGGCGAGCUGCUGCUGGCGCCUCUGCUUGCGCGACGGACGCAGAGUGCGACAGUCCAGCUCGUUGGCGACUGC -------------(((....(((((((((..((((.(((((((((.(.((...)).).)))))).))).)))).))))))))))))...... ( -37.40) >consensus GGUAG_UUCUUCCGCUCCAGGACCUGCUGCUGCUGUCGCUUUUGUUUCCGAGACGGACGCAGGGUGCGACAGUCCAGUUCGGUGGAAGCGGC ...........((((((((......((((..((((((((((((((.((((...)))).)))))).)))))))).))))....))).))))). (-29.70 = -29.65 + -0.05)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:31:44 2006