
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 18,728,691 – 18,728,845 |
| Length | 154 |
| Max. P | 0.841151 |

| Location | 18,728,691 – 18,728,811 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.50 |
| Mean single sequence MFE | -44.72 |
| Consensus MFE | -38.42 |
| Energy contribution | -37.90 |
| Covariance contribution | -0.52 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.75 |
| SVM RNA-class probability | 0.841151 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
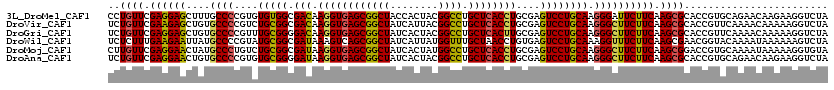
>3L_DroMel_CAF1 18728691 120 - 23771897 CCUGUUCGAGGAGCUUUGCCCCGUGUGUGGCGACAAGGUGAGCGGCUACCACUACGGCCUGCUCACCUGCGAGUCCUGCAAGGGAUUCUUCAAGCGCACCGUGCAGAACAAGAAGGUCUA ..((((((((((((...))(((...((..(.(((.((((((((((((........)))).))))))))....))))..)).)))..)))))..((((...)))).))))).......... ( -47.10) >DroVir_CAF1 18785 120 - 1 UCUGUUCGAAGAGCUGUGCCCCGUCUGCGGCGACAAGGUGAGCGGCUAUCAUUACGGCCUGCUCACCUGCGAGUCCUGCAAGGGCUUCUUCAAGCGCACCGUUCAAAACAAAAAGGUCUA ..........((((.((((.((...(((((.(((.((((((((((((........)))).))))))))....)))))))).))((((....)))))))).))))................ ( -46.30) >DroGri_CAF1 25567 120 - 1 UCUGUUCGAGGAGCUGUGCCCCGUUUGCGGGGACAAGGUGAGCGGCUAUCACUACGGCCUGCUCACUUGCGAGUCCUGCAAGGGCUUCUUCAAGCGCACCGUUCAAAACAAAAAGGUCUA ..((((....((((.((((.((...(((((((...((((((((((((........)))).)))))))).....))))))).))((((....)))))))).))))..)))).......... ( -45.80) >DroWil_CAF1 24399 120 - 1 UCUCUUUGAAGAAUUAUGCCCCGUAUGCGGCGAUAAAGUCAGCGGCUAUCAUUAUGGUUUGCUAACCUGUGAGUCCUGCAAAGGUUUCUUCAAGCGAACGGUACAAAAUAAAAAAGUCUA ...((((.....(((.((..((((.((((((......))).((((...((((...((((....)))).))))...))))..............))).))))..)).)))...)))).... ( -27.20) >DroMoj_CAF1 21547 120 - 1 CUUGUUCGAGGAACUAUGCCCUGUCUGCGGCGAUAAGGUGAGCGGCUAUCACUAUGGCCUGCUCACCUGCGAGUCCUGCAAGGGCUUCUUCAAGCGGACCGUGCAAAAUAAAAAGGUGUA .(((((.((((((....(((((...(((((.(((.((((((((((((((....)))))).))))))))....))))))))))))))))))).)))))(((..............)))... ( -48.14) >DroAna_CAF1 18580 120 - 1 UCUGUUCGAGGAACUGUGCCCCGUGUGCGGGGAUAAGGUGAGCGGCUAUCACUACGGCCUGCUCACCUGCGAGUCCUGCAAGGGCUUCUUCAAGCGCACCGUGCAGAACAAGAAGGUCUA (((((((((((((....((((....(((((((...((((((((((((........)))).)))))))).....))))))).))))))))))..((((...)))).)))).)))....... ( -53.80) >consensus UCUGUUCGAGGAACUGUGCCCCGUCUGCGGCGACAAGGUGAGCGGCUAUCACUACGGCCUGCUCACCUGCGAGUCCUGCAAGGGCUUCUUCAAGCGCACCGUGCAAAACAAAAAGGUCUA ..((((.((((((....((((....(((((.(((.((((((((((((........)))).))))))))....)))))))).)))))))))).))))........................ (-38.42 = -37.90 + -0.52)

| Location | 18,728,731 – 18,728,845 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.74 |
| Mean single sequence MFE | -45.40 |
| Consensus MFE | -33.58 |
| Energy contribution | -33.01 |
| Covariance contribution | -0.58 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.737383 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
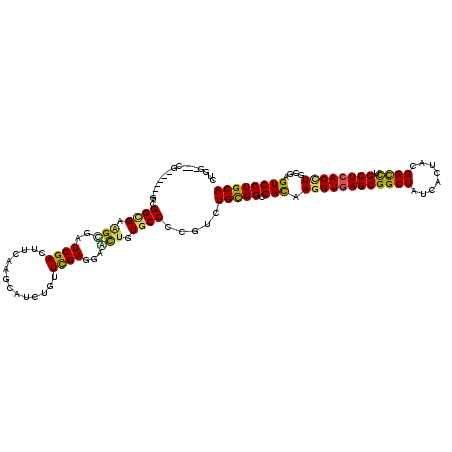
>3L_DroMel_CAF1 18728731 114 - 23771897 CGGGACACG------GCGGCGAGGUGAUCGACUUCAAGCACCUGUUCGAGGAGCUUUGCCCCGUGUGUGGCGACAAGGUGAGCGGCUACCACUACGGCCUGCUCACCUGCGAGUCCUGCA ....(((((------(.((((((((..((((...((......)).))))...))))))))))))))(..(.(((.((((((((((((........)))).))))))))....))))..). ( -54.10) >DroVir_CAF1 18825 111 - 1 CUGG---CG------GCGGCGAGGCCAUCGACUUUAAGCAUCUGUUCGAAGAGCUGUGCCCCGUCUGCGGCGACAAGGUGAGCGGCUAUCAUUACGGCCUGCUCACCUGCGAGUCCUGCA ..((---((------(.((((.((((.(((((...........).)))).).))).))))))))).((((.(((.((((((((((((........)))).))))))))....))))))). ( -51.20) >DroGri_CAF1 25607 111 - 1 CUGG---CA------GUGGUGAAGCGAUCGAUUUUAAGCAUCUGUUCGAGGAGCUGUGCCCCGUUUGCGGGGACAAGGUGAGCGGCUAUCACUACGGCCUGCUCACUUGCGAGUCCUGCA ...(---((------(.(((...((...........(((.(((......))))))((.(((((....))))))).((((((((((((........)))).))))))))))..))))))). ( -41.40) >DroWil_CAF1 24439 118 - 1 --GGACAUAAUGCAUCCGGUGAAGCUAUCGAUUUCAAGCAUCUCUUUGAAGAAUUAUGCCCCGUAUGCGGCGAUAAAGUCAGCGGCUAUCAUUAUGGUUUGCUAACCUGUGAGUCCUGCA --((((((((((...(((.(((...(((((.(((((((......))))))).........(((....))))))))...))).)))....))))))((((....)))).....)))).... ( -32.20) >DroMoj_CAF1 21587 111 - 1 CUGG---CG------GAGGAGAAGCCAUCGACUUCAAGCACUUGUUCGAGGAACUAUGCCCUGUCUGCGGCGAUAAGGUGAGCGGCUAUCACUAUGGCCUGCUCACCUGCGAGUCCUGCA ..((---((------(.((.....((.((((...(((....))).)))))).......))))))).((((.(((.((((((((((((((....)))))).))))))))....))))))). ( -44.50) >DroAna_CAF1 18620 114 - 1 CAGGACAUG------CCGGCGAUGUGAUCGACUUCAAGCAUCUGUUCGAGGAACUGUGCCCCGUGUGCGGGGAUAAGGUGAGCGGCUAUCACUACGGCCUGCUCACCUGCGAGUCCUGCA ((((((.((------((((((((...)))).((((.(((....))).))))..)))..(((((....)))))...((((((((((((........)))).))))))))))).)))))).. ( -49.00) >consensus CUGG___CG______GCGGCGAAGCGAUCGACUUCAAGCAUCUGUUCGAGGAACUGUGCCCCGUCUGCGGCGACAAGGUGAGCGGCUAUCACUACGGCCUGCUCACCUGCGAGUCCUGCA .................((((.(((..((((..............))))...))).)))).....(((((.(((.((((((((((((........)))).))))))))....)))))))) (-33.58 = -33.01 + -0.58)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:33:56 2006