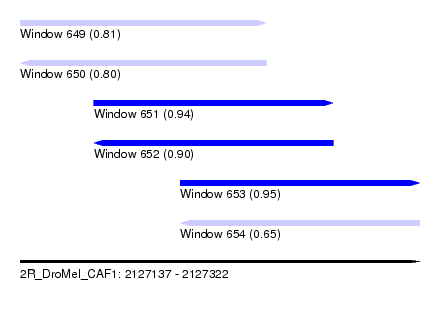
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 2,127,137 – 2,127,322 |
| Length | 185 |
| Max. P | 0.952898 |

| Location | 2,127,137 – 2,127,251 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 82.38 |
| Mean single sequence MFE | -50.89 |
| Consensus MFE | -30.11 |
| Energy contribution | -32.58 |
| Covariance contribution | 2.47 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.91 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.65 |
| SVM RNA-class probability | 0.812604 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2127137 114 + 20766785 ACCAGCUGCCCG---UACAGCUUCCUAAUCUAGUCCAGCUUCCGCCUCAGCUGGCGGCUGCCGCCAAUAUUAUGCAGCUUAAUAACGUGGCCAAAGCUGCAGUGGCUGCGGCGGCCA .(((((((..((---...((((..(((...)))...))))..))...))))))).(((((((((...(((..((((((((.............)))))))))))...))))))))). ( -46.42) >DroVir_CAF1 128749 114 + 1 AUCAGCUGCCGG---UGCAGCUGCCCAAUUUGGUGCAUCUGCCGCCGCAGCUCGCCGCCGCCGCCAACAUAAUGCAGCUGAACAAUGUGGCCAAAGCGGCCGUCGCCGCCGCUGCCA .((((((((.((---(((((.(((((.....)).))).))).))))((.((.....)).))............))))))))......((((...((((((.......)))))))))) ( -48.50) >DroWil_CAF1 150714 117 + 1 AUCAGCUGCCCGCUGUCCAACUGCCCAAUCUAGUCCAGCUGCCACCACAAUUGGCAGCUGCCGCCAAUAUCAUGCAACUGAAUAAUGUGGCCAAGGCGGCGGUGGCAGCGGCGGCUA ...(((((((.((((.(((.(((((...........((((((((.......))))))))(((((((...(((......)))......))))...)))))))))))))))))))))). ( -55.00) >DroMoj_CAF1 174827 114 + 1 ACCAGCUGCCGG---UGCAGCUGCCGAACCUGGUGCAUCUGCCGCCGCAGCUGGCGGCCGCCGCCAACAUCAUGCAGCUGAACAACGUGGCCAAGGCAGCAGUUGCCGCCGCCGCCA ...((((((.((---(((((.((((.......).))).))).))))))))))((((((.(((((.....(((......))).....)))))...(((.((....)).))))))))). ( -52.50) >DroAna_CAF1 110499 114 + 1 ACCAGCUGCCCA---UGCAACUGCCCAAUCUAGUCCAGCUGCCGCCCCAGUUGGCGGCCGCUGCCAACAUCAUGCAGCUGAACAACGUGGCCAAGGCGGCGGUGGCAGCGGCGGCCA ....((((((..---(((.((((((.......((.(((((((.......((((((((...)))))))).....))))))).))......((....)))))))).)))..)))))).. ( -52.50) >DroPer_CAF1 120885 114 + 1 ACCAGCUGCCCG---UGCAGCUGCCGAAUCUAGUCCAACUGCCGCCCCAACUGGCGGCCGCCGCCAACAUAAUGCAGCUGAACAACGUGGCCAAGGCGGCGAUGGCCACGGCAGCGA ....((((((.(---(.(((((((.......((.....))((((((......))))))...............))))))).))...(((((((.(....)..))))))))))))).. ( -50.40) >consensus ACCAGCUGCCCG___UGCAGCUGCCCAAUCUAGUCCAGCUGCCGCCCCAGCUGGCGGCCGCCGCCAACAUCAUGCAGCUGAACAACGUGGCCAAGGCGGCGGUGGCCGCGGCGGCCA ....((((((.......(((((((.......((.....))((((((......))))))...............)))))))......(((((((.........))))))))))))).. (-30.11 = -32.58 + 2.47)

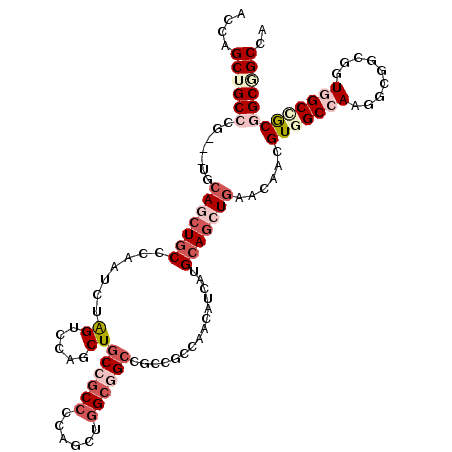
| Location | 2,127,137 – 2,127,251 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 82.38 |
| Mean single sequence MFE | -55.89 |
| Consensus MFE | -38.98 |
| Energy contribution | -38.96 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.28 |
| Structure conservation index | 0.70 |
| SVM decision value | 0.60 |
| SVM RNA-class probability | 0.795973 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2127137 114 - 20766785 UGGCCGCCGCAGCCACUGCAGCUUUGGCCACGUUAUUAAGCUGCAUAAUAUUGGCGGCAGCCGCCAGCUGAGGCGGAAGCUGGACUAGAUUAGGAAGCUGUA---CGGGCAGCUGGU .(((.(((((......((((((((((((...))))..))))))))........))))).)))((((((((...((..((((...(((...)))..))))...---))..)))))))) ( -49.44) >DroVir_CAF1 128749 114 - 1 UGGCAGCGGCGGCGACGGCCGCUUUGGCCACAUUGUUCAGCUGCAUUAUGUUGGCGGCGGCGGCGAGCUGCGGCGGCAGAUGCACCAAAUUGGGCAGCUGCA---CCGGCAGCUGAU ((((...((((((....))))))...))))......((((((((.........((.(((((.....))))).)).((((.(((.((.....))))).)))).---...)))))))). ( -58.90) >DroWil_CAF1 150714 117 - 1 UAGCCGCCGCUGCCACCGCCGCCUUGGCCACAUUAUUCAGUUGCAUGAUAUUGGCGGCAGCUGCCAAUUGUGGUGGCAGCUGGACUAGAUUGGGCAGUUGGACAGCGGGCAGCUGAU ((((.(((((((((((.(((((....)).......(((((((((((.((((((((((...))))))).))).)).)))))))))........))).).))).)))).))).)))).. ( -51.10) >DroMoj_CAF1 174827 114 - 1 UGGCGGCGGCGGCAACUGCUGCCUUGGCCACGUUGUUCAGCUGCAUGAUGUUGGCGGCGGCCGCCAGCUGCGGCGGCAGAUGCACCAGGUUCGGCAGCUGCA---CCGGCAGCUGGU .((((((((((((....))))))...))).((((((.((((........)))))))))))))(((((((((.(..((((.(((..........))).)))).---.).))))))))) ( -60.90) >DroAna_CAF1 110499 114 - 1 UGGCCGCCGCUGCCACCGCCGCCUUGGCCACGUUGUUCAGCUGCAUGAUGUUGGCAGCGGCCGCCAACUGGGGCGGCAGCUGGACUAGAUUGGGCAGUUGCA---UGGGCAGCUGGU .....(((((((((..((((.(((((((..((((((.((((........))))))))))...)))))..))))))(((((((..(......)..))))))).---..)))))).))) ( -55.70) >DroPer_CAF1 120885 114 - 1 UCGCUGCCGUGGCCAUCGCCGCCUUGGCCACGUUGUUCAGCUGCAUUAUGUUGGCGGCGGCCGCCAGUUGGGGCGGCAGUUGGACUAGAUUCGGCAGCUGCA---CGGGCAGCUGGU ..(((((((((((((.........)))))))..(((.(((((((......((((((((.((((((......)))))).))))..)))).....))))))).)---)))))))).... ( -59.30) >consensus UGGCCGCCGCGGCCACCGCCGCCUUGGCCACGUUGUUCAGCUGCAUGAUGUUGGCGGCGGCCGCCAGCUGCGGCGGCAGCUGGACUAGAUUGGGCAGCUGCA___CGGGCAGCUGGU ((((((..((.((....)).))..))))))......((((((((.........(((((.((((((......)))))).......((......))..))))).......)))))))). (-38.98 = -38.96 + -0.02)
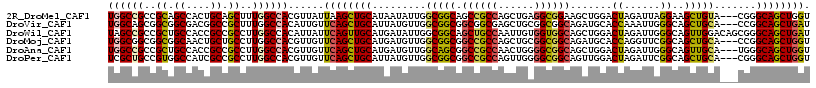
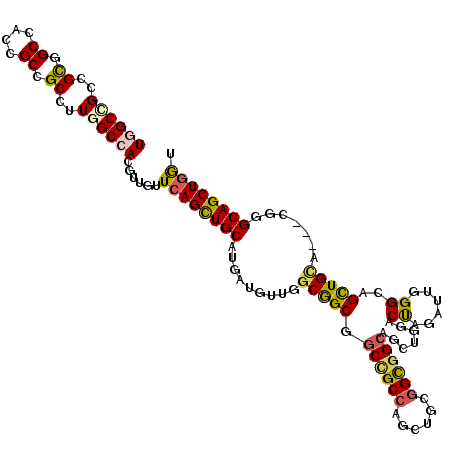
| Location | 2,127,171 – 2,127,282 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 79.99 |
| Mean single sequence MFE | -53.01 |
| Consensus MFE | -30.63 |
| Energy contribution | -31.49 |
| Covariance contribution | 0.86 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.75 |
| Structure conservation index | 0.58 |
| SVM decision value | 1.29 |
| SVM RNA-class probability | 0.938541 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2127171 111 + 20766785 GCUUCCGCCUCAGCUGGCGGCUGCCGCCAAUAUUAUGCAGCUUAAUAACGUGGCCAAAGCUGCAGUGGCUGCGGCGGCCACCAACAACGCGGCCGCGCAAG------CGGCACAGGC ......((((..(.(((.(((((((((...(((..((((((((.............)))))))))))...))))))))).))).)......(((((....)------))))..)))) ( -54.32) >DroVir_CAF1 128783 117 + 1 UCUGCCGCCGCAGCUCGCCGCCGCCGCCAACAUAAUGCAGCUGAACAAUGUGGCCAAAGCGGCCGUCGCCGCCGCUGCCAGCAACAAUGCGGCUGCAGCAGCUGCCUCUGCGCAGGC (((((.((.((((((.((.((.(((((.........(((((((..(.....)..))..(((((....))))).)))))..........))))).)).))))))))....))))))). ( -50.21) >DroWil_CAF1 150751 111 + 1 GCUGCCACCACAAUUGGCAGCUGCCGCCAAUAUCAUGCAACUGAAUAAUGUGGCCAAGGCGGCGGUGGCAGCGGCGGCUAACAACAAUGCAGCCGCUCAAG------CAGCACAGGC (((((..(((....)))..(((((((((....(((......))).....((.((....)).)))))))))))(((((((...........)))))))...)------))))...... ( -46.60) >DroYak_CAF1 106872 111 + 1 GCUUCCGCCCCAGCUGGCGGCCGCCGCCAAUAUUAUGCAGCUGAACAACGUGGCCAAAGCUGCAGUGGCCGCGGCGGCCACUAACAACGCAGCCGCCCAGG------CGGCGCAGGC ......(((..((.((((.(((((.((((......(((((((...............))))))).)))).))))).))))))......((.(((((....)------)))))).))) ( -55.06) >DroMoj_CAF1 174861 117 + 1 UCUGCCGCCGCAGCUGGCGGCCGCCGCCAACAUCAUGCAGCUGAACAACGUGGCCAAGGCAGCAGUUGCCGCCGCCGCCAGCAACAAUGCAGCAGCAGCAGCCGCCUCGGCGCAGGC (((((.(((((((((((((((.(((((.....(((......))).....)))))...(((.((....)).)))))))))))).....))).((.((....)).))...)))))))). ( -53.70) >DroAna_CAF1 110533 111 + 1 GCUGCCGCCCCAGUUGGCGGCCGCUGCCAACAUCAUGCAGCUGAACAACGUGGCCAAGGCGGCGGUGGCAGCGGCGGCCAACAACAACGCCGCAGCUCAAG------CGGCCCAGGC ...(((......((((((.((((((((((...(((......)))....(((.((....)).))).)))))))))).))))))......(((((.......)------))))...))) ( -58.20) >consensus GCUGCCGCCCCAGCUGGCGGCCGCCGCCAACAUCAUGCAGCUGAACAACGUGGCCAAAGCGGCAGUGGCCGCGGCGGCCAACAACAACGCAGCCGCACAAG______CGGCGCAGGC ......(((...((((((.(((((.((((......(((.(((...............))).))).)))).))))).))))))......((....))..................))) (-30.63 = -31.49 + 0.86)
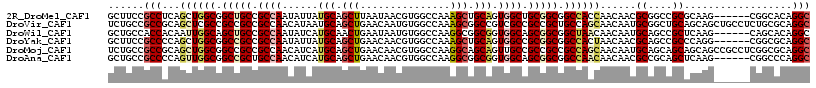

| Location | 2,127,171 – 2,127,282 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 79.99 |
| Mean single sequence MFE | -59.56 |
| Consensus MFE | -32.64 |
| Energy contribution | -32.37 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.61 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.03 |
| SVM RNA-class probability | 0.903507 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2127171 111 - 20766785 GCCUGUGCCG------CUUGCGCGGCCGCGUUGUUGGUGGCCGCCGCAGCCACUGCAGCUUUGGCCACGUUAUUAAGCUGCAUAAUAUUGGCGGCAGCCGCCAGCUGAGGCGGAAGC ((((((((((------(....)))).)))...(((((((((.(((((......((((((((((((...))))..))))))))........))))).)))))))))..))))...... ( -56.14) >DroVir_CAF1 128783 117 - 1 GCCUGCGCAGAGGCAGCUGCUGCAGCCGCAUUGUUGCUGGCAGCGGCGGCGACGGCCGCUUUGGCCACAUUGUUCAGCUGCAUUAUGUUGGCGGCGGCGGCGAGCUGCGGCGGCAGA ((((......)))).((((((((((((((.(((((((..(((((((((....)((((.....))))..........)))))....)))..)))))))..))).)))))))))))... ( -64.50) >DroWil_CAF1 150751 111 - 1 GCCUGUGCUG------CUUGAGCGGCUGCAUUGUUGUUAGCCGCCGCUGCCACCGCCGCCUUGGCCACAUUAUUCAGUUGCAUGAUAUUGGCGGCAGCUGCCAAUUGUGGUGGCAGC ..........------...(.(((((((((....)).))))))))(((((((((((.......((((.(((((.(....).)))))..))))(((....)))....))))))))))) ( -46.70) >DroYak_CAF1 106872 111 - 1 GCCUGCGCCG------CCUGGGCGGCUGCGUUGUUAGUGGCCGCCGCGGCCACUGCAGCUUUGGCCACGUUGUUCAGCUGCAUAAUAUUGGCGGCGGCCGCCAGCUGGGGCGGAAGC ..((((.(((------.((((.((((((((((((.((((((((...)))))))))))))....((((.(((((.(....).)))))..)))).))))))))))).))).)))).... ( -59.90) >DroMoj_CAF1 174861 117 - 1 GCCUGCGCCGAGGCGGCUGCUGCUGCUGCAUUGUUGCUGGCGGCGGCGGCAACUGCUGCCUUGGCCACGUUGUUCAGCUGCAUGAUGUUGGCGGCGGCCGCCAGCUGCGGCGGCAGA ..(((((((((((((((((((((((((((..........))))))))))))...)))))))))))...........((((((....((((((((...)))))))))))))).)))). ( -71.80) >DroAna_CAF1 110533 111 - 1 GCCUGGGCCG------CUUGAGCUGCGGCGUUGUUGUUGGCCGCCGCUGCCACCGCCGCCUUGGCCACGUUGUUCAGCUGCAUGAUGUUGGCAGCGGCCGCCAACUGGGGCGGCAGC (((...((((------(.......)))))(((.(.((((((.((((((((((..(((.....))).((((..(.(....).)..)))))))))))))).)))))).).))))))... ( -58.30) >consensus GCCUGCGCCG______CUUGAGCGGCUGCAUUGUUGCUGGCCGCCGCGGCCACUGCCGCCUUGGCCACGUUGUUCAGCUGCAUGAUAUUGGCGGCGGCCGCCAGCUGCGGCGGCAGC ...((.(((..........(.(((((((((....)).))))))))((.((....)).))...))))).........(((((.........))))).((((((......))))))... (-32.64 = -32.37 + -0.27)

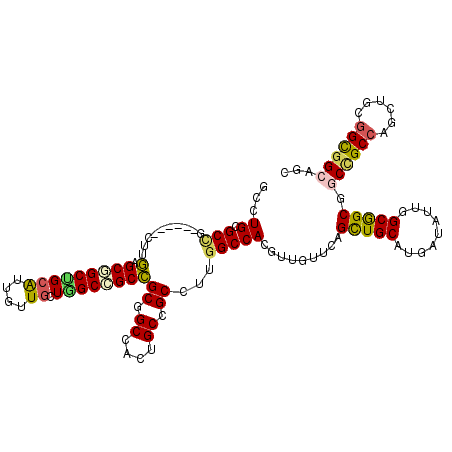
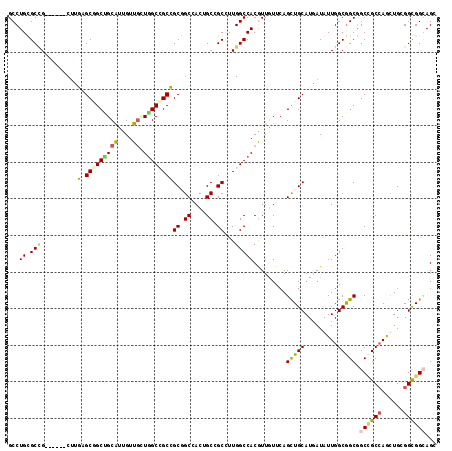
| Location | 2,127,211 – 2,127,322 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 77.02 |
| Mean single sequence MFE | -48.73 |
| Consensus MFE | -25.36 |
| Energy contribution | -27.67 |
| Covariance contribution | 2.31 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.41 |
| Structure conservation index | 0.52 |
| SVM decision value | 1.43 |
| SVM RNA-class probability | 0.952898 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2127211 111 + 20766785 CUUAAUAACGUGGCCAAAGCUGCAGUGGCUGCGGCGGCCACCAACAACGCGGCCGCGCAAG------CGGCACAGGCUGCUCAGGCAGCUGCUGCCCAGUACUUCCUGAACGGCACU .........((((((...(((((((...)))))))))))))......(...(((((....)------))))...)((((.(((((.((.((((....)))))).))))).))))... ( -53.10) >DroVir_CAF1 128823 117 + 1 CUGAACAAUGUGGCCAAAGCGGCCGUCGCCGCCGCUGCCAGCAACAAUGCGGCUGCAGCAGCUGCCUCUGCGCAGGCGGCACAAGCGGCAGCCGCCCAAUACUUUCUGAACGGCACC .........((((((.....))))).)(((((.(((((.(((.........)))))))).(((((((......)))))))....))))).((((..((........))..))))... ( -49.80) >DroEre_CAF1 104610 111 + 1 CUUAACAACGUAGCCAAAGCUGCAGUGGCCGCGGCGGCCACCAACAACGCGGCCGCCCAGG------CAGCGCAGGCUGCUCAGGCAGCUGCUGCCCAGUAUUUUCUGAACGGCACU .....((..(((((....)))))..))((((.(((((((.(.......).)))))))..((------((((...((((((....))))))))))))(((......)))..))))... ( -52.10) >DroWil_CAF1 150791 111 + 1 CUGAAUAAUGUGGCCAAGGCGGCGGUGGCAGCGGCGGCUAACAACAAUGCAGCCGCUCAAG------CAGCACAGGCGGCACAAGCUGCAGCUGCCCAGUUCUUUCUCAAUGGAACG ............((....))((((((.((((((((((((...........)))))))...(------(.((....)).))....))))).))))))..(((((........))))). ( -42.50) >DroYak_CAF1 106912 111 + 1 CUGAACAACGUGGCCAAAGCUGCAGUGGCCGCGGCGGCCACUAACAACGCAGCCGCCCAGG------CGGCGCAGGCUGCUCAGGCAGCUGCUGCCCAAUAUUUUUUGAACGGCACU .........(..((....))..)((((((((...))))))))......(((((.(((..((------((((....))))))..))).)))))(((((((......)))...)))).. ( -48.90) >DroMoj_CAF1 174901 111 + 1 CUGAACAACGUGGCCAAGGCAGCAGUUGCCGCCGCCGCCAGCAACAAUGCAGCAGCAGCAGCCGCCUCGGCGCAGGCGGCGCAGGCAG------CGCAGUAUUUCCUCAACGGGACC (((......((.((....)).)).((((((..(((((((.((.....(((....)))...((((...)))))).)))))))..)))))------).)))....((((....)))).. ( -46.00) >consensus CUGAACAACGUGGCCAAAGCUGCAGUGGCCGCGGCGGCCACCAACAACGCAGCCGCACAAG______CGGCGCAGGCGGCUCAGGCAGCUGCUGCCCAGUACUUUCUGAACGGCACU .........((.(((......((((..(((((.((((((...........))))))...........((((....)))).....)))))..)))).(((......)))...))))). (-25.36 = -27.67 + 2.31)

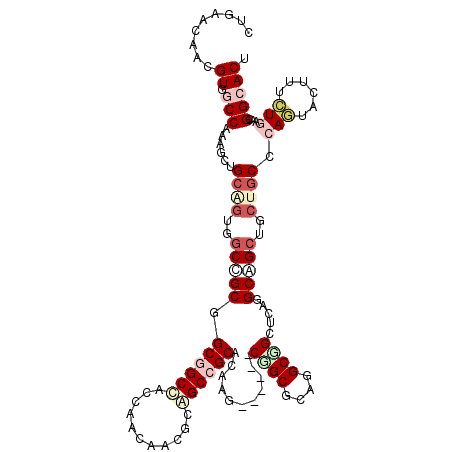

| Location | 2,127,211 – 2,127,322 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 77.02 |
| Mean single sequence MFE | -51.89 |
| Consensus MFE | -27.80 |
| Energy contribution | -28.13 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.77 |
| Structure conservation index | 0.54 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.651737 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 2127211 111 - 20766785 AGUGCCGUUCAGGAAGUACUGGGCAGCAGCUGCCUGAGCAGCCUGUGCCG------CUUGCGCGGCCGCGUUGUUGGUGGCCGCCGCAGCCACUGCAGCUUUGGCCACGUUAUUAAG .(((((((((((......))))))...((((((...((((((.((.((((------(....))))))).))))))((((((.......))))))))))))..)).)))......... ( -48.70) >DroVir_CAF1 128823 117 - 1 GGUGCCGUUCAGAAAGUAUUGGGCGGCUGCCGCUUGUGCCGCCUGCGCAGAGGCAGCUGCUGCAGCCGCAUUGUUGCUGGCAGCGGCGGCGACGGCCGCUUUGGCCACAUUGUUCAG ((((((((((((......))))))))).)))((((.(((.(....)))).)))).((((((((.((((((....))).))).))))))))...((((.....))))........... ( -57.00) >DroEre_CAF1 104610 111 - 1 AGUGCCGUUCAGAAAAUACUGGGCAGCAGCUGCCUGAGCAGCCUGCGCUG------CCUGGGCGGCCGCGUUGUUGGUGGCCGCCGCGGCCACUGCAGCUUUGGCUACGUUGUUAAG .(((((((((((......)))))).((.(((((((..(((((....))))------)..))))))).))(((((.((((((((...)))))))))))))...))).))......... ( -56.90) >DroWil_CAF1 150791 111 - 1 CGUUCCAUUGAGAAAGAACUGGGCAGCUGCAGCUUGUGCCGCCUGUGCUG------CUUGAGCGGCUGCAUUGUUGUUAGCCGCCGCUGCCACCGCCGCCUUGGCCACAUUAUUCAG ........((((.........((((((.(((((.............))))------)..(.(((((((((....)).))))))))))))))...(((.....))).......)))). ( -38.32) >DroYak_CAF1 106912 111 - 1 AGUGCCGUUCAAAAAAUAUUGGGCAGCAGCUGCCUGAGCAGCCUGCGCCG------CCUGGGCGGCUGCGUUGUUAGUGGCCGCCGCGGCCACUGCAGCUUUGGCCACGUUGUUCAG .(((((((((((......)))))).((((((((((..((.((....)).)------)..))))))))))(((((.((((((((...)))))))))))))...)).)))......... ( -56.20) >DroMoj_CAF1 174901 111 - 1 GGUCCCGUUGAGGAAAUACUGCG------CUGCCUGCGCCGCCUGCGCCGAGGCGGCUGCUGCUGCUGCAUUGUUGCUGGCGGCGGCGGCAACUGCUGCCUUGGCCACGUUGUUCAG (((.((.....)).......(((------(.....)))).)))((.(((((((((((((((((((((((..........))))))))))))...))))))))))))).......... ( -54.20) >consensus AGUGCCGUUCAGAAAAUACUGGGCAGCAGCUGCCUGAGCAGCCUGCGCCG______CUUGGGCGGCCGCAUUGUUGGUGGCCGCCGCGGCCACUGCAGCUUUGGCCACGUUGUUCAG .(((((((((((......)))))).((((((((....((.....))..............((((((((((....)).)))))))))))))...)))......))).))......... (-27.80 = -28.13 + 0.34)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:35:28 2006