
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 1,420,987 – 1,421,133 |
| Length | 146 |
| Max. P | 0.999858 |

| Location | 1,420,987 – 1,421,101 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.61 |
| Mean single sequence MFE | -37.90 |
| Consensus MFE | -30.10 |
| Energy contribution | -31.50 |
| Covariance contribution | 1.40 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.79 |
| SVM decision value | 1.83 |
| SVM RNA-class probability | 0.979316 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1420987 114 + 20766785 GCCACGCC------CACGCCCAAACGCAUACGCACAUGCUGGGACGAACACACACACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUU ..(((((.------..((((((...((((......)))))))).)).............(((((..(((((((..(((..(((....)))..)))...)))))))..))))).))))).. ( -40.00) >DroSec_CAF1 39430 116 + 1 GCCACGCCCACGCCCACGCCCAAACGCAUACGCACAUGCUGGGACGAACAC----ACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUU ....((((((.((....))......((((......)))))))).))(((((----.((.(((((..(((((((..(((..(((....)))..)))...)))))))..))))))).))))) ( -40.40) >DroSim_CAF1 44488 110 + 1 GCCACACC------CACGCCCAAACGCAUACGCACAUGCUAGGACGAACAC----ACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUU ........------..((.((....((((......))))..)).))(((((----.((.(((((..(((((((..(((..(((....)))..)))...)))))))..))))))).))))) ( -35.20) >DroEre_CAF1 37781 109 + 1 -CCACGCC------CACACCCACACACAUACGCAUACGCAGGGACGAGCAC----ACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUU -...((((------(.........................))).))(((((----.((.(((((..(((((((..(((..(((....)))..)))...)))))))..))))))).))))) ( -34.01) >DroYak_CAF1 38806 109 + 1 -UCACUCC------CAAAGCCACACGCAUACGCACAUGCUGGGGCGAGCAC----ACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUU -.......------....(((.((.((((......)))))).))).(((((----.((.(((((..(((((((..(((..(((....)))..)))...)))))))..))))))).))))) ( -39.90) >consensus GCCACGCC______CACGCCCAAACGCAUACGCACAUGCUGGGACGAACAC____ACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUU ..(((((...........((((...((((......))))))))................(((((..(((((((..(((..(((....)))..)))...)))))))..))))).))))).. (-30.10 = -31.50 + 1.40)

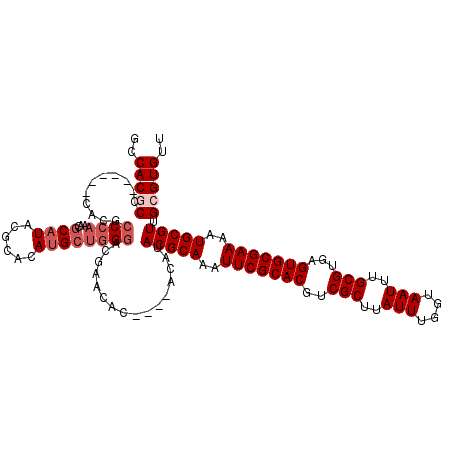
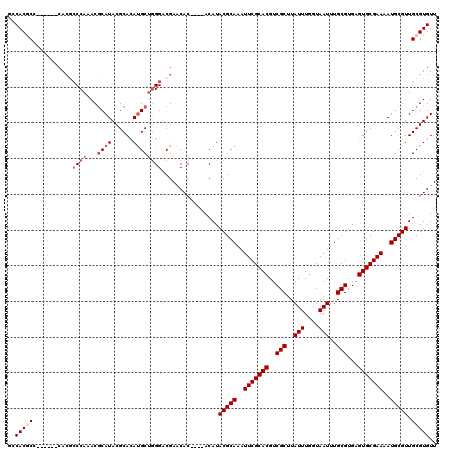
| Location | 1,420,987 – 1,421,101 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.61 |
| Mean single sequence MFE | -44.84 |
| Consensus MFE | -37.50 |
| Energy contribution | -37.26 |
| Covariance contribution | -0.24 |
| Combinations/Pair | 1.15 |
| Mean z-score | -3.06 |
| Structure conservation index | 0.84 |
| SVM decision value | 3.45 |
| SVM RNA-class probability | 0.999225 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1420987 114 - 20766785 AACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGUGUGUGUGUUCGUCCCAGCAUGUGCGUAUGCGUUUGGGCGUG------GGCGUGGC .((((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))))))((.(.(((((..(((((....)))))....))))).)------.))..... ( -45.10) >DroSec_CAF1 39430 116 - 1 AACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGU----GUGUUCGUCCCAGCAUGUGCGUAUGCGUUUGGGCGUGGGCGUGGGCGUGGC (((((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))----)))))((.((((.....(((.(((((......))))).))))))))).... ( -47.00) >DroSim_CAF1 44488 110 - 1 AACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGU----GUGUUCGUCCUAGCAUGUGCGUAUGCGUUUGGGCGUG------GGUGUGGC (((((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))----)))))(((((..(((((....)))))....)))))..------........ ( -43.00) >DroEre_CAF1 37781 109 - 1 AACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGU----GUGCUCGUCCCUGCGUAUGCGUAUGUGUGUGGGUGUG------GGCGUGG- ..(((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))----)))(.(((((.(((.((((((....)))))).))).)------)))).).- ( -44.00) >DroYak_CAF1 38806 109 - 1 AACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGU----GUGCUCGCCCCAGCAUGUGCGUAUGCGUGUGGCUUUG------GGAGUGA- ..(((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))----))).(((((((((((..(((....)))..))...)))------)).))))- ( -45.10) >consensus AACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGU____GUGUUCGUCCCAGCAUGUGCGUAUGCGUUUGGGCGUG______GGCGUGGC ..(((((.(((((..(((((((...(((..(((....)))..)))..)))))))..))))).............(((((..(((((....)))))....))))).........))))).. (-37.50 = -37.26 + -0.24)

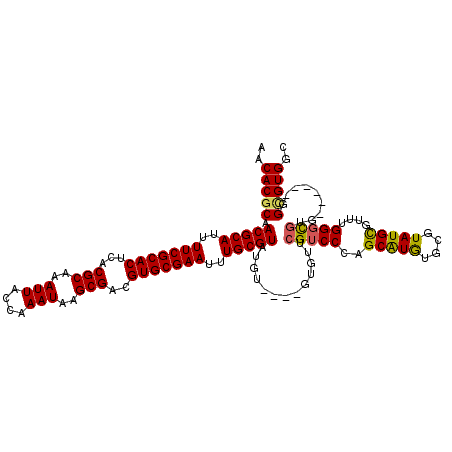
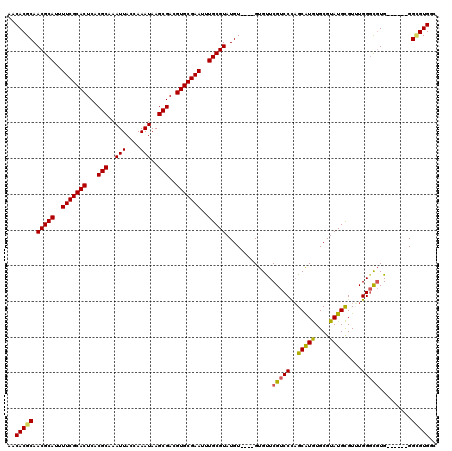
| Location | 1,421,021 – 1,421,133 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.20 |
| Mean single sequence MFE | -46.30 |
| Consensus MFE | -40.40 |
| Energy contribution | -40.24 |
| Covariance contribution | -0.16 |
| Combinations/Pair | 1.09 |
| Mean z-score | -4.51 |
| Structure conservation index | 0.87 |
| SVM decision value | 4.28 |
| SVM RNA-class probability | 0.999858 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1421021 112 + 20766785 GGGACGAACACACACACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUUUGCCUAACAUGCAGUCCGUCCAUA--------UGACUGCG (((.(((((((.((.....(((((..(((((((..(((..(((....)))..)))...)))))))..))))))).)))))))))).....((((((........--------.)))))). ( -44.00) >DroSec_CAF1 39470 108 + 1 GGGACGAACAC----ACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUUUGCCUAACAUGCAGUCCGUCCAUA--------UGACUGCG (((.(((((((----.((.(((((..(((((((..(((..(((....)))..)))...)))))))..))))))).)))))))))).....((((((........--------.)))))). ( -46.10) >DroSim_CAF1 44522 108 + 1 AGGACGAACAC----ACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUUUGCCUAACAUGCAGUCCGUCCAUA--------UGACUGCG (((.(((((((----.((.(((((..(((((((..(((..(((....)))..)))...)))))))..))))))).)))))))))).....((((((........--------.)))))). ( -46.10) >DroEre_CAF1 37814 108 + 1 GGGACGAGCAC----ACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUUUGCCUAACAUGCAGUGCGUCCAUG--------UGACUGCA (((.(((((((----.((.(((((..(((((((..(((..(((....)))..)))...)))))))..))))))).))))))))))....(((((((((....))--------).)))))) ( -45.30) >DroYak_CAF1 38839 116 + 1 GGGGCGAGCAC----ACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUUUGCCUAACAUUCGGUCCGUACAUGACCGUAUAUGACUGCG .((((((((((----.((.(((((..(((((((..(((..(((....)))..)))...)))))))..))))))).))))))))))..(((.(((((.......)))))...)))...... ( -50.00) >consensus GGGACGAACAC____ACAUACGCAAAUUCGCACGUCGCUUAUUUGGUAAUUUGCGUGAGUGCGAAAAUGCGUUGCGUGUUUGCCUAACAUGCAGUCCGUCCAUA________UGACUGCG .(((((.((..........(((((..(((((((..(((..(((....)))..)))...)))))))..)))))((((((((.....)))))))))).)))))................... (-40.40 = -40.24 + -0.16)



| Location | 1,421,021 – 1,421,133 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.20 |
| Mean single sequence MFE | -43.22 |
| Consensus MFE | -34.35 |
| Energy contribution | -35.19 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.03 |
| Mean z-score | -4.48 |
| Structure conservation index | 0.79 |
| SVM decision value | 3.03 |
| SVM RNA-class probability | 0.998204 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 1421021 112 - 20766785 CGCAGUCA--------UAUGGACGGACUGCAUGUUAGGCAAACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGUGUGUGUGUUCGUCCC ........--------...(((((((((((.......))).((((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))))))..)))))))). ( -45.50) >DroSec_CAF1 39470 108 - 1 CGCAGUCA--------UAUGGACGGACUGCAUGUUAGGCAAACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGU----GUGUUCGUCCC .((((((.--------........))))))......(((.(((((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))----))))).))).. ( -44.90) >DroSim_CAF1 44522 108 - 1 CGCAGUCA--------UAUGGACGGACUGCAUGUUAGGCAAACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGU----GUGUUCGUCCU .((((((.--------........))))))......(((.(((((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))----))))).))).. ( -44.90) >DroEre_CAF1 37814 108 - 1 UGCAGUCA--------CAUGGACGCACUGCAUGUUAGGCAAACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGU----GUGCUCGUCCC ........--------...((((((.(((.....)))))...(((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))----)))...)))). ( -38.30) >DroYak_CAF1 38839 116 - 1 CGCAGUCAUAUACGGUCAUGUACGGACCGAAUGUUAGGCAAACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGU----GUGCUCGCCCC .......((((.(((((.......))))).))))..(((...(((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))----)))...))).. ( -42.50) >consensus CGCAGUCA________UAUGGACGGACUGCAUGUUAGGCAAACACGCAACGCAUUUUCGCACUCACGCAAAUUACCAAAUAAGCGACGUGCGAAUUUGCGUAUGU____GUGUUCGUCCC .((((((.................))))))......(((.(((((((((((((..(((((((...(((..(((....)))..)))..)))))))..))))).)))....))))).))).. (-34.35 = -35.19 + 0.84)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:30:23 2006