
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 4,223,269 – 4,223,429 |
| Length | 160 |
| Max. P | 0.984191 |

| Location | 4,223,269 – 4,223,389 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.42 |
| Mean single sequence MFE | -39.58 |
| Consensus MFE | -33.44 |
| Energy contribution | -34.08 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.97 |
| SVM RNA-class probability | 0.984191 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4223269 120 + 20766785 ACUAGUGCGAAGAUCACGAAUGGAGUUAGUACGAUCUGUAAACAGUUGAGCAUCUUGCUCCAACCGUGCUUGACCAGCUGGUAGUCCACCAAUGGAACCACCAAAUGGAGCAUUAGCGUG .(((((((.(((..((((..((((((..((.(((.(((....)))))).)).....))))))..)))))))..(((..((((..((((....))))...))))..))).))))))).... ( -39.40) >DroSec_CAF1 35122 120 + 1 ACUAGUGCGAAGAUCACGAAUGGAGUCAGGACGAUCUGUAAGCAGUUGAGCAUCUUGCUCCAACCGUGCUUGACCAGCUGGUAGUCCACCAAUGGAACCACCAAAUGGAGCAUCAGAGUG .((.((((.(((..((((..(((((.((((((((.(((....))))))....))))))))))..)))))))..(((..((((..((((....))))...))))..))).)))).)).... ( -39.30) >DroSim_CAF1 62832 120 + 1 ACUAGUGCGAAGAUCACGAAUGGAGUCAGGACGAUCUGCAAGCAGUUGAGCAUCUUGCUCCAACCGUGCUUGACCAGCUGGUAGUCCACCAAUGGAACCACCAAAUGGAGCAUUAGAGUG .(((((((.(((..((((..(((((.((((((((.(((....))))))....))))))))))..)))))))..(((..((((..((((....))))...))))..))).))))))).... ( -43.10) >DroEre_CAF1 36653 120 + 1 ACUAGAGCGACGAUCACGAAAGGAGUCAGUACAAUUUGUAGGCAGUUGAGCAUCUUGCUCCAGCCGUGCUUGACCUUCUGGUAGUCCACCAAUGGAACCACCAACUGGAGCAUUAGGUUG ....((((((.(((..(....)..))).((.(((((.(....)))))).))...))))))((((((((((...((...((((..((((....))))...))))...)))))))..))))) ( -35.40) >DroYak_CAF1 37770 120 + 1 AUUAGUGCGACGAUCACGAAAGGAGUCAGUACGAUUUGUAAGCAGUUGAGCAUCUUGCUCCAGCCGUGCUUGACCAUCUGGUAAUCCACCAAUGGAAGCACCAACUGGAGCAUAAGGUUG ....((((...(((..(....)..))).)))).........((......))((((((((((((..((((((..((((.((((.....))))))))))))))...))))))))..)))).. ( -40.70) >consensus ACUAGUGCGAAGAUCACGAAUGGAGUCAGUACGAUCUGUAAGCAGUUGAGCAUCUUGCUCCAACCGUGCUUGACCAGCUGGUAGUCCACCAAUGGAACCACCAAAUGGAGCAUUAGAGUG .(((((((...((.((((..((((((.((..(((.(((....)))))).....)).))))))..)))).))..(((..((((..((((....))))...))))..))).))))))).... (-33.44 = -34.08 + 0.64)

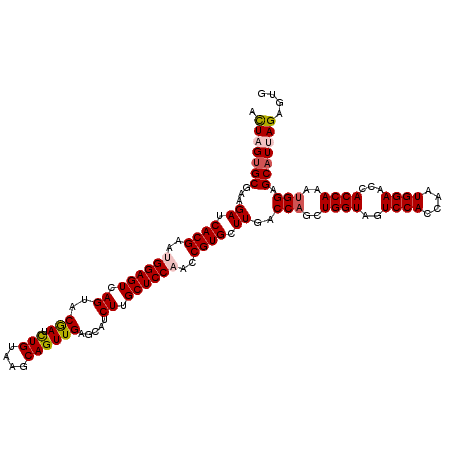
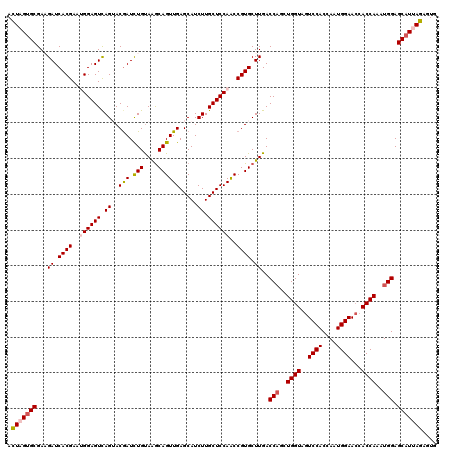
| Location | 4,223,269 – 4,223,389 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.42 |
| Mean single sequence MFE | -39.38 |
| Consensus MFE | -32.14 |
| Energy contribution | -33.50 |
| Covariance contribution | 1.36 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.58 |
| SVM RNA-class probability | 0.965223 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4223269 120 - 20766785 CACGCUAAUGCUCCAUUUGGUGGUUCCAUUGGUGGACUACCAGCUGGUCAAGCACGGUUGGAGCAAGAUGCUCAACUGUUUACAGAUCGUACUAACUCCAUUCGUGAUCUUCGCACUAGU ...((((.(((.(((.((((((((.(((....))))))))))).))).....((((..(((((..((.(((....(((....)))...)))))..)))))..))))......))).)))) ( -41.00) >DroSec_CAF1 35122 120 - 1 CACUCUGAUGCUCCAUUUGGUGGUUCCAUUGGUGGACUACCAGCUGGUCAAGCACGGUUGGAGCAAGAUGCUCAACUGCUUACAGAUCGUCCUGACUCCAUUCGUGAUCUUCGCACUAGU ....(((.(((.(((.((((((((.(((....))))))))))).))).....((((..(((((((.((((.((...........)).)))).)).)))))..))))......))).))). ( -41.20) >DroSim_CAF1 62832 120 - 1 CACUCUAAUGCUCCAUUUGGUGGUUCCAUUGGUGGACUACCAGCUGGUCAAGCACGGUUGGAGCAAGAUGCUCAACUGCUUGCAGAUCGUCCUGACUCCAUUCGUGAUCUUCGCACUAGU ....(((.(((.(((.((((((((.(((....))))))))))).))).....((((..(((((((.((((.....(((....)))..)))).)).)))))..))))......))).))). ( -42.60) >DroEre_CAF1 36653 120 - 1 CAACCUAAUGCUCCAGUUGGUGGUUCCAUUGGUGGACUACCAGAAGGUCAAGCACGGCUGGAGCAAGAUGCUCAACUGCCUACAAAUUGUACUGACUCCUUUCGUGAUCGUCGCUCUAGU ........((((((..((((((((.(((....)))))))))))..))...))))..((((((((..((((.(((......(((.....)))..((......)).))).)))))))))))) ( -35.10) >DroYak_CAF1 37770 120 - 1 CAACCUUAUGCUCCAGUUGGUGCUUCCAUUGGUGGAUUACCAGAUGGUCAAGCACGGCUGGAGCAAGAUGCUCAACUGCUUACAAAUCGUACUGACUCCUUUCGUGAUCGUCGCACUAAU ........(((((((((((.((((((((((((((...))))).))))..)))))))))))))))).((((.(((......(((.....)))..((......)).))).))))........ ( -37.00) >consensus CACCCUAAUGCUCCAUUUGGUGGUUCCAUUGGUGGACUACCAGCUGGUCAAGCACGGUUGGAGCAAGAUGCUCAACUGCUUACAGAUCGUACUGACUCCAUUCGUGAUCUUCGCACUAGU ....(((.(((.(((.((((((((.(((....))))))))))).))).....(((((.(((((..((..((......)).(((.....)))))..))))).)))))......))).))). (-32.14 = -33.50 + 1.36)

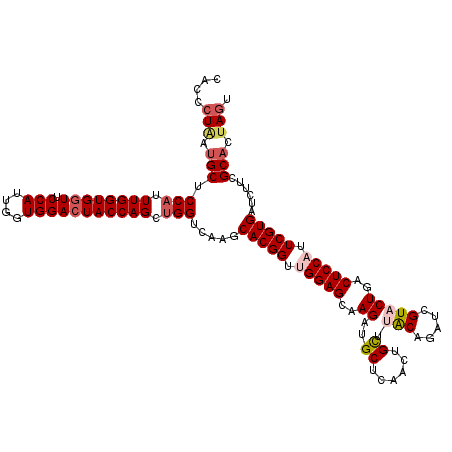
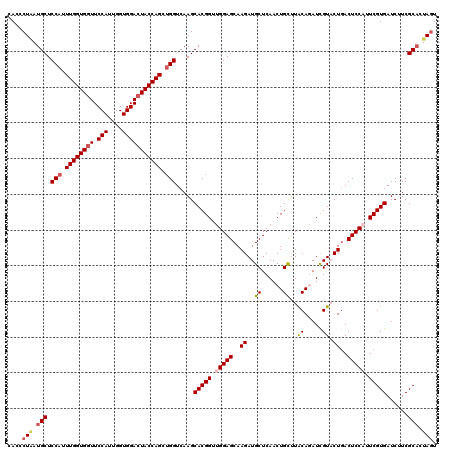
| Location | 4,223,309 – 4,223,429 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.67 |
| Mean single sequence MFE | -39.54 |
| Consensus MFE | -32.32 |
| Energy contribution | -33.36 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.33 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.533560 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4223309 120 - 20766785 GUACCUGUGCACGCAUUUUGCAAAUCCUCAAGGCCCCGGUCACGCUAAUGCUCCAUUUGGUGGUUCCAUUGGUGGACUACCAGCUGGUCAAGCACGGUUGGAGCAAGAUGCUCAACUGUU (((....)))..(((((((((................((((.((((((((..((((...))))...)))))))))))).(((((((........))))))).)))))))))......... ( -42.00) >DroSec_CAF1 35162 120 - 1 GUACCUGUGCACGCGUUUUGCAAAUCCUCAAGGCCCCGGUCACUCUGAUGCUCCAUUUGGUGGUUCCAUUGGUGGACUACCAGCUGGUCAAGCACGGUUGGAGCAAGAUGCUCAACUGCU (((....)))..(((((((((...(((....(((....)))...(((.(((((((.((((((((.(((....))))))))))).)))...)))))))..))))))))))))......... ( -41.60) >DroSim_CAF1 62872 120 - 1 GUACCUGUGCACGCGUUUUGCAAAUCCUCAAGGCCCCGGUCACUCUAAUGCUCCAUUUGGUGGUUCCAUUGGUGGACUACCAGCUGGUCAAGCACGGUUGGAGCAAGAUGCUCAACUGCU (((....)))..(((((((((..........(((....)))..((((((((((((.((((((((.(((....))))))))))).)))...))))...))))))))))))))......... ( -40.30) >DroEre_CAF1 36693 120 - 1 GUACAUGCACCCGAAUCCUACACAUCCUCAAGGCCCCGGUCAACCUAAUGCUCCAGUUGGUGGUUCCAUUGGUGGACUACCAGAAGGUCAAGCACGGCUGGAGCAAGAUGCUCAACUGCC ......(((......................((((..((....))...((((((..((((((((.(((....)))))))))))..))...)))).)))).((((.....))))...))). ( -33.70) >DroYak_CAF1 37810 120 - 1 GUACAUGUGCACGCAUUUUACAUAUCCUCAAAGCCCCGGUCAACCUUAUGCUCCAGUUGGUGCUUCCAUUGGUGGAUUACCAGAUGGUCAAGCACGGCUGGAGCAAGAUGCUCAACUGCU (((....)))..(((((((..................((....))....((((((((((.((((((((((((((...))))).))))..))))))))))))))))))))))......... ( -40.10) >consensus GUACCUGUGCACGCAUUUUGCAAAUCCUCAAGGCCCCGGUCACCCUAAUGCUCCAUUUGGUGGUUCCAUUGGUGGACUACCAGCUGGUCAAGCACGGUUGGAGCAAGAUGCUCAACUGCU (((....)))..(((((((((...(((....(((....))).......(((((((.((((((((.(((....))))))))))).)))...)))).....))))))))))))......... (-32.32 = -33.36 + 1.04)

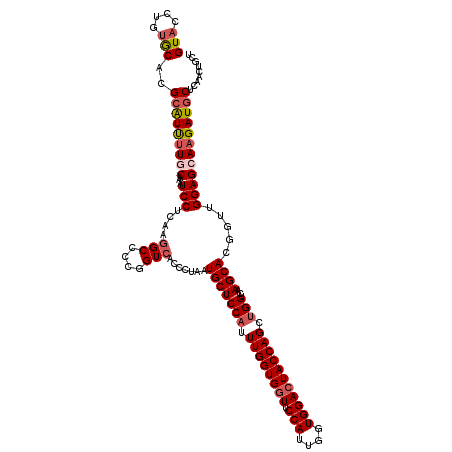
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:54:26 2006