Epigenetik:
Epigenetik ist ein Gebiet in der Biologie, das sich mit den Vorgängen und Modifikation auf den Phänotyp befasst, die nicht von der Genregulation und Genexpression beeinflusst werden. Die Fragestellung ist, wie die Aktivität von Genen beeinflusst werden kann und damit auch die Entwicklung von Zellen bzw. des Organismus. Die Grundlagen bilden Modifikationen am Chromosom, die dabei keine Veränderung der DNA-Sequenz beinhaltet. Zu den Veränderungen zählen DNA-Methylierung oder die Modifikation der Histone.
Histone sind Proteine, die im Zellkern der Eukaryoten vorkommen und die DNA 'verpacken'. Dabei wickelt sich die DNA ca.1,5 mal um den Histonkomplex und bildet mit ihm ein Nukleosom.

Abbildung 1: Nucleosome structure (Das Bild per Mausklick ran und weg zoomen)
Dieser Komplex besteht aus 8 Histonen, je zwei Kopien von den Histonen H2A, H2B, H3, und H4, diese bilden ein Oktamer. Die Histone sind aus einem globalen Zentrum aufgebaut mit endständigen Armen, den Histon-Tails.
Durch die chemische Veränderung der Histon-Proteine hat die Histonmodifikation zum Teil auch einen Einfluss auf die Transkription. Dies umfasst unter anderem Mehylierung, Phosphorylierung, Sumoylierung, Ubiquitinylierung und Acetylierung, sowohl auch deren Rückreaktion.
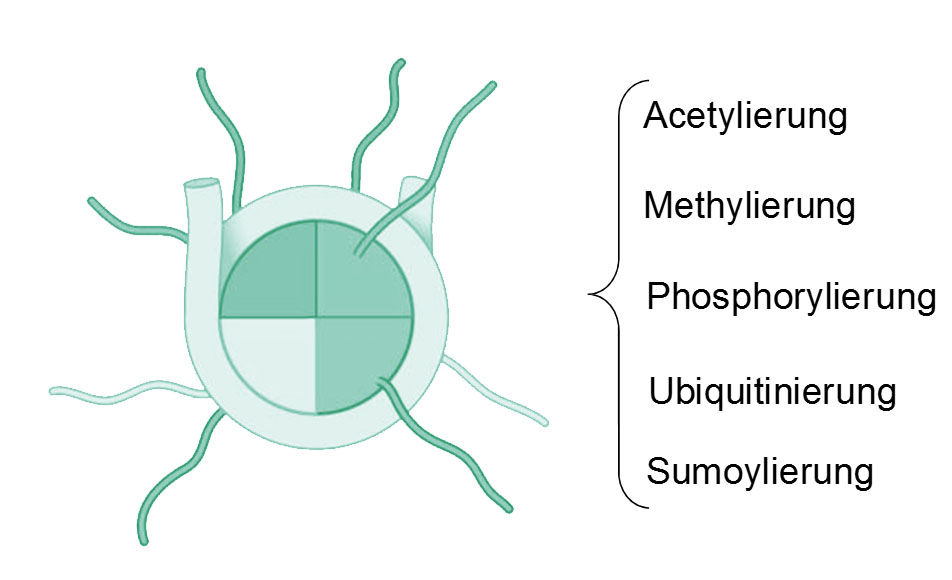
Abbildung 2: Histonmodifikation (Das Bild per Mausklick ran und weg zoomen)
Nomenklatur der modifzierenden Enzyme:
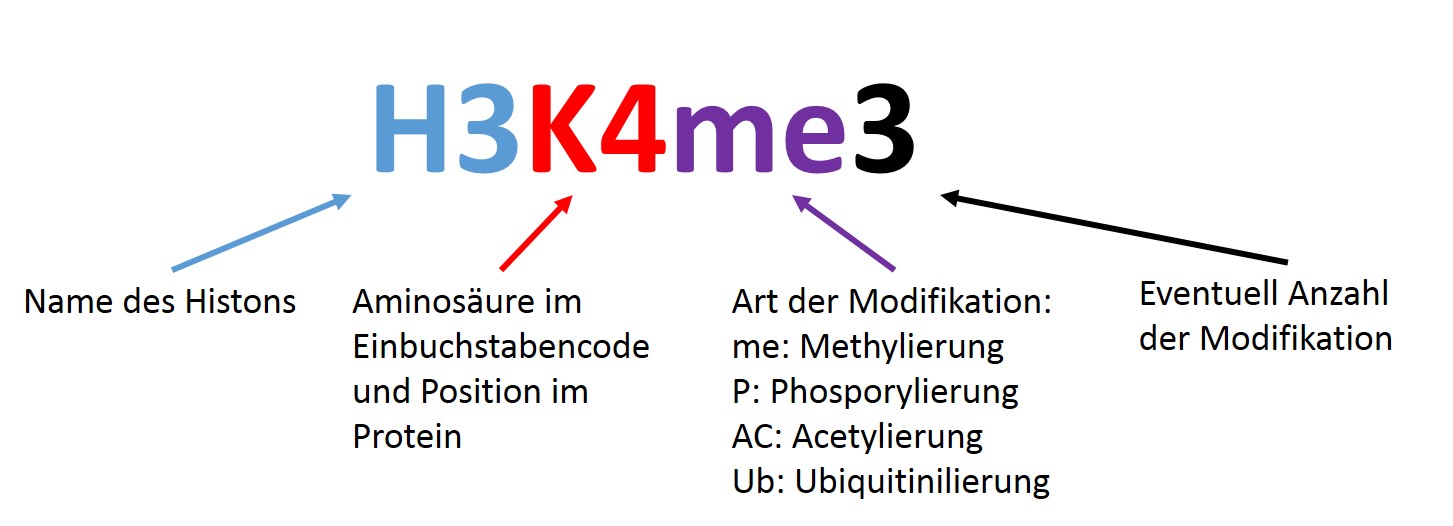
Abbildung 3: Nomenklatur (Das Bild per Mausklick ran und weg zoomen)
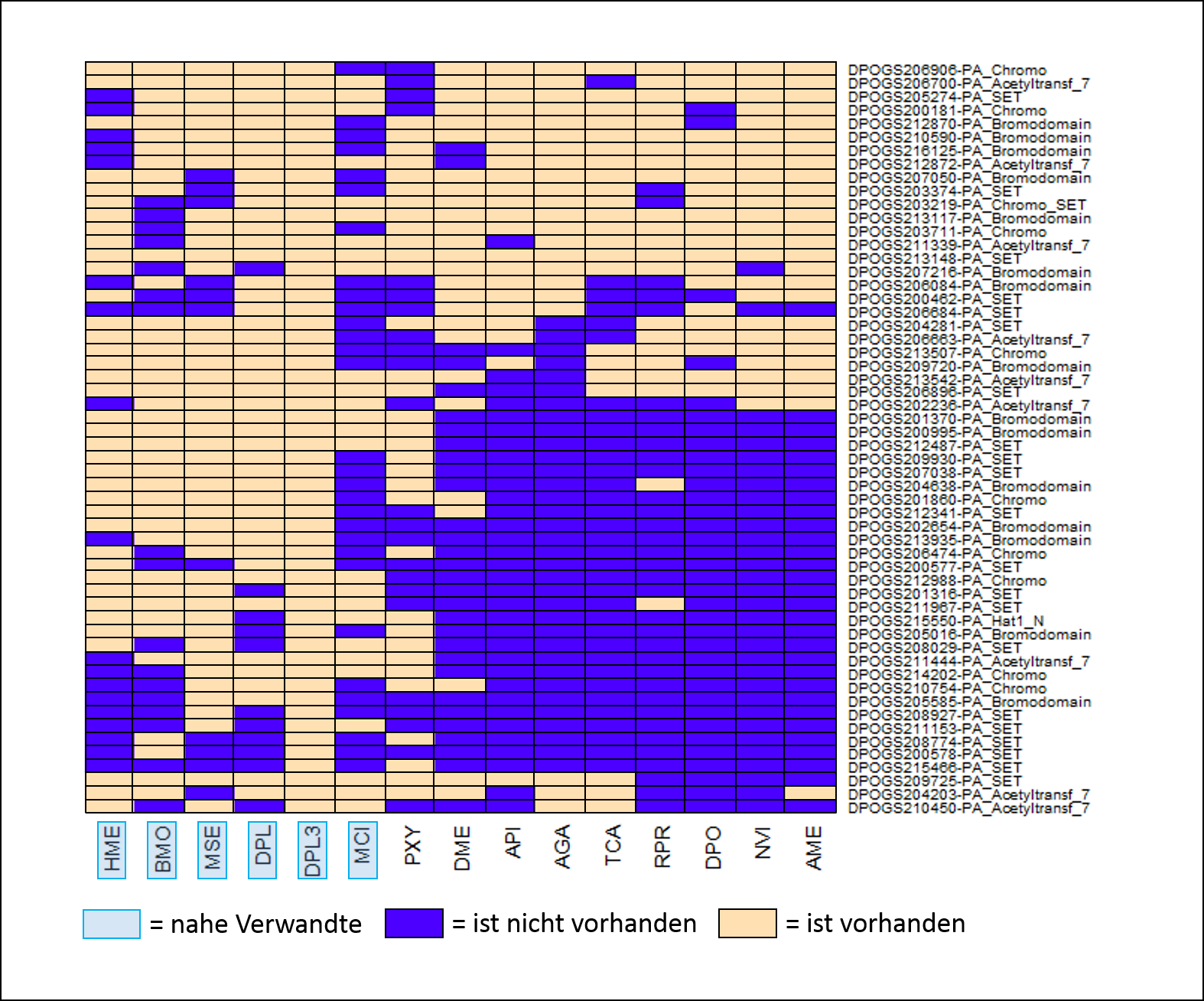
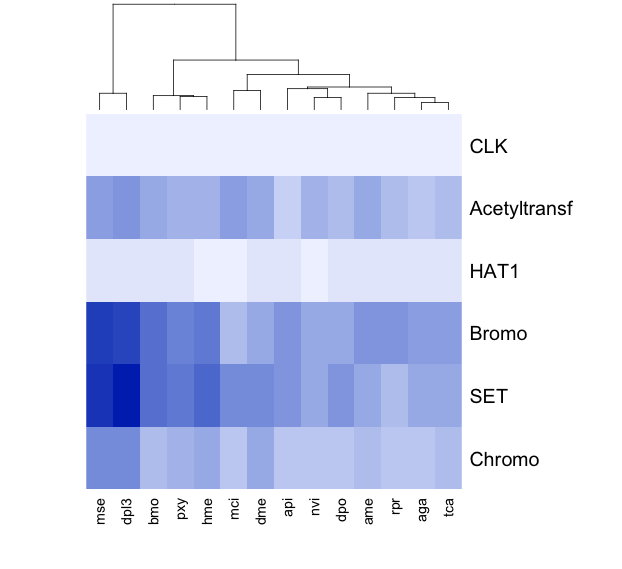
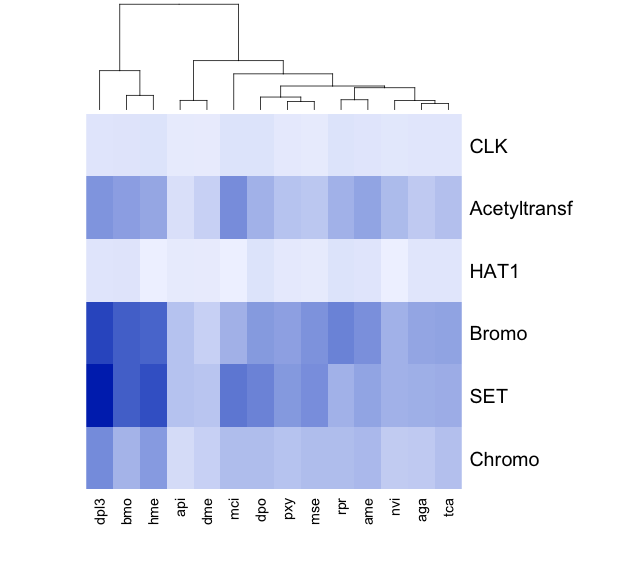
Im Praktikum wurde sich mit folgenden histonmodifzierenden Domänen auseinandergesetzt:
- SET - Protein-Lyin-Methyltransferase
- Bromodomäne - 'liest' monoacetylierte Lysine
- Chromo - bindet an Methylierte Histone
- HAT1_N - Acetyltransferase
- Acetyltransf_7 - Acetyltransferase - überträgt Acetyl-Rest
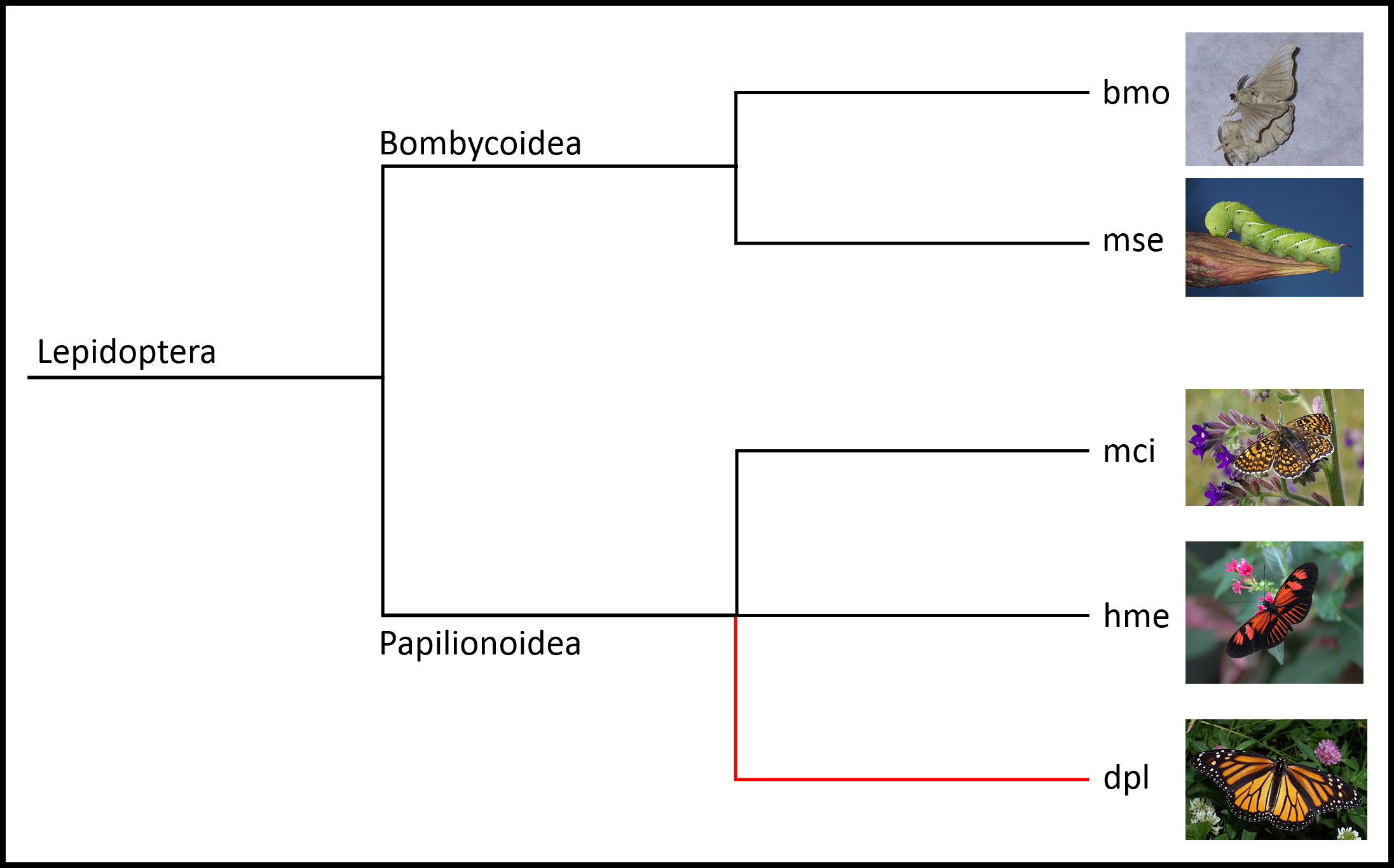
Der Danaus plexippus (Monarchfalter) ist eine im Norder der USA (und Süden Kanda) lebender Schmetterling. Aufgrund von der Kälte im Winter, dem Fressverhalten (Milkweed als Larve und Nektar als Schmetterling) und dem Brutverhalten migriert der Monarchfalter jeden Herbst nach Zentral-Mexiko. Die Herbst-Generation des Schmetterlings frisst sehr viel, speichert diese Energie in Form von Fett ab und tritt in die Diapause (Pause von Vermehrung). Er flieg im Herbst Südwest nach Mexiko um dort zu überwintern und fängt im Frühjahr an zurück Richtung Nordost zu fliegen. Die währendessen erzeugte Frühlings-Generation fliegt weiter nach Nordost bis Sie den Norder der USA erreicht haben. Die danach kommende Sommer-Generation bleibt im Norden der USA.
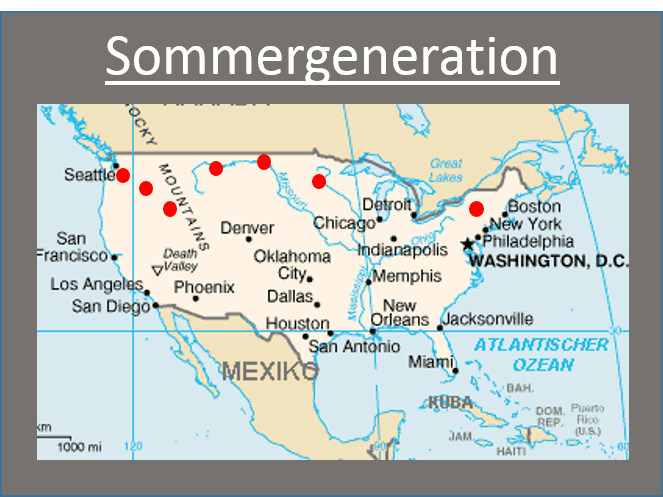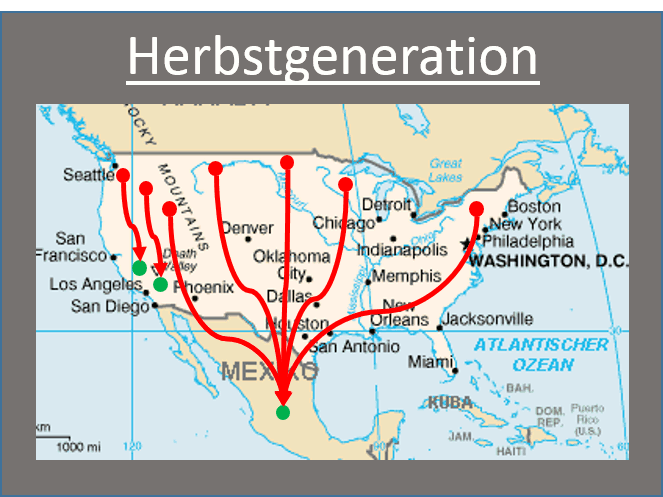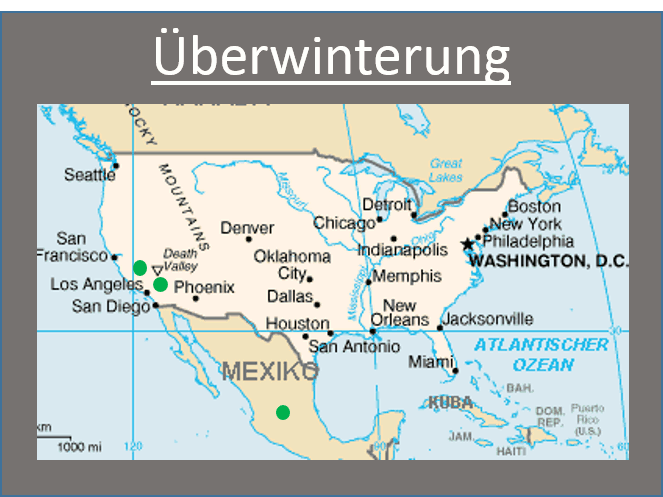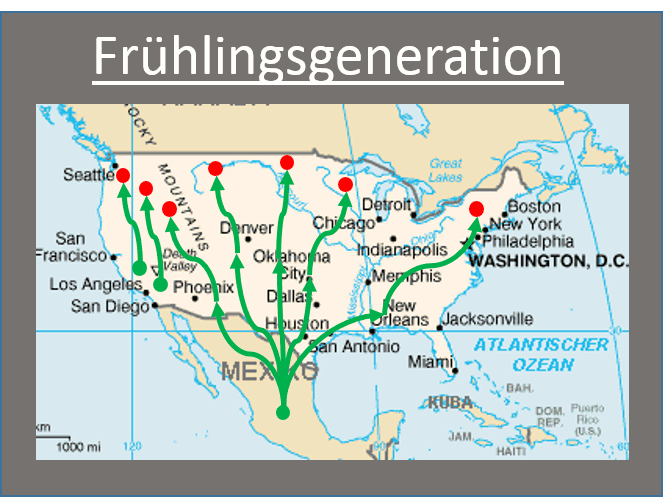
Animation: Migrationsverhalten Danaus plexippus
Es ist derzeit unklar wie die jeweilige Generation weiß wohin diese fliegen muss. Der Monarchfalter lernt das Migrationsverhalten weder von der Elter-Generation (wie bei Singvögeln), noch erinnert er sich an deinen Geburtstort (wie z.B. der Lachs) oder hat ein dementsprechendes Schwarmverhalten (wie bei Schwarmfischen). Eine Mögliche Erklärung wären Einflüsse aus der Umwelt. So könnten Beispielsweise die Tageslänge in Kombination mit dem Lichtspektrum, die Temperatur, veränderte Fressbedingungen (weniger Pflanzen im Herbst) oder der Erdmagnetismus dem Monarchfalter die vorherrschende Jahreszeit verraten und somit sein Migrationsverhalten beeinflussen.
Eine weitere Möglichkeit stellt die Epigenetik dar. So wäre es z.B. möglich das der Monarchfalter über ein generationsübergreifendes, epigenetisches Gedächtnis sein Migrationsverhalten regelt.Durch die Modifikationen und dem damit einhergehenden an- und abschalten von bestimmten Genen könnte eine Ursache für das Migrationsverhalten des Danaus plexippus bestimmt werden. Dies würde für vorhandene Modifikationen der Histone sprechen, sodass bestimmte Gene und deren Expression behindert oder gestegert werden können. So würde sich auch eine Erklärung für die Veränderung der körperlichen Merkmale im Herbst finden.
Das Ziel des Projektes ist mit Hilfe von bio-informatischen Tools mögliche Domänen zu finden, die speziell im Danaus plexippus vorkommen und einen Hinweis auf das Migrationsverhalten liefern könnten.
> Gibt es Proteine die nur in Danaus plexippus vorkommen?
> Wie sieht es bei den nahen Verwandten aus?