Auswertung
Phylogenetische Untersuchung von IFN-Gamma
Ausgehend von Sequenzalignments des humanen IFN-Gamma-Proteins gegen verschiedene Deuterostomiagenome wurde eine homologe IFN-Gamma-Version in fast allen Saeugetieren mit einigen Ausnahmen (vor allem Beuteltiere) gefunden. Stammbaum
Als Grenze der IFN-Gamma-Konservierung wurde Macropus eugenii mit einem Score von 271, E-value von 2,6e-22 und einer prozentualen Identitaet von 51,56% festgelegt.
Die genetische Karte von IFN-Gamma ist in Abbildung 2 gezeigt.

Abbildung 2: Genstruktur von humanen IFN-Gamma. Exons sind als Kaesten und Introns als Linien dargestellt. Die Zahlen geben die Anzahl an bp eines Segmentes an.
Ermittlung der PKR-Aktivatorsequenz
Es konnte die PKR-Aktivatorsequenz in verschiedenen Organismen der Deuterostomia gefunden werden. Die alignierten Sequenzen zeigten eine grosse Uebereinstimmung untereinander.
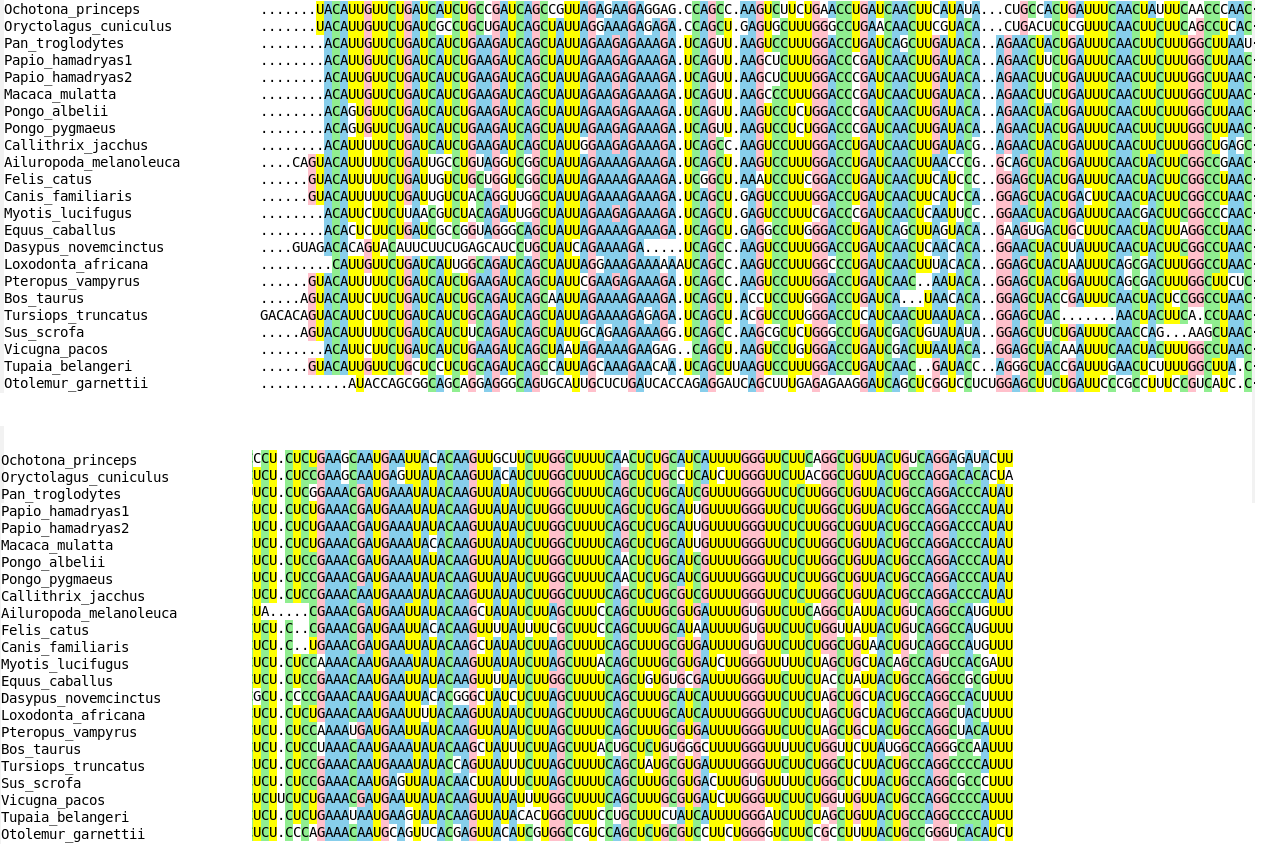 Abbildung 3: DNA-Sequenzalignment verschiedener Deuterostomiagenome gegen die humane PKR-Aktivierungssequenz von IFN-Gamma. Das Alignment wurde fuer alle Sequenzen gleichzeitig mit ClustalW durchgefuehrt.
Abbildung 3: DNA-Sequenzalignment verschiedener Deuterostomiagenome gegen die humane PKR-Aktivierungssequenz von IFN-Gamma. Das Alignment wurde fuer alle Sequenzen gleichzeitig mit ClustalW durchgefuehrt.
Strukturaufklaerung der Aktivatorsequenz

Fuer die Strukturaufklaerung der PKR-Aktivierungssequenz wurden keine Konsensus-Sequenzen verwendet, sondern lediglich ermittelte RNA-Sequenzen einzelner Tierarten. Es lag bereits ein Modell fuer die Sekundaerstruktur vor (Chalamish et al., 2009). Ausgehend von diesem Modell wurde zunaechst vesucht, mit den in der Methodik dargestellten Faltungsprogrammen die Struktur von Chalamish et al. zu generieren. Weiterhin beschreiben Chalamish et al., dass fuer eine Aktivierung von PKR ein postulierter Pseudoknoten,ein kink turn, Helices S1, S2 aS2, S3, Apikalhelix A und ein Pseudoknoten-loop M3 bzw. aM3 notwendig ist (Abbildung 4).
Abbildung 4: Postulierte Sekundaerstruktur der PKR-Aktivierungssequenz von IFN-Gamma. Bereiche fuer PKR-Aktivierung sind farbig unterlegt.
- Strukturvorhersagen mit verschiedenen Programmen:
Sekundaerstruktur nach Chalamish (human):
.......[[[[[[[((((((((..(((((..(((((.(((((((((((..(((..((((....]]]]]]].......)))).))))))))))))))))))).)))))....))).)))))(((((((...((.((((((...).))))(((....)))).))))))))).((((...(((((((.(...).))))))))))).
minimum free energy = -19,84 kcal/mol
Alternativstruktur nach Chalamish:
((((((([[[[[[[((((((((..(((((..(((((.(((((((((((...((((...)))).]]]]]]]((((...))))....)))))))))))))))).)))))....))).))))))))))))(((((.((((((...).))))(((....)))).)).....)))((((...(((((((.(...).))))))))))).
minimum free energy = -6,31 kcal/mol bzw. -19,21 kcal/mol fuer M2-Minimum
( Struktur zerlegt und Einzelstrukturen mit RNAeval berechnet)
Stockholm-Programm:
...............((((((((..(((((..(((((.....((((((....((((...))))))))))..((((...))))...............))))).)))))....))).))))).((((((.......((((......))))(((....)))....))))))..((((...(((((((.......)))))))))))...
minimum free energy = -53.20 kcal/mol (RNAeval)
RNA-fold:
...............((((((((..(((((..(((((.....((((((....((((...))))))))))..((((...))))...............))))).)))))....))).))))).((((((.......((((......))))(((....)))....))))))..((((...(((((((.......)))))))))))...
minimum free energy = -53.20 kcal/mol
pknotsRG
........[[[[[[.{{{{{.]]]]]]...}}}}}((((((.((((((....((((...)))))))))).(((.((.....(((........)))....))...)))....))))))..(((((((((.......((((......))))(((....)))....)))))))))(((...(((((((.......))))))))))....
minimum free energy = -56.30 kcal/mol
vsfold5.
37 Grad/ Kuhn aus.
.....(((((((((.[[[[[.)))).....]]]]].......[[[[[[[...(((.....)))...(((.....)))...))))).]]]]]]].(((.....)))......(((.....))).(((((.......((((......))))(((....)))....)))))...(((((((....(((.......))).)))))))....
minimum free energy = -53.34 kcal/mol
37 Grad Kuhn 8.
.........(((((.[[[[[.)))))....]]]]].(((((.((((((((..........)).))))))....(((..((((((..............))).)))..))).)))))...(((((((((..(((((.....)))))...((((....))))...)))))))))(((((((...(((.......))))))))))....
minimum free energy = -31.62 kcal/mol
Der Versuch, mithilfe von Faltungsprogrammen die postulierte Struktur zu generieren, schlug fehl. Kein verwendetes Programm konnte die Struktur erzeugen. Manche Programme bauten keinen Pseudoknoten ein, andere Programme, welche speziell fuer die Vorhersage von Pseudoknoten vorgesehen sind, konnten Pseudoknoten erzeugen, jedoch nicht an der erwarteten Position. Die berechnete freie Energie fuer die postulierte Struktur betrug -19,84 kcal/mol bzw. -6,31 oder -19,21 kcal/mol fuer die alternative Struktur. Dagegen konnten mehrere Strukturen, teilweise ohne oder mit Pseudoknoten an einer anderen Stelle, ermittelt werden, welche einen negativeren Betrag fuer die freie Energie lieferten. Die Berechnung der freien Energie erfolgte mit den Programmen, welche auch die Faltung durchfuehrten. Es entstand ein Widerspruch zwischen der postulierten Struktur und den mittels Faltungsprogrammen ermittelten Strukturen. Beispielsweise bewertete RNAeval die von RNAfold erzeugte Struktur ohne Pseudoknoten mit einer freien Energie von -53,2 kcal/mol als stabiler im Vergleich zu der von Chalamish postulierten Struktur mit Pseudoknoten. Demzufolge ist entweder die postulierte Struktur falsch oder die Programme arbeiten fehlerhaft.




Abbildung 5: Sekundaerstrukturvorhersagen mittels verschiedenen Faltungsprogrammen.
- Mit dem Programm R-Coffee wurden die erhaltenen RNA-Sequenzen aligniert mit Beruecksichtigung von Strukturkonservierung. Dieses Alignment diente als Ausgangsdatei fuer das Programm RNAalifold. Dieses Programm erstellte anhand des von R-Coffee erzeugten Alignments eine Konsensus-Sequenz mit dazugehoeriger Sekundaerstruktur.
R-Coffee Alignment mit anschliessender Erzeugung einer Konsensus Sequenz mit Sekundaerstruktur durch RNAalifold:
___________ACAUUGUUCUGAUCAUCUGCAGAUCAGCUAUUAGAAAAGA_AAGAUCAGCU_AAGUCCU_UUGGACCUGAUCAACUUAA
UACAGGAGCUACUGAUUUCAACUACUUUGGCCUAACUC_UCUCCGAAACAAUGAAUUAUACAAGUUAUAUCUUGGCUUUUCAGCUCUGCAUCAUU_UUGG
GUUCUUCUG_GCUGUUACUGCCAGGCCCCAUUU
...................((((((.......)))))).............................................................................................(((((....(((........((((((.....))))))..........))).....)))))....(((..((.......)).)))........
minimum free energy = -9.20 kcal/mol
- Das Programm locaRNA erstellte aus den verschiedenen Aktivatorsequenzen ebenfalls ein strukturbasiertes Alignment und erzeugte parallel dazu eine Sekundaerstruktur. Alignment nach locaRNA / Sekundaerstruktur
-
Es wurde ein weiteres Programm, RMdetect, zur Strukturvorhersage von kink-turn Motifen verwendet. Fuer das verwendete Alignment verschiedener Deuterostomia-PKR-Aktivatorsequenzen wurde von RMdetect ein kink turn Motif in der Schimpansensequenz vorhergesagt, welcher dem kink turn der von Chalamish et al. postulierten Struktur entsprach. Ergebnisse
In drei weiteren Tierarten wurde ein anderes kink-turn Motif gefunden. Auffallend war, dass die Sequenzbereiche der kink-turn Motife in verschiedenen Arten identisch waren, das Programm RMdetect diese aber nicht erkannte. So zeigte sich beispielsweise in allen Primaten die gleiche Sequenz innerhalb des kink turn Motifs. Wir vermuten daher, dass der kink turn in allen Primaten vorhanden ist und das Programm RMdetect unvollstaendige Ergebnisse lieferte. Auch ist nicht ersichtlich, weshalb gerade Pferd, Elefant und Lama den kink turn #2 haben sollten und alle anderen Organismen nicht.
 Abbildung 6: Alignment der PKR-Aktivatorsequenz verschiedener Deuterostomiae mit ermittelten kink turns. Die Sequenz des kink turn #1 wurde in 7 Spezies (Primaten,Gepard) und die des kink turn #2 nur in Elefant und Pferd identifiziert.
Abbildung 6: Alignment der PKR-Aktivatorsequenz verschiedener Deuterostomiae mit ermittelten kink turns. Die Sequenz des kink turn #1 wurde in 7 Spezies (Primaten,Gepard) und die des kink turn #2 nur in Elefant und Pferd identifiziert.
Zusammenfassend konnte festgestellt werden, dass die von uns mit bioinformatischen Methoden ermittelten Strukturen nicht in Einklang mit der von Chalamish et al. postulierten Struktur zu bringen sind. Es konnte der postulierte Pseudoknoten zu keinem Zeitpunkt generiert werden und die ermittelten kink-turn Motife stimmen nur sehr eingeschraenkt ueberein. Die ermittelte freie Energie der postulierten Struktur betrug mit -19,84 kcal/mol bzw. -6,31/-19,21 kcal/mol nur einen Bruchteil der von dem Programm RNAfold ermittelten freien Energie mit einem Betrag von -53,2 kcal/mol.
Da die bioinformatischen Programme stets andere Strukturen vorschlagen, fragt sich, ob die Struktur von Chalamish et. al korrekt ist. Zumindest basiert deren Strukturvorschlag teilweise auf experimentellen Daten. Die bioinformatischen Programme liefern zwar bessere freie Energiewerte, aber erzeugen immer wieder andere Strukturen und koennen keine Uebereinstimmung zeigen. Die von uns noch am meisten favorisierte Struktur von RNAfold kommt ohne Pseudoknoten aus. Jedoch konnte nur fuer einen Teil der fuer das Alignment verwendeten Sequenzen mit dieser Struktur ein negativer Betrag fuer die freie Energie erhalten werden, sodass diese Struktur vermutlich nicht eine wesentliche Rolle bei der PKR-Aktivierung spielt, da lediglich dem Menschen sehr nahe Verwandte (Primaten) stark negative Betraege fuer die freie Energie lieferten.
Da die Tertiaerstruktur der gefalteten RNA nicht von den Faltungsprogrammen beruecksichtigt wird, laesst sich keine Aussage treffen, inwieweit die Tertiaerstruktur die Gesamtstruktur beeinflusst. Jedoch ist zu vermuten, dass eine spezifische Sekundaerstruktur durch die Tertiaerstruktur soweit stabilisert wird, sodass eine Sekundaerstruktur vorherrscht, dessen guenstiger Energiebetrag nicht allein durch die Sekundaerstruktur beschrieben werden kann, was das Scheitern der Programme erklaeren wuerde. Beispielsweise muesste die Tertiaerstrukturfaltung 63% der Energie liefern, damit die postulierte Struktur von Chalamish mit ihrer freien Energie von -19,84 kcal/mol auf einen aehnlich negativen Wert kommt wie RNAfold. Es ist von grosser Bedeutung, dass die 3D-Struktur der PKR-Aktivatorsequenz zumindest in einem Teil des Molekuels ueber die Tierarten hinweg konserviert vorliegt. Denn so bleibt die Funktion des Regulationselementes vorhanden, welches im Vergleich zur Abwesenheit dieses Regulationselements einen evolutionaeren Vorteil mit sich bringt.





 Abbildung 3: DNA-Sequenzalignment verschiedener Deuterostomiagenome gegen die humane PKR-Aktivierungssequenz von IFN-Gamma. Das Alignment wurde fuer alle Sequenzen gleichzeitig mit ClustalW durchgefuehrt.
Abbildung 3: DNA-Sequenzalignment verschiedener Deuterostomiagenome gegen die humane PKR-Aktivierungssequenz von IFN-Gamma. Das Alignment wurde fuer alle Sequenzen gleichzeitig mit ClustalW durchgefuehrt.

 Abbildung 6: Alignment der PKR-Aktivatorsequenz verschiedener Deuterostomiae mit ermittelten kink turns. Die Sequenz des kink turn #1 wurde in 7 Spezies (Primaten,Gepard) und die des kink turn #2 nur in Elefant und Pferd identifiziert.
Abbildung 6: Alignment der PKR-Aktivatorsequenz verschiedener Deuterostomiae mit ermittelten kink turns. Die Sequenz des kink turn #1 wurde in 7 Spezies (Primaten,Gepard) und die des kink turn #2 nur in Elefant und Pferd identifiziert.