
| Sequence ID | Banth.0 |
|---|---|
| Location | 4,650,323 – 4,650,443 |
| Length | 120 |
| Max. P | 0.939649 |

| Location | 4,650,323 – 4,650,443 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 62.82 |
| Mean single sequence MFE | -36.86 |
| Consensus MFE | -18.79 |
| Energy contribution | -16.58 |
| Covariance contribution | -2.21 |
| Combinations/Pair | 1.72 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.51 |
| SVM decision value | 1.30 |
| SVM RNA-class probability | 0.939649 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>Banth.0 4650323 120 + 5227293/0-120 GUCAGAUGCUCUACCAACUGAGCUAAUGGCUCAUUUUGAAAGUGGUGCCGGCUAGAGGACUUGAACCCCCAACCUACUGAUUACAAGUCAGUUGCUCUACCAAUUGAGCUAAGCCGGCUU ((((...((((........))))...))))(((((.....))))).(((((((..(((..(((......))))))((((((.....)))))).((((........))))..))))))).. ( -38.10) >Bcere.0 4649302 120 + 5224283/0-120 GUCAGAUGCUCUACCAACUGAGCUAAUGGCUCAUUUUGAAAGUGGUGCCGGCUAGAGGACUUGAACCCCCAACCUACUGAUUACAAGUCAGUUGCUCUACCAAUUGAGCUAAGCCGGCUU ((((...((((........))))...))))(((((.....))))).(((((((..(((..(((......))))))((((((.....)))))).((((........))))..))))))).. ( -38.10) >Bsubt.0 3171336 119 + 4214630/0-120 GGCUGAGGUUUUACCA-CUAAACUACACCCGCAAUUUUUAUUUGGGGCGAUUGAUGGGAAUCGAACCCACGAAUGCCAGAGCCACAAUCUGGUGCGUUAACCACUUCGCCACAACCGCCA (((((.(((((.....-..))))).))..............(((.(((((.((..(((.......)))(((...((((((.......)))))).)))....))..))))).)))..))). ( -31.10) >Blich.0 3115876 117 + 4222334/0-120 GGCUGAGGUUUUACCA-CUAAACUACACCCGCAUUUCGCA--UGGGGCGGUUGAUGGGAAUCGAACCCACGAAUGCCAGAGCCACAAUCUGGUGCGUUAACCACUUCGCCACAACCGCCG ((.((.(((((.....-..))))).)).))((.....)).--...((((((((.((((((........(((...((((((.......)))))).))).......))).))))))))))). ( -38.16) >Bhalo.0 2948622 113 + 4202352/0-120 AGGUGGUGUUCUACCA-CUGAACUACUUCCGCCUAUUG---GCUGGGCUAGCU---GGAUUCGAACCAGCGCAUCACGGAGUCAAAGUCCGUUGCCUUACCGCUUGGCUAUAGCCCAAUA .((.(((((((.....-..)))).))).))(((....)---))((((((((((---((.......))))).....(((((.......))))).(((.........)))..)))))))... ( -40.40) >Bclau.0 2627182 112 + 4303871/0-120 AGGUGAUGUUCUACCA-CUGAACUACUUCCGCAUAU-G---GCUGGGCUACCU---GGAUUCGAACCAGGGAAUCACGGAAUCAAAAUCCGUUGCCUUACCGCUUGGCUAUAGCCCAUUA .((....((((.....-..)))).....))((.(((-(---((..(((..(((---((.......))))).....(((((.......))))).........)))..))))))))...... ( -35.30) >consensus GGCUGAUGUUCUACCA_CUGAACUACUCCCGCAUUUUG_A_GUGGGGCCAGCUA__GGAAUCGAACCCACGAAUCACAGAGUCAAAAUCAGUUGCCUUACCAAUUGAGCAAAGCCCGCUA (((((..((((........))))..)))))...............((((.(((......................(((((.......))))).((((........))))..))).)))). (-18.79 = -16.58 + -2.21)

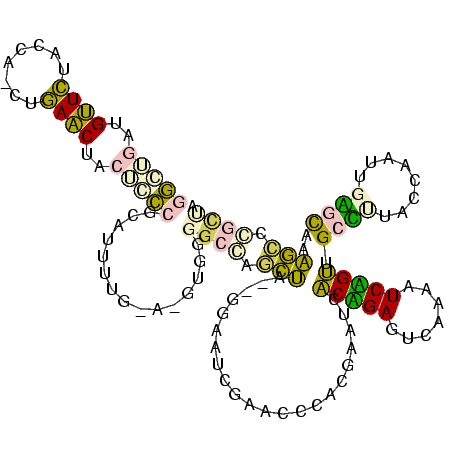
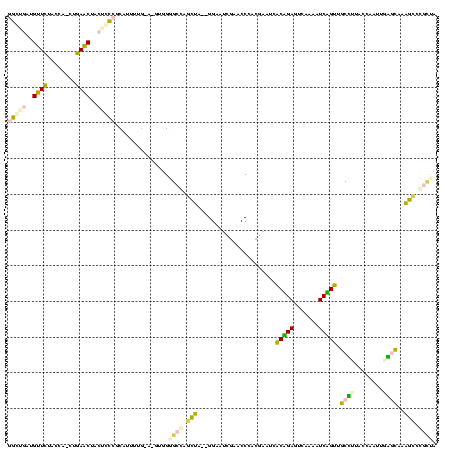
| Location | 4,650,323 – 4,650,435 |
|---|---|
| Length | 112 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 62.41 |
| Mean single sequence MFE | -36.56 |
| Consensus MFE | -25.19 |
| Energy contribution | -22.92 |
| Covariance contribution | -2.27 |
| Combinations/Pair | 1.70 |
| Mean z-score | -1.21 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.52 |
| SVM RNA-class probability | 0.768782 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>Banth.0 4650323 112 + 5227293/120-240 GCG--------ACUUCAAAAUGGUGGAUCGGGACGGAAUCGAACCGCCGACACGAGGAUUUUCAGUCCUCUGCUCUACCGACUGAGCUACCGAGCCUUUAUGUAAAAAUGGCGGUCCCGA .(.--------(((.......))).).(((((((((..(((......)))...((((((.....)))))).((((........))))..))..(((.............))).))))))) ( -34.92) >Bcere.0 4649302 112 + 5224283/120-240 GCG--------ACUUCAAAAUGGUGGCUCGGGACGGAAUCGAACCGCCGACACGAGGAUUUUCAGUCCUCUGCUCUACCGACUGAGCUACCGAGCCUUUAUGUAAAAAUGGCGGUCCCGA (.(--------(((......(((((((((((..(((..(((......)))...((((((.....)))))).......))).))))))))))).(((.............))))))).).. ( -40.02) >Bsubt.0 3171336 115 + 4214630/120-240 AAGCAU-----AUUGAUAAAUGGUGGCUCGGGACGGAAUCGAACCGCCGACACACGGAUUUUCAGUCCGUUGCUCUACCAACUGAGCUACCGAGCCUAAGUAUUUAAAUGGCGGUCCGGA ..((.(-----(((..((((((.((((((((((((((...(((...(((.....)))...)))..))))))((((........))))..)))))))..).))))))))))))........ ( -37.90) >Blich.0 3115876 118 + 4222334/120-240 CAAUAUAUAGAAUUGUAAAAUGGUGGCUCGGGACGGAAUCGAACCGCCGACACACGGAUUUUCAGUCCGUUGCUCUACCAACUGAGCUACCGAGCCUU--UGUUAAAAUGGCGGUCCGGA .........(.(((((..((..(.(((((((((((((...(((...(((.....)))...)))..))))))((((........))))..))))))).)--..))......))))).)... ( -37.10) >Bhalo.0 2948622 112 + 4202352/120-240 UCA--------AAAGCAAAAUGGGGCGACUGAUGGGAAUCGAACCCACGAGUGCCGGAGCCACAAUCCGGUGCGUUAACCACUUCGCCACAGUCGCCAAGACUAUAAAUGGUGGAGGGGG ...--------..(((....((((.....((((....))))..))))...(..(((((.......)))))..))))..((.((((((((.((((.....)))).....)))))))).)). ( -37.60) >Bclau.0 2627182 97 + 4303871/120-240 UUU--------G---------GUUGCGGGGGACGG-AUUUGAACCGCCGACCUUCGGGUUAUGAGCCCGACGAGCUA-CCAGACUGCUCCACCCCGCGUCGCUACUAAUGAAUAUG---- ..(--------(---------((((((((((.(((-.......))).).....((((((.....)))))).((((..-.......))))..)))))))..))))............---- ( -31.80) >consensus GCG________ACUGCAAAAUGGUGGCUCGGGACGGAAUCGAACCGCCGACACACGGAUUUUCAGUCCGCUGCUCUACCAACUGAGCUACCGAGCCUUUAUGUAAAAAUGGCGGUCCGGA ........................(((((((.......(((......)))...((((((.....)))))).((((........))))..)))))))........................ (-25.19 = -22.92 + -2.27)

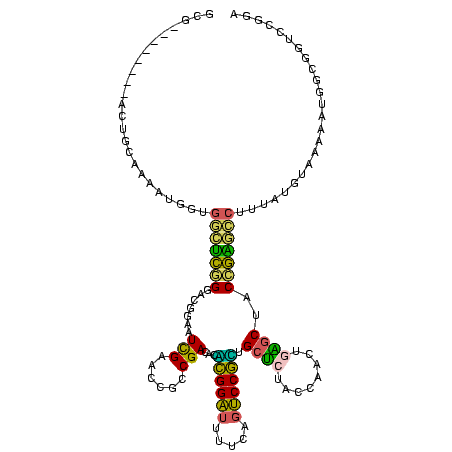
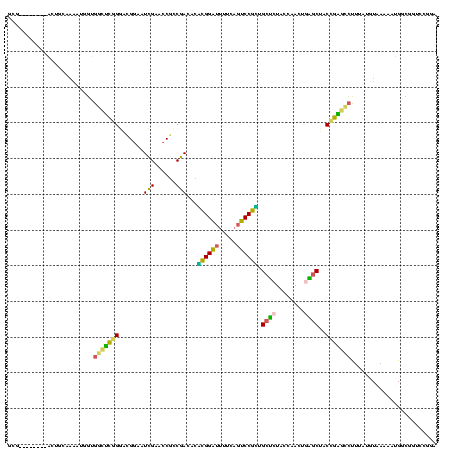
| Location | 4,650,323 – 4,650,441 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 52.84 |
| Mean single sequence MFE | -32.13 |
| Consensus MFE | -10.98 |
| Energy contribution | -11.68 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.71 |
| Mean z-score | -1.25 |
| Structure conservation index | 0.34 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.762943 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>Banth.0 4650323 118 + 5227293/440-560 AAAAUAUUUAAUUAAGAAAAAAUGGAGGAGGUAGAGGGAUUCGAACCCCCGCGCGACUCUCGC-CGCCUGUCGGUU-UUCAAGACCGAUCCCUUCAGCCGAACUUGGGUAUACCUCCGAG ..........................(((((((((((((...........(((((.....)).-)))...((((((-.....))))))))))))..(((.......))).)))))))... ( -34.90) >Bcere.0 4649302 118 + 5224283/440-560 AAAAUAUUUACUUUAGAAAAAAUGGAGGAGGUAGAGGGAUUCGAACCCCCGCGCGACUCUCGC-CGCCUGUCGGUU-UUCAAGACCGAUCCCUUCAGCCGAACUUGGGUAUACCUCCGAG ..........................(((((((((((((...........(((((.....)).-)))...((((((-.....))))))))))))..(((.......))).)))))))... ( -34.90) >Bsubt.0 3171336 105 + 4214630/440-560 UAGGAAUAUGU----------AUGGCGGAGGAAGAGGGAUUCGAACCCCCGCG-GGCUUUGAC-ACCCUGUCGGUU-UUCAAGACCGAUCCCUUCAGCCAGACUUGGGUAUUCCUCCG-- .((((((((.(----------((((((((((...((((..((((((((....)-)).))))).-.))))(((((((-.....))))))).))))).))))....)).))))))))...-- ( -42.50) >Blich.0 3115876 113 + 4222334/440-560 CAAAAUAAUAAAU---AAUAUAUGGCGGAGGAAGAGGGAUUCGAACCCCCGCG-GGCUUUGACACCCCUGUCGGUU-UUCAAGACCGAUCCCUUCAGCCAGACUUGGGUAUUCCUCCG-- .............---.........((((((((((((((.((((((((....)-)).)))))........((((((-.....))))))))))))..(((.......))).))))))))-- ( -36.30) >Bhalo.0 2948622 102 + 4202352/440-560 AUAAA--GUAA----------AUGGAUGUUGU--UUUUUUCUAAAGAAGCAGGACC----UCUCCUCUUCUCGCUUCCUCUCAGACGCUAUCAUGCGUCGAGGUUACUCGUUGCUUUCUC ..(((--((((----------..((.......--..........(((((.((((..----..))))))))).....((((...(((((......)))))))))...))..)))))))... ( -25.70) >Bclau.0 2627182 101 + 4303871/440-560 AUUAAUGAUGA----------AAUUUUGCUGUGCUAUUGUUUGAAAGAGGAUAACUGUCUCUUCCUCUGCUUGCUCAUUCAGGGAUGUU-UGGUGCGUCGAGACAACUCACU-------- ...........----------.........(((...((((((...((((((...........)))))).((((......))))(((((.-....))))).))))))..))).-------- ( -18.50) >consensus AAAAAAUAUAA__________AUGGAGGAGGUAGAGGGAUUCGAACCCCCGCG_GACUCUCAC_CCCCUGUCGGUU_UUCAAGACCGAUCCCUUCAGCCGAACUUGGGUAUUCCUCCG__ ..........................(((((((..(((........)))....................(((((((......))))))).....................)))))))... (-10.98 = -11.68 + 0.70)
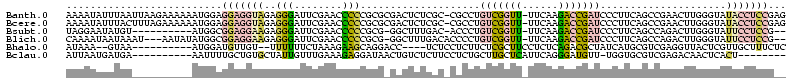
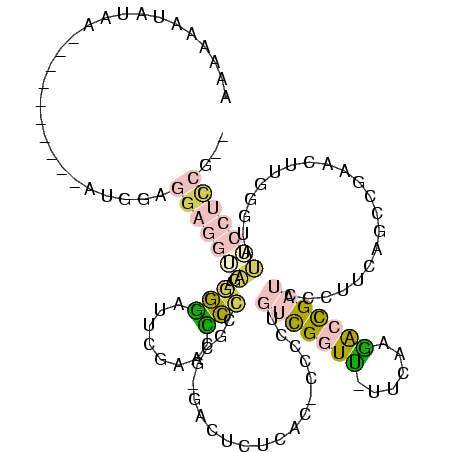

| Location | 4,650,323 – 4,650,443 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 63.20 |
| Mean single sequence MFE | -40.97 |
| Consensus MFE | -25.74 |
| Energy contribution | -26.63 |
| Covariance contribution | 0.90 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.20 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.42 |
| SVM RNA-class probability | 0.729226 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>Banth.0 4650323 120 + 5227293/600-720 AGCUAGGCUAGAGCGUAUGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCGCCGCUACCAUAAAGGACCUUUAGCUCAGCUGGUUAGAGCAGACGGCUCAUAACCGUCCGGUCGU ((((..(((((((..((((((((((....))))((.((((((........))))))))...))))))......)))))))..))))(((((((((.....)))).))))).......... ( -45.90) >Bcere.0 4649302 119 + 5224283/600-720 AGCU-GGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCGCCGCUACCAUAAAGGACCUUUAGCUCAGCUGGUUAGAGCAGACGGCUCAUAACCGUCCGGUCGU (((.-.(((.((((....(((((......(((.((.((((((........))))))))..)))......)))))....)))))))..))).((.(.(((((.......))))).).)).. ( -46.70) >Bsubt.0 3171336 117 + 4214630/600-720 AGCU-GGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCGCCGCUACC--AAUGGACCUUUAGCUCAGUUGGUUAGAGCAGACGGCUCAUAACCGUCCGGUCGU (((.-.(((.((((....(((((((....(((.((.((((((........))))))))..))).--.)))))))....)))))))..))).((.(.(((((.......))))).).)).. ( -47.30) >Blich.0 3115876 117 + 4222334/600-720 AGCU-GGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUUCCCUCCGCCGCUAUU--AAAGGACCUUUAGCUCAGUUGGUUAGAGCAGACGGCUCAUAACCGUCCGGUCGU (((.-.(((.((((....(((((....((....)).((((((........))))))........--...)))))....)))))))..))).((.(.(((((.......))))).).)).. ( -44.70) >Bhalo.0 2948622 110 + 4202352/600-720 AGUCUGGCUGAAGGGAUCGGUCUUGAAAACCGA--CAGGCGGGUUAAACCGCGCGGGGGUU-----CGAAUCCCUCCUCCUCCGCCACUUUA---CAACAACAUCCAUUUACUUUAUAAC ....((((.((.((((..((..(((((..(((.--...((((......)))).)))...))-----)))..)).))))..)).)))).....---......................... ( -29.90) >Bclau.0 2627182 101 + 4303871/600-720 AGUCUGGCUGAAGGGAUCGGUCUUGAAAACCGA--CAGGCGGGUUACACCGCGCGGGGGUU-----CGAAUCCCUCUUCCUCCGCCAUUCU-----------UUUGAUUUAUUCU-UAAC ((..((((.((.(((((((((.......)))))--...((((......))))..(((((..-----.....))))))))))).))))..))-----------.............-.... ( -31.30) >consensus AGCU_GGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCGCCGCUACC__AAAGGACCUUUAGCUCAGCUGGUUAGAGCAGACGGCUCAUAACCGUCCGGUCGU .....((((.((((....(((((..........(((.(((((......)))).).)))...........)))))....))))))))(((((((((.....)))).))))).......... (-25.74 = -26.63 + 0.90)
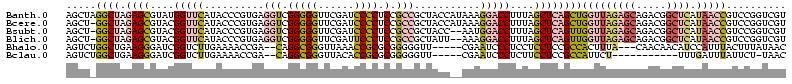
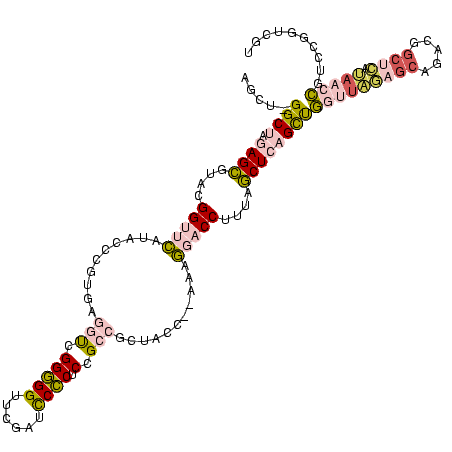
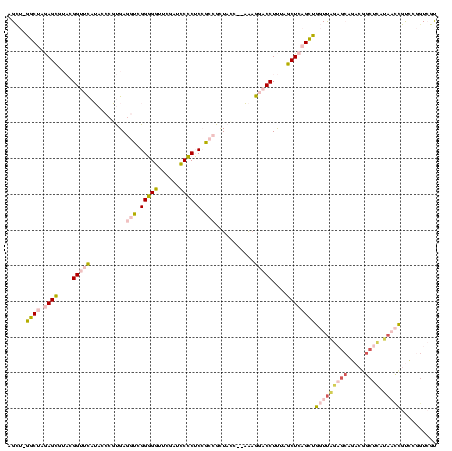
| Location | 4,650,323 – 4,650,443 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 65.56 |
| Mean single sequence MFE | -45.12 |
| Consensus MFE | -24.12 |
| Energy contribution | -26.65 |
| Covariance contribution | 2.53 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.874299 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
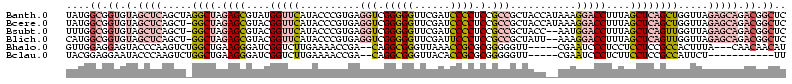
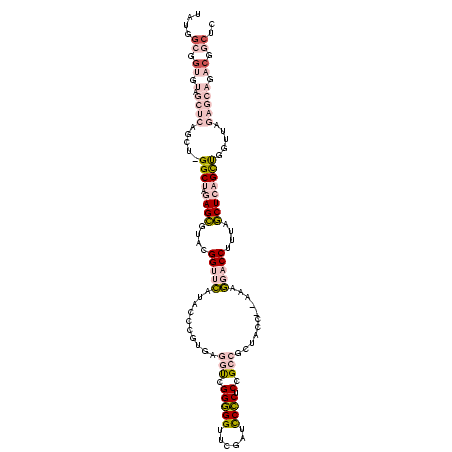
>Banth.0 4650323 120 + 5227293/616-736 UAUGGCGGUGUAGCUCAGCUAGGCUAGAGCGUAUGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCGCCGCUACCAUAAAGGACCUUUAGCUCAGCUGGUUAGAGCAGACGGCUC ...(((.((.(.((((((((((.((.((((....(((((......(((.((.((((((........))))))))..)))......)))))....)))))))))))).))))).)).))). ( -47.80) >Bcere.0 4649302 119 + 5224283/616-736 UAUGGCGGUGUAGCUCAGCU-GGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCGCCGCUACCAUAAAGGACCUUUAGCUCAGCUGGUUAGAGCAGACGGCUC ...(((.((.(.(((((((.-.(((.((((....(((((......(((.((.((((((........))))))))..)))......)))))....)))))))..))).))))).)).))). ( -49.90) >Bsubt.0 3171336 117 + 4214630/616-736 UUUGGCGGUGUAGCUCAGCU-GGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCGCCGCUACC--AAUGGACCUUUAGCUCAGUUGGUUAGAGCAGACGGCUC ...(((.((.(.(((((((.-.(((.((((....(((((((....(((.((.((((((........))))))))..))).--.)))))))....)))))))..))).))))).)).))). ( -50.50) >Blich.0 3115876 117 + 4222334/616-736 CAUGGCGGUGUAGCUCAGCU-GGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUUCCCUCCGCCGCUAUU--AAAGGACCUUUAGCUCAGUUGGUUAGAGCAGACGGCUC ...(((.((.(.(((((((.-.(((.((((....(((((....((....)).((((((........))))))........--...)))))....)))))))..))).))))).)).))). ( -47.90) >Bhalo.0 2948622 110 + 4202352/616-736 GUUGGAGGAGUACCCAAGUCUGGCUGAAGGGAUCGGUCUUGAAAACCGA--CAGGCGGGUUAAACCGCGCGGGGGUU-----CGAAUCCCUCCUCCUCCGCCACUUUA---CAACAACAU (.((((((((..(((...((.....)).))).(((((.......)))))--...((((......))))..(((((..-----.....))))))))))))).)......---......... ( -37.40) >Bclau.0 2627182 102 + 4303871/616-736 UACGGAGGAAUACCCAAGUCUGGCUGAAGGGAUCGGUCUUGAAAACCGA--CAGGCGGGUUACACCGCGCGGGGGUU-----CGAAUCCCUCUUCCUCCGCCAUUCU-----------UU ..((((((((..(((...((.....)).))).(((((.......)))))--...((((......))))..(((((..-----.....))))))))))))).......-----------.. ( -37.20) >consensus UAUGGCGGUGUAGCUCAGCU_GGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCGCCGCUACC__AAAGGACCUUUAGCUCAGCUGGUUAGAGCAGACGGCUC ....((.((.(.((((.....((((.((((....(((((..........(((.(((((......)))).).)))...........)))))....)))))))).....))))).)).)).. (-24.12 = -26.65 + 2.53)

Generated by rnazCluster.pl (part of RNAz $RNAz::rnazVersion) on Thu Apr 6 17:15:09 2006