
| Sequence ID | Blich.0 |
|---|---|
| Location | 36,244 – 36,360 |
| Length | 116 |
| Max. P | 0.938544 |

| Location | 36,244 – 36,360 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.76 |
| Mean single sequence MFE | -42.57 |
| Consensus MFE | -39.80 |
| Energy contribution | -39.08 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.29 |
| SVM RNA-class probability | 0.938544 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
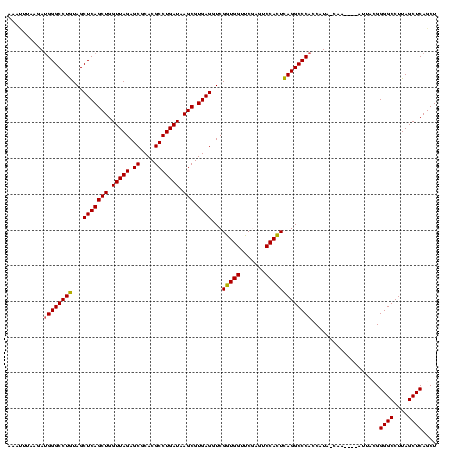
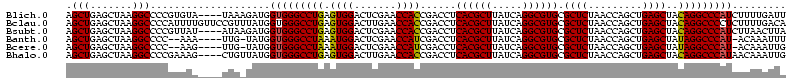
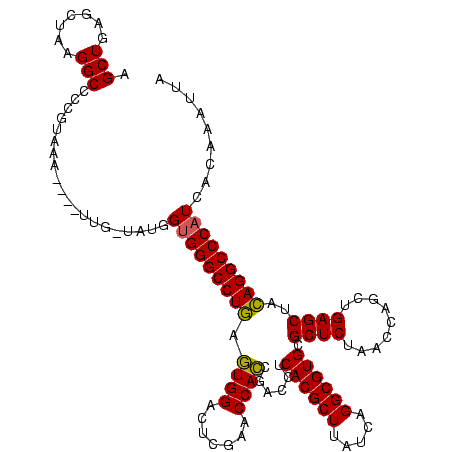
>Blich.0 36244 116 + 4222334/0-120 AAUCAAAAGAUGGGCCUGUAGCUCAGCUGGUUAGAGCGCACGCCUGAUAAGCGUGAGGUCGGUGGUUCGAGUCCACUCAGGCCCACCAUCUUUA----UACACGGGGCCUUAGCUCAGCU .....(((((((((((((...(((((((.(((((.((....))))))).))).))))...(((((.......)))))))))))))...))))).----......((((....)))).... ( -42.90) >Bclau.0 935484 120 + 4303871/0-120 UGUCAAAAGAGGGGCCUGUAGCUCAGCUGGUUAGAGCGCACGCCUGAUAAGCGUGAGGUCGGUGGUUCAAGUCCACUCAGGCCCACCAUAAACGGAACAAAAUGGGGCCUUAGCUCAGCU ........((((((((((...(((((((.(((((.((....))))))).))).))))...(((((.......)))))))))))).........((..(......)..))....))).... ( -41.80) >Bsubt.0 31916 116 + 4214630/0-120 UAAGUUAAGAUGGGCCUGUAGCUCAGCUGGUUAGAGCGCACGCCUGAUAAGCGUGAGGUCGGUGGUUCGAGUCCACUCAGGCCCACCAUCUUAU----AUAACGGGGCCUUAGCUCAGCU ..((((.((..((.(((((......(((.(((((.((....))))))).)))(((((((.(((((..((((....))).)..))))))))))))----...))))).))....)).)))) ( -44.60) >Banth.0 30764 112 + 5227293/0-120 AAAUUUGU-AUGGGCCUAUAGCUCAGCUGGUUAGAGCGCACGCCUGAUAAGCGUGAGGUCGAUGGUUCGAGUCCAUUUAGGCCCACCAUA-CAA----UUU--GGGGCCUUAGCUCAGCU ....((((-(((((((((...(((((((.(((((.((....))))))).))).))))...(((((.......)))))))))))))...))-)))----...--.((((....)))).... ( -39.80) >Bcere.0 30743 112 + 5224283/0-120 CAAUUUGU-AUGGGCCUAUAGCUCAGCUGGUUAGAGCGCACGCCUGAUAAGCGUGAGGUCGAUGGUUCGAGUCCAUUUAGGCCCACCAUA-CAA----CUU--GGGGCCUUAGCUCAGCU (((.((((-(((((((((...(((((((.(((((.((....))))))).))).))))...(((((.......)))))))))))))...))-)))----.))--)((((....)))).... ( -42.10) >Bhalo.0 119355 116 + 4202352/0-120 CAAUUUGUUAUGGGCCUGUAGCUCAGCUGGUUAGAGCGCACGCCUGAUAAGCGUGAGGUCGGUGGUUCAAGUCCACUCAGGCCCACCAUAACAG----CUUUCGGGGCCUUAGCUCAGCU .....(((((((((((((...(((((((.(((((.((....))))))).))).))))...(((((.......)))))))))))))...)))))(----((....((((....))))))). ( -44.20) >consensus AAAUUUAAGAUGGGCCUGUAGCUCAGCUGGUUAGAGCGCACGCCUGAUAAGCGUGAGGUCGGUGGUUCGAGUCCACUCAGGCCCACCAUA_CAA____AUUACGGGGCCUUAGCUCAGCU ..........((((((((...(((((((.(((((.((....))))))).))).))))...(((((.......)))))))))))))...................((((....)))).... (-39.80 = -39.08 + -0.72)


| Location | 36,244 – 36,360 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.76 |
| Mean single sequence MFE | -37.60 |
| Consensus MFE | -34.37 |
| Energy contribution | -33.65 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.25 |
| Structure conservation index | 0.91 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.516549 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
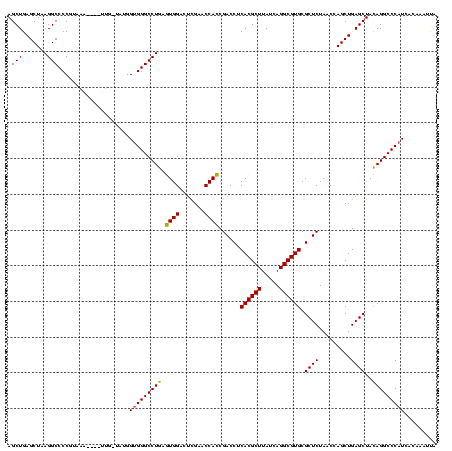
>Blich.0 36244 116 + 4222334/0-120 AGCUGAGCUAAGGCCCCGUGUA----UAAAGAUGGUGGGCCUGAGUGGACUCGAACCACCGACCUCACGCUUAUCAGGCGUGCGCUCUAACCAGCUGAGCUACAGGCCCAUCUUUUGAUU .((.(.((....)).).))...----..((((.((((((((((.((((.......))))......((((((.....)))))).((((.........))))..)))))))))))))).... ( -39.10) >Bclau.0 935484 120 + 4303871/0-120 AGCUGAGCUAAGGCCCCAUUUUGUUCCGUUUAUGGUGGGCCUGAGUGGACUUGAACCACCGACCUCACGCUUAUCAGGCGUGCGCUCUAACCAGCUGAGCUACAGGCCCCUCUUUUGACA ....((((.(((......))).)))).(((...((.(((((((.((((.......))))......((((((.....)))))).((((.........))))..)))))))..))...))). ( -37.20) >Bsubt.0 31916 116 + 4214630/0-120 AGCUGAGCUAAGGCCCCGUUAU----AUAAGAUGGUGGGCCUGAGUGGACUCGAACCACCGACCUCACGCUUAUCAGGCGUGCGCUCUAACCAGCUGAGCUACAGGCCCAUCUUAACUUA ...((((.(((((..(((((..----....)))))((((((((.((((.......))))......((((((.....)))))).((((.........))))..))))))))))))).)))) ( -40.80) >Banth.0 30764 112 + 5227293/0-120 AGCUGAGCUAAGGCCCC--AAA----UUG-UAUGGUGGGCCUAAAUGGACUCGAACCAUCGACCUCACGCUUAUCAGGCGUGCGCUCUAACCAGCUGAGCUAUAGGCCCAU-ACAAAUUU .(((.......)))...--...----(((-(...(((((((((...((..((((....)))))).((((((.....)))))).((((.........))))..)))))))))-)))).... ( -34.60) >Bcere.0 30743 112 + 5224283/0-120 AGCUGAGCUAAGGCCCC--AAG----UUG-UAUGGUGGGCCUAAAUGGACUCGAACCAUCGACCUCACGCUUAUCAGGCGUGCGCUCUAACCAGCUGAGCUAUAGGCCCAU-ACAAAUUG .(((.......)))..(--((.----(((-(...(((((((((...((..((((....)))))).((((((.....)))))).((((.........))))..)))))))))-)))).))) ( -35.60) >Bhalo.0 119355 116 + 4202352/0-120 AGCUGAGCUAAGGCCCCGAAAG----CUGUUAUGGUGGGCCUGAGUGGACUUGAACCACCGACCUCACGCUUAUCAGGCGUGCGCUCUAACCAGCUGAGCUACAGGCCCAUAACAAAUUG ......((((..((..(....)----..))..))))(((((((.((((.......))))......((((((.....)))))).((((.........))))..)))))))........... ( -38.30) >consensus AGCUGAGCUAAGGCCCCGUAAA____UUG_UAUGGUGGGCCUGAGUGGACUCGAACCACCGACCUCACGCUUAUCAGGCGUGCGCUCUAACCAGCUGAGCUACAGGCCCAUCACAAAUUA .(((.......)))....................(((((((((.((((.......))))......((((((.....)))))).((((.........))))..)))))))))......... (-34.37 = -33.65 + -0.72)
Generated by rnazCluster.pl (part of RNAz $RNAz::rnazVersion) on Thu Apr 6 17:27:36 2006