
| Sequence ID | Blich.0 |
|---|---|
| Location | 1,488,845 – 1,488,954 |
| Length | 109 |
| Max. P | 0.999990 |

| Location | 1,488,845 – 1,488,954 |
|---|---|
| Length | 109 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 67.94 |
| Mean single sequence MFE | -37.12 |
| Consensus MFE | -25.96 |
| Energy contribution | -25.71 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.11 |
| Mean z-score | -3.68 |
| Structure conservation index | 0.70 |
| SVM decision value | 5.58 |
| SVM RNA-class probability | 0.999990 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>Blich.0 1488845 109 + 4222334/40-160 GGCAACCGACUUUUU-------AAGCACGGUGCUAAUUCUUGCAGCUGACG-CUGAGAGAUAAGGAUUCGAACCUGAAAAGCGAAACCUCUUUUAGCUAAA--GCAGCA-AAAGAGGUUU (....)((.((((((-------(.(..(((..((...(((..((((....)-)))..)))...))..)))..).)))))))))(((((((((((.(((...--..))))-)))))))))) ( -33.30) >Bsubt.0 1426183 110 + 4214630/40-160 GGCAACCGACUUAUA-------AAGCACGGUGCUAAUUCUUGCAGCUAGCGGCUGAGAGAUAAG-AUUCGGAC--GAGAAACGAAACCUCUUUAGACGCGAUUGCAGUUUGAAGAGGUUU .....((((((((..-------.(((.....)))...(((..((((.....))))..)))))))-..))))..--........((((((((((((((((....)).)))))))))))))) ( -36.20) >Bcere.0 3836046 102 + 5224283/40-160 GGCAACCGAUCUACA--AUUGUAGACACGGUGCUAAUUCUCGCAGCAUUACGCUGACAGAUAAGGAGCUGGUU----GUAAAAAAACCUCUCCU----------UAGCU--GAGAGGUUU ((((.(((.(((((.--...)))))..)))))))...((((.((((.....)))).(((.(((((((..((((----.......)))).)))))----------)).))--).))))... ( -38.80) >Banth.0 3892930 104 + 5227293/40-160 GGCAACCGAUCUACAUAAUUGUAGACACGGUGCUAAUUCUCGCAGCAUUACGCUGACAGAUAAGGAGCUGGUU----GUAAAAAAACCUCUCCU----------UAGCU--GAGAGGUUU ((((.(((.((((((....))))))..)))))))...((((.((((.....)))).(((.(((((((..((((----.......)))).)))))----------)).))--).))))... ( -40.20) >consensus GGCAACCGACCUACA_______AAACACGGUGCUAAUUCUCGCAGCAUUACGCUGACAGAUAAGGAGCCGGAC____GAAAAAAAACCUCUCCU__________CAGCU__AAGAGGUUU ((((.(((.(((((......)))))..)))))))...(((..((((.....))))..))).......................(((((((((...................))))))))) (-25.96 = -25.71 + -0.25)

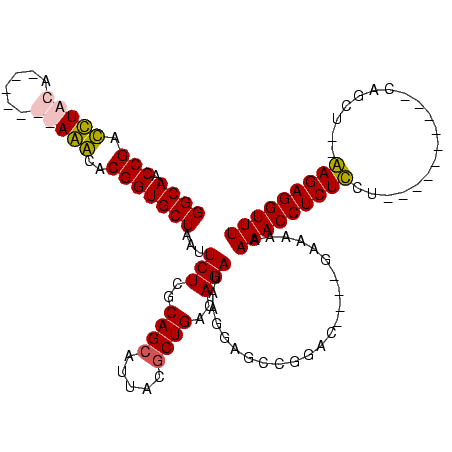
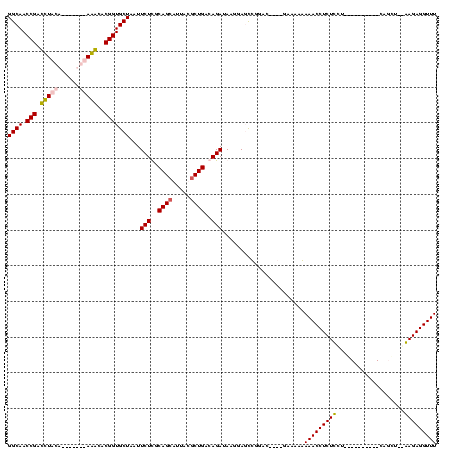
| Location | 1,488,845 – 1,488,954 |
|---|---|
| Length | 109 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 67.94 |
| Mean single sequence MFE | -31.88 |
| Consensus MFE | -24.93 |
| Energy contribution | -24.68 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.73 |
| Structure conservation index | 0.78 |
| SVM decision value | 4.53 |
| SVM RNA-class probability | 0.999916 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>Blich.0 1488845 109 + 4222334/40-160 AAACCUCUUU-UGCUGC--UUUAGCUAAAAGAGGUUUCGCUUUUCAGGUUCGAAUCCUUAUCUCUCAG-CGUCAGCUGCAAGAAUUAGCACCGUGCUU-------AAAAAGUCGGUUGCC ((((((((((-(((((.--..)))).)))))))))))(((((((.((((.((....((..(((..(((-(....))))..)))...))...)).))))-------.))))).))...... ( -31.40) >Bsubt.0 1426183 110 + 4214630/40-160 AAACCUCUUCAAACUGCAAUCGCGUCUAAAGAGGUUUCGUUUCUC--GUCCGAAU-CUUAUCUCUCAGCCGCUAGCUGCAAGAAUUAGCACCGUGCUU-------UAUAAGUCGGUUGCC (((((((((.(.((.((....)))).).)))))))))........--........-....(((..((((.....))))..)))....((((((.(((.-------....)))))).))). ( -25.30) >Bcere.0 3836046 102 + 5224283/40-160 AAACCUCUC--AGCUA----------AGGAGAGGUUUUUUUAC----AACCAGCUCCUUAUCUGUCAGCGUAAUGCUGCGAGAAUUAGCACCGUGUCUACAAU--UGUAGAUCGGUUGCC .....((((--((.((----------(((((.((((.......----))))..))))))).))(.((((.....)))))))))....((((((.((((((...--.))))))))).))). ( -35.10) >Banth.0 3892930 104 + 5227293/40-160 AAACCUCUC--AGCUA----------AGGAGAGGUUUUUUUAC----AACCAGCUCCUUAUCUGUCAGCGUAAUGCUGCGAGAAUUAGCACCGUGUCUACAAUUAUGUAGAUCGGUUGCC .....((((--((.((----------(((((.((((.......----))))..))))))).))(.((((.....)))))))))....((((((.(((((((....)))))))))).))). ( -35.70) >consensus AAACCUCUC__AGCUA__________AAAAGAGGUUUCGUUAC____AACCAAAUCCUUAUCUCUCAGCCGAAAGCUGCAAGAAUUAGCACCGUGCCU_______UAUAAAUCGGUUGCC (((((((((...................))))))))).......................(((..((((.....))))..)))....((((((.((((((......))))))))).))). (-24.93 = -24.68 + -0.25)

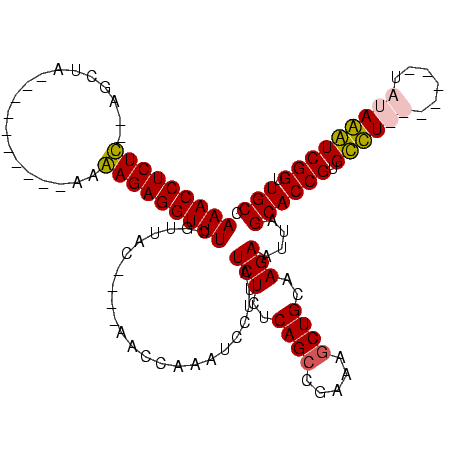
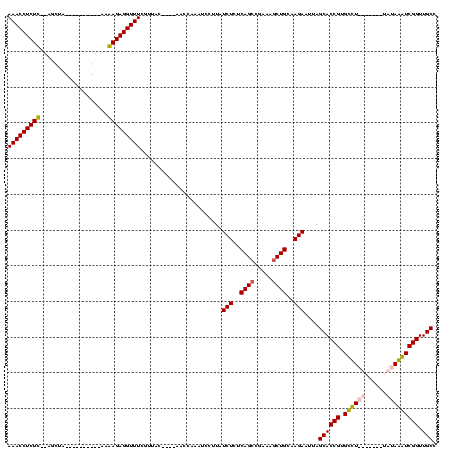
| Location | 1,488,845 – 1,488,954 |
|---|---|
| Length | 109 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 67.50 |
| Mean single sequence MFE | -34.18 |
| Consensus MFE | -24.68 |
| Energy contribution | -25.06 |
| Covariance contribution | 0.38 |
| Combinations/Pair | 1.25 |
| Mean z-score | -3.34 |
| Structure conservation index | 0.72 |
| SVM decision value | 5.34 |
| SVM RNA-class probability | 0.999984 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
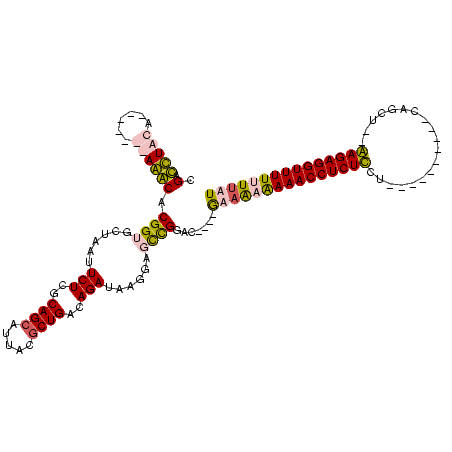
>Blich.0 1488845 109 + 4222334/46-166 CGACUUUUU-------AAGCACGGUGCUAAUUCUUGCAGCUGACG-CUGAGAGAUAAGGAUUCGAACCUGAAAAGCGAAACCUCUUUUAGCUAAA--GCAGCA-AAAGAGGUUUUUUUAU ((.((((((-------(.(..(((..((...(((..((((....)-)))..)))...))..)))..).)))))))))(((((((((((.(((...--..))))-))))))))))...... ( -33.00) >Bsubt.0 1426183 110 + 4214630/46-166 CGACUUAUA-------AAGCACGGUGCUAAUUCUUGCAGCUAGCGGCUGAGAGAUAAG-AUUCGGAC--GAGAAACGAAACCUCUUUAGACGCGAUUGCAGUUUGAAGAGGUUUUUUGAU (((((((..-------.(((.....)))...(((..((((.....))))..)))))))-..)))...--.......(((((((((((((((((....)).)))))))))))))))..... ( -33.90) >Bcere.0 3836046 102 + 5224283/46-166 CGAUCUACA--AUUGUAGACACGGUGCUAAUUCUCGCAGCAUUACGCUGACAGAUAAGGAGCUGGUU----GUAAAAAAACCUCUCCU----------UAGCU--GAGAGGUUUUUUUAU .(.(((((.--...)))))).((((.((...(((..((((.....))))..)))...)).))))...----(((((((((((((((..----------.....--))))))))))))))) ( -34.20) >Banth.0 3892930 104 + 5227293/46-166 CGAUCUACAUAAUUGUAGACACGGUGCUAAUUCUCGCAGCAUUACGCUGACAGAUAAGGAGCUGGUU----GUAAAAAAACCUCUCCU----------UAGCU--GAGAGGUUUUUUUAU .(.((((((....))))))).((((.((...(((..((((.....))))..)))...)).))))...----(((((((((((((((..----------.....--))))))))))))))) ( -35.60) >consensus CGACCUACA_______AAACACGGUGCUAAUUCUCGCAGCAUUACGCUGACAGAUAAGGAGCCGGAC____GAAAAAAAACCUCUCCU__________CAGCU__AAGAGGUUUUUUUAU .(.(((((......)))))).((((......(((..((((.....))))..)))......)))).......(((((((((((((((...................))))))))))))))) (-24.68 = -25.06 + 0.38)


| Location | 1,488,845 – 1,488,954 |
|---|---|
| Length | 109 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 67.50 |
| Mean single sequence MFE | -28.88 |
| Consensus MFE | -17.78 |
| Energy contribution | -19.54 |
| Covariance contribution | 1.75 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.62 |
| SVM decision value | 4.31 |
| SVM RNA-class probability | 0.999867 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
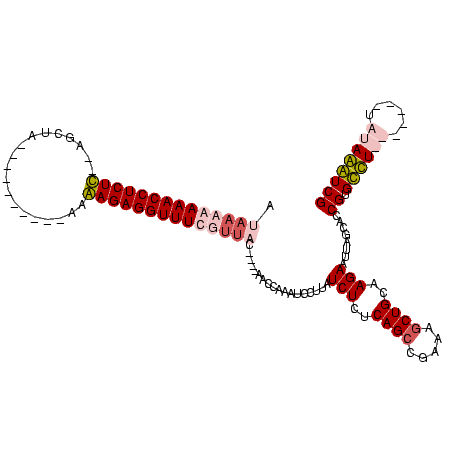
>Blich.0 1488845 109 + 4222334/46-166 AUAAAAAAACCUCUUU-UGCUGC--UUUAGCUAAAAGAGGUUUCGCUUUUCAGGUUCGAAUCCUUAUCUCUCAG-CGUCAGCUGCAAGAAUUAGCACCGUGCUU-------AAAAAGUCG ......((((((((((-(((((.--..)))).))))))))))).((((((.((((.((....((..(((..(((-(....))))..)))...))...)).))))-------.)))))).. ( -30.70) >Bsubt.0 1426183 110 + 4214630/46-166 AUCAAAAAACCUCUUCAAACUGCAAUCGCGUCUAAAGAGGUUUCGUUUCUC--GUCCGAAU-CUUAUCUCUCAGCCGCUAGCUGCAAGAAUUAGCACCGUGCUU-------UAUAAGUCG ......(((((((((.(.((.((....)))).).)))))))))........--...(((..-(((((((..((((.....))))..)))...(((.....))).-------..))))))) ( -21.40) >Bcere.0 3836046 102 + 5224283/46-166 AUAAAAAAACCUCUC--AGCUA----------AGGAGAGGUUUUUUUAC----AACCAGCUCCUUAUCUGUCAGCGUAAUGCUGCGAGAAUUAGCACCGUGUCUACAAU--UGUAGAUCG .((((((((((((((--.....----------..)))))))))))))).----.....(((.....(((..((((.....))))..)))...)))..((.((((((...--.)))))))) ( -31.40) >Banth.0 3892930 104 + 5227293/46-166 AUAAAAAAACCUCUC--AGCUA----------AGGAGAGGUUUUUUUAC----AACCAGCUCCUUAUCUGUCAGCGUAAUGCUGCGAGAAUUAGCACCGUGUCUACAAUUAUGUAGAUCG .((((((((((((((--.....----------..)))))))))))))).----.....(((.....(((..((((.....))))..)))...)))..((.(((((((....))))))))) ( -32.00) >consensus AUAAAAAAACCUCUC__AGCUA__________AAAAGAGGUUUCGUUAC____AACCAAAUCCUUAUCUCUCAGCCGAAAGCUGCAAGAAUUAGCACCGUGCCU_______UAUAAAUCG .((((((((((((((...................))))))))))))))..................(((..((((.....))))..)))........((.((((((......)))))))) (-17.78 = -19.54 + 1.75)

Generated by rnazCluster.pl (part of RNAz $RNAz::rnazVersion) on Thu Apr 6 17:25:08 2006