
| Sequence ID | sy_au.0 |
|---|---|
| Location | 1,975,842 – 1,975,962 |
| Length | 120 |
| Max. P | 0.980418 |

| Location | 1,975,842 – 1,975,956 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.05 |
| Mean single sequence MFE | -38.08 |
| Consensus MFE | -33.33 |
| Energy contribution | -31.88 |
| Covariance contribution | -1.44 |
| Combinations/Pair | 1.15 |
| Mean z-score | -4.06 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.84 |
| SVM RNA-class probability | 0.979655 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
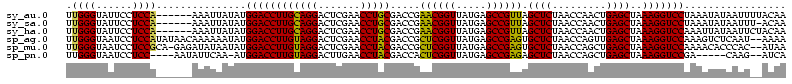
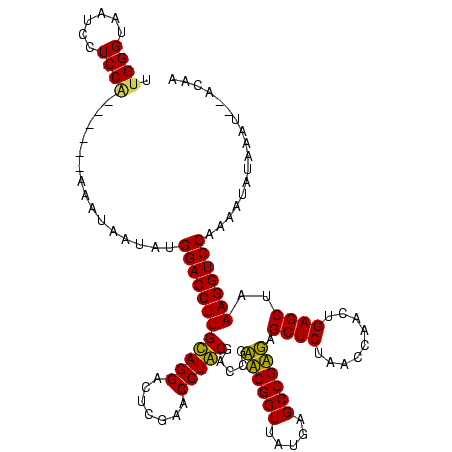
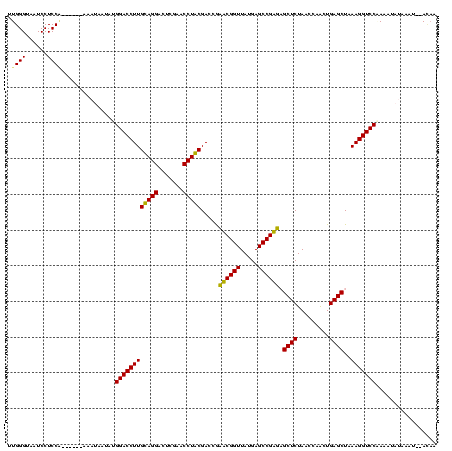
>sy_au.0 1975842 114 + 2809422/80-200 UUGGGUAUUCCUCCA------AAAUUAUAUGGACCUUGCAGGACUCGAACCUGCGACCGAACGGUUAUGAGCCGUUAGCUCUAACCAACUGAGCUAAAGGUCCUAAAUAUAAUUUUACAA ..(((......)))(------(((((((((((((((((((((.......))))).....((((((.....))))))(((((.........))))).)))))))...)))))))))).... ( -38.10) >sy_sa.0 1550718 113 - 2516575/80-200 UUGGGUAUUCCUCCA------AAAUUAUAUGGACCUUGCAGGACUCGAACCUGCGACCGAACGGUUAUGAGCCGUUAGCUCUAACCAACUGAGCUAAAGGUCCUAAAUAUAAUUU-ACAA .((((......))))------(((((((((((((((((((((.......))))).....((((((.....))))))(((((.........))))).)))))))...)))))))))-.... ( -38.10) >sy_ha.0 1575664 114 - 2685015/80-200 UUGGGUAUUCCUCCA------AAAUUAUAUGGACCUUGCAGGACUCGAACCUGCGACCGAACGGUUAUGAGCCGUUAGCUCUAACCAACUGAGCUAAAGGUCCAAAUUAUAAUUCUACAA (((((......))))------)((((((((((((((((((((.......))))).....((((((.....))))))(((((.........))))).))))))))...)))))))...... ( -37.80) >sp_ag.0 2132052 118 - 2160267/80-200 UUGGGUAAUCCUCCAUAUAACAAAAAAUAUGGACCUUGUAGGACUCGAACCUACGACCGCUCGGUUAUGAGCCGAGUGCUCUAACCAGUUGAGCUAAAGGUCCAAAGUCUCAAU--AAAA (((((......(((((((........))))))).((((((((.......)))))(((((((((((.....)))))))((((.........))))....))))..))).))))).--.... ( -36.40) >sp_mu.0 2007783 117 - 2030921/80-200 UUGGGUAAUCCUCCGCA-GAGAUAUAAUAUGGACCUUGUAGGACUCGAACCUACGACCGCUCGGUUAUGAGCCGAGUGCUCUAACCAGCUGAGCUAAAGGUCCAAAACACCCAC--AUAA .(((((....(((....-)))........(((((((((((((.......)))))....((((((((..((((.....)))).....))))))))..))))))))....))))).--.... ( -42.30) >sp_pn.0 1691231 108 + 2038615/80-200 UUGGGUAAUCCUCC----AAUAUUCAA-AUGGACCUUGUAGGACUUGAACCUACGACCACUCGGUUAUGAGCCGAGAGCUCUAACCAGCUGAGCUAAAGGUCCGA-----CAAG--AUCA (((((......)))----)).......-.(((((((((((((.......))))).....((((((.....))))))(((((.........))))).)))))))).-----....--.... ( -35.80) >consensus UUGGGUAAUCCUCCA______AAAUAAUAUGGACCUUGCAGGACUCGAACCUACGACCGAACGGUUAUGAGCCGAGAGCUCUAACCAACUGAGCUAAAGGUCCAAAAUAUAAAU__ACAA .((((......))))...............((((((((((((.......))))).....((((((.....)))))).((((.........))))..)))))))................. (-33.33 = -31.88 + -1.44)
| Location | 1,975,842 – 1,975,956 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.05 |
| Mean single sequence MFE | -38.93 |
| Consensus MFE | -32.78 |
| Energy contribution | -32.28 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.04 |
| Mean z-score | -3.11 |
| Structure conservation index | 0.84 |
| SVM decision value | 1.58 |
| SVM RNA-class probability | 0.965353 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
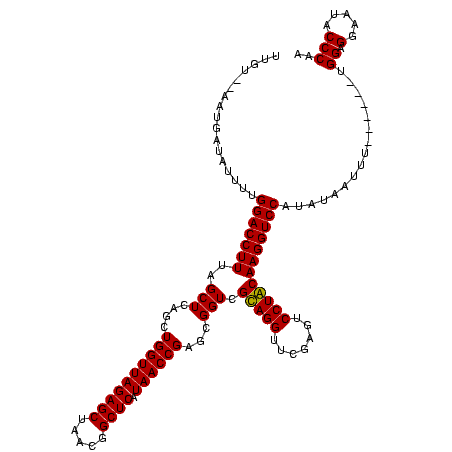
>sy_au.0 1975842 114 + 2809422/80-200 UUGUAAAAUUAUAUUUAGGACCUUUAGCUCAGUUGGUUAGAGCUAACGGCUCAUAACCGUUCGGUCGCAGGUUCGAGUCCUGCAAGGUCCAUAUAAUUU------UGGAGGAAUACCCAA .(.(((((((((((...(((((((..(((....((((((((((.....)))).))))))...))).(((((.......)))))))))))))))))))))------)).)((.....)).. ( -39.40) >sy_sa.0 1550718 113 - 2516575/80-200 UUGU-AAAUUAUAUUUAGGACCUUUAGCUCAGUUGGUUAGAGCUAACGGCUCAUAACCGUUCGGUCGCAGGUUCGAGUCCUGCAAGGUCCAUAUAAUUU------UGGAGGAAUACCCAA ....-(((((((((...(((((((..(((....((((((((((.....)))).))))))...))).(((((.......)))))))))))))))))))))------(((.(.....)))). ( -37.50) >sy_ha.0 1575664 114 - 2685015/80-200 UUGUAGAAUUAUAAUUUGGACCUUUAGCUCAGUUGGUUAGAGCUAACGGCUCAUAACCGUUCGGUCGCAGGUUCGAGUCCUGCAAGGUCCAUAUAAUUU------UGGAGGAAUACCCAA .(.((((((((((...((((((((..(((....((((((((((.....)))).))))))...))).(((((.......)))))))))))))))))))))------)).)((.....)).. ( -40.00) >sp_ag.0 2132052 118 - 2160267/80-200 UUUU--AUUGAGACUUUGGACCUUUAGCUCAACUGGUUAGAGCACUCGGCUCAUAACCGAGCGGUCGUAGGUUCGAGUCCUACAAGGUCCAUAUUUUUUGUUAUAUGGAGGAUUACCCAA ....--..........((((((((..((((....(((((((((.....)))).)))))))))....(((((.......)))))))))))))..............(((.(.....)))). ( -36.60) >sp_mu.0 2007783 117 - 2030921/80-200 UUAU--GUGGGUGUUUUGGACCUUUAGCUCAGCUGGUUAGAGCACUCGGCUCAUAACCGAGCGGUCGUAGGUUCGAGUCCUACAAGGUCCAUAUUAUAUCUC-UGCGGAGGAUUACCCAA ....--.((((((...((((((((..((((....(((((((((.....)))).)))))))))....(((((.......))))))))))))).......((((-....))))..)))))). ( -45.20) >sp_pn.0 1691231 108 + 2038615/80-200 UGAU--CUUG-----UCGGACCUUUAGCUCAGCUGGUUAGAGCUCUCGGCUCAUAACCGAGUGGUCGUAGGUUCAAGUCCUACAAGGUCCAU-UUGAAUAUU----GGAGGAUUACCCAA ...(--(..(-----(.(((((((..(..((..((((((((((.....)))).))))))..))..)(((((.......))))))))))))))-..))...((----((.(.....))))) ( -34.90) >consensus UUGU__AAUGAUAUUUUGGACCUUUAGCUCAGCUGGUUAGAGCUAACGGCUCAUAACCGAGCGGUCGCAGGUUCGAGUCCUACAAGGUCCAUAUAAUUU______UGGAGGAAUACCCAA .................(((((((..(((....((((((((((.....)))).))))))...))).(((((.......))))))))))))................((.(.....))).. (-32.78 = -32.28 + -0.50) # Strand winner: reverse (0.56)

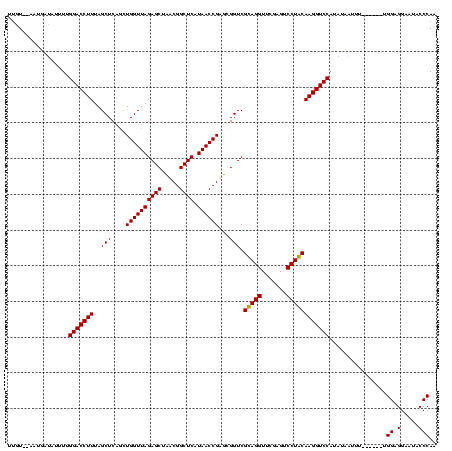
| Location | 1,975,842 – 1,975,962 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.35 |
| Mean single sequence MFE | -34.68 |
| Consensus MFE | -28.77 |
| Energy contribution | -27.85 |
| Covariance contribution | -0.92 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.39 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.606227 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>sy_au.0 1975842 120 + 2809422/120-240 GGACUCGAACCUGCGACCGAACGGUUAUGAGCCGUUAGCUCUAACCAACUGAGCUAAAGGUCCUAAAUAUAAUUUUACAACUAAUAAAUAGUGGCGGUGGAGGGGAUCGAACCCCCGACC ((.(((..(((.((((((.((((((.....))))))(((((.........)))))...)))).................((((.....)))).))))).)))(((......))))).... ( -34.20) >sy_sa.0 1550718 119 - 2516575/120-240 GGACUCGAACCUGCGACCGAACGGUUAUGAGCCGUUAGCUCUAACCAACUGAGCUAAAGGUCCUAAAUAUAAUUU-ACAACUAAUAAUUAGUGGCGGUGGAGGGGAUCGAACCCCCGACC ((.(((..(((.((((((.((((((.....))))))(((((.........)))))...)))).............-...((((.....)))).))))).)))(((......))))).... ( -34.20) >sy_ha.0 1575664 120 - 2685015/120-240 GGACUCGAACCUGCGACCGAACGGUUAUGAGCCGUUAGCUCUAACCAACUGAGCUAAAGGUCCAAAUUAUAAUUCUACAACUAAUAAGUAGUGGCGGUGGAGGGGAUCGAACCCCCGACC ((..((((.(((.(.((((...(((((((((((.(((((((.........))))))).))).....))))))))((((.(((....))).)))))))).)..))).))))..))...... ( -36.70) >sp_ag.0 2132052 111 - 2160267/120-240 GGACUCGAACCUACGACCGCUCGGUUAUGAGCCGAGUGCUCUAACCAGUUGAGCUAAAGGUCCAAAGUCUCAAU--AAAAUAAAU-------AGCGGCGGAGGGGAUCGAACCCCCGACC ((..((((.(((.((.(((((.(((((.((((.....))))))))).((((((((..........)).))))))--.........-------)))))))...))).))))..))...... ( -37.30) >sp_mu.0 2007783 115 - 2030921/120-240 GGACUCGAACCUACGACCGCUCGGUUAUGAGCCGAGUGCUCUAACCAGCUGAGCUAAAGGUCCAAAACACCCAC--AUAAUUAAU---UUAUAGCGGCGAAGGGGAUCGAACCCCCGACC ((..((((.(((..(((((((((((.....)))))))((((.........))))....)))).......(((.(--((((.....---)))).).)).)...))).))))..))...... ( -33.50) >sp_pn.0 1691231 105 + 2038615/120-240 GGACUUGAACCUACGACCACUCGGUUAUGAGCCGAGAGCUCUAACCAGCUGAGCUAAAGGUCCGA-----CAAG--AUCAUUA--------UAGCGGCGAAGGGGAUCGAACCCCCGACC .((((((.......((((.((((((.....))))))(((((.........)))))...))))...-----))))--.))....--------....((((..((((......)))))).)) ( -32.20) >consensus GGACUCGAACCUACGACCGAACGGUUAUGAGCCGAGAGCUCUAACCAACUGAGCUAAAGGUCCAAAAUAUAAAU__ACAACUAAU___UAGUAGCGGCGGAGGGGAUCGAACCCCCGACC (((((..............((((((.....)))))).((((.........))))....)))))................................(((((.(((.......)))))).)) (-28.77 = -27.85 + -0.92)

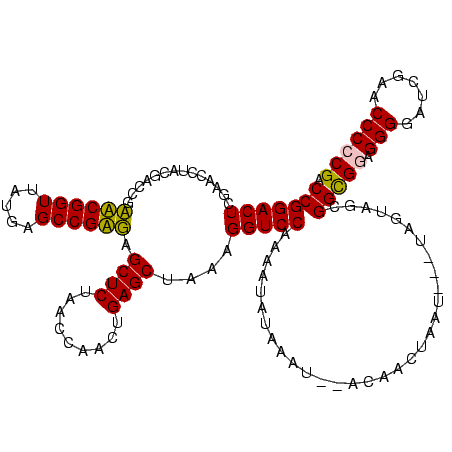
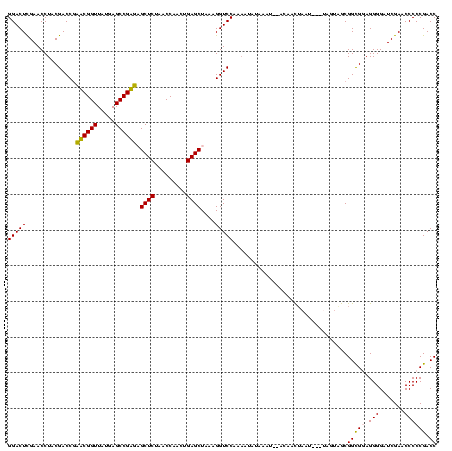
| Location | 1,975,842 – 1,975,962 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.35 |
| Mean single sequence MFE | -37.85 |
| Consensus MFE | -33.28 |
| Energy contribution | -33.03 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.27 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.03 |
| SVM RNA-class probability | 0.547269 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>sy_au.0 1975842 120 + 2809422/120-240 GGUCGGGGGUUCGAUCCCCUCCACCGCCACUAUUUAUUAGUUGUAAAAUUAUAUUUAGGACCUUUAGCUCAGUUGGUUAGAGCUAACGGCUCAUAACCGUUCGGUCGCAGGUUCGAGUCC (((.(((((......)))).).)))((.((((.....)))).)).............((((((.(..((....((((((((((.....)))).))))))...))..).))))))...... ( -36.40) >sy_sa.0 1550718 119 - 2516575/120-240 GGUCGGGGGUUCGAUCCCCUCCACCGCCACUAAUUAUUAGUUGU-AAAUUAUAUUUAGGACCUUUAGCUCAGUUGGUUAGAGCUAACGGCUCAUAACCGUUCGGUCGCAGGUUCGAGUCC (((.(((((......)))).).)))((.((((.....)))).))-............((((((.(..((....((((((((((.....)))).))))))...))..).))))))...... ( -35.60) >sy_ha.0 1575664 120 - 2685015/120-240 GGUCGGGGGUUCGAUCCCCUCCACCGCCACUACUUAUUAGUUGUAGAAUUAUAAUUUGGACCUUUAGCUCAGUUGGUUAGAGCUAACGGCUCAUAACCGUUCGGUCGCAGGUUCGAGUCC (((.(((((......)))).).)))....((((.........)))).......((((((((((.(..((....((((((((((.....)))).))))))...))..).)))))))))).. ( -38.90) >sp_ag.0 2132052 111 - 2160267/120-240 GGUCGGGGGUUCGAUCCCCUCCGCCGCU-------AUUUAUUUU--AUUGAGACUUUGGACCUUUAGCUCAACUGGUUAGAGCACUCGGCUCAUAACCGAGCGGUCGUAGGUUCGAGUCC ((.((((((........))))))))...-------.........--.....(((((.((((((...((((....(((((((((.....)))).)))))))))......))))))))))). ( -40.80) >sp_mu.0 2007783 115 - 2030921/120-240 GGUCGGGGGUUCGAUCCCCUUCGCCGCUAUAA---AUUAAUUAU--GUGGGUGUUUUGGACCUUUAGCUCAGCUGGUUAGAGCACUCGGCUCAUAACCGAGCGGUCGUAGGUUCGAGUCC ((.(((((((...))))))....((((.((((---.....))))--)))).....((((((((...(((..((((((((((((.....)))).))))).))))))...))))))))).)) ( -39.70) >sp_pn.0 1691231 105 + 2038615/120-240 GGUCGGGGGUUCGAUCCCCUUCGCCGCUA--------UAAUGAU--CUUG-----UCGGACCUUUAGCUCAGCUGGUUAGAGCUCUCGGCUCAUAACCGAGUGGUCGUAGGUUCAAGUCC (((.(((((......)))))..)))....--------.......--....-----..((((((...(((..((((((((((((.....)))).))))).))))))...))))))...... ( -35.70) >consensus GGUCGGGGGUUCGAUCCCCUCCACCGCCACUA___AUUAAUUGU__AAUGAUAUUUUGGACCUUUAGCUCAGCUGGUUAGAGCUAACGGCUCAUAACCGAGCGGUCGCAGGUUCGAGUCC (((.(((((......)))))..)))................................((((((...(((....((((((((((.....)))).))))))...)))...))))))...... (-33.28 = -33.03 + -0.25) # Strand winner: reverse (0.99)

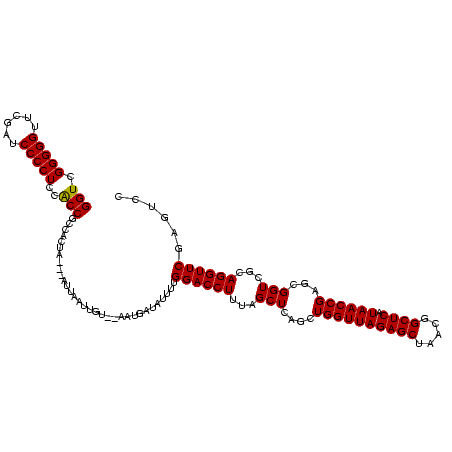
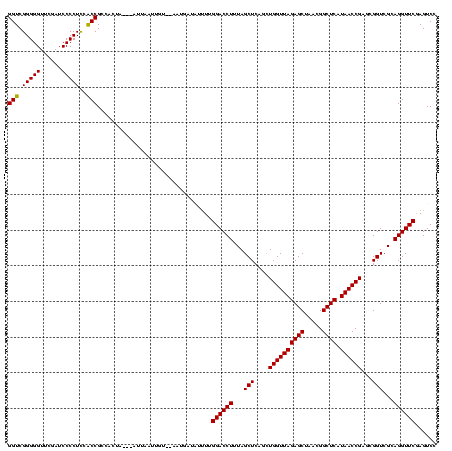
| Location | 1,975,842 – 1,975,962 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.88 |
| Mean single sequence MFE | -35.40 |
| Consensus MFE | -26.27 |
| Energy contribution | -25.68 |
| Covariance contribution | -0.58 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.74 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.523382 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>sy_au.0 1975842 120 + 2809422/160-280 CUAACCAACUGAGCUAAAGGUCCUAAAUAUAAUUUUACAACUAAUAAAUAGUGGCGGUGGAGGGGAUCGAACCCCCGACCUCACGGGUAUGAACCGUACGCUCUAGCCAGCUGAGCUACA ........((.((((...(((..((((......))))..)))..........((((((.(.((((......))))).)))..((((.......))))........))))))).))..... ( -32.20) >sy_sa.0 1550718 119 - 2516575/160-280 CUAACCAACUGAGCUAAAGGUCCUAAAUAUAAUUU-ACAACUAAUAAUUAGUGGCGGUGGAGGGGAUCGAACCCCCGACCUCACGGGUAUGAACCGUACGCUCUAGCCAGCUGAGCUACA .....((.(((.((((.(((((((((.(((.....-.......))).)))).)))(((.(.((((......))))).)))..((((.......))))...)).))))))).))....... ( -31.80) >sy_ha.0 1575664 120 - 2685015/160-280 CUAACCAACUGAGCUAAAGGUCCAAAUUAUAAUUCUACAACUAAUAAGUAGUGGCGGUGGAGGGGAUCGAACCCCCGACCUCACGGGUAUGAACCGUACGCUCUAGCCAGCUGAGCUACA .....((.(((.((((.(((((...........(((((.((((.....))))....)))))((((.......))))))))).((((.......))))......))))))).))....... ( -34.10) >sp_ag.0 2132052 111 - 2160267/160-280 CUAACCAGUUGAGCUAAAGGUCCAAAGUCUCAAU--AAAAUAAAU-------AGCGGCGGAGGGGAUCGAACCCCCGACCUCCCGGGUAUGAACCGGACGCUCUAGCCAGCUGAGCUACA .....((((((.((((.((((((...........--.........-------.....(((.((((.(((......))))))))))(((....))))))).)).))))))))))....... ( -37.30) >sp_mu.0 2007783 115 - 2030921/160-280 CUAACCAGCUGAGCUAAAGGUCCAAAACACCCAC--AUAAUUAAU---UUAUAGCGGCGAAGGGGAUCGAACCCCCGACCUCCCGGGUAUGAACCGGACGCUCUAGCCAGCUGAGCUACA .....((((((.((((.((((((...........--.........---......(((.((.((((.......))))....)))))(((....))))))).)).))))))))))....... ( -38.50) >sp_pn.0 1691231 105 + 2038615/160-280 CUAACCAGCUGAGCUAAAGGUCCGA-----CAAG--AUCAUUA--------UAGCGGCGAAGGGGAUCGAACCCCCGACCUCCCGGGUAUGAACCGGACGCUCUAGCCAGCUGAGCUACA .....((((((.((((.((((((..-----....--.......--------...(((.((.((((.......))))....)))))(((....))))))).)).))))))))))....... ( -38.50) >consensus CUAACCAACUGAGCUAAAGGUCCAAAAUAUAAAU__ACAACUAAU___UAGUAGCGGCGGAGGGGAUCGAACCCCCGACCUCACGGGUAUGAACCGGACGCUCUAGCCAGCUGAGCUACA ..........((((...(((((..................(((........))).......((((.......))))))))).((((.......))))..))))(((((....).)))).. (-26.27 = -25.68 + -0.58)

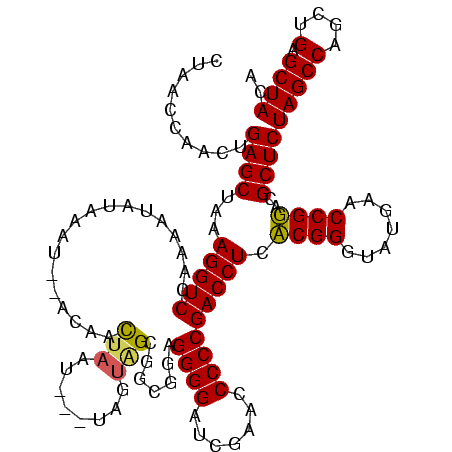
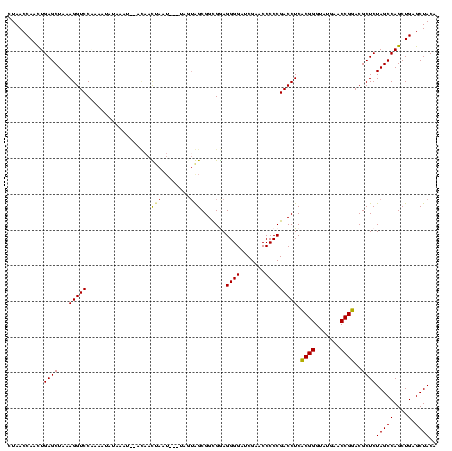
| Location | 1,975,842 – 1,975,962 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.88 |
| Mean single sequence MFE | -43.60 |
| Consensus MFE | -38.92 |
| Energy contribution | -38.08 |
| Covariance contribution | -0.83 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.71 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.86 |
| SVM RNA-class probability | 0.980418 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>sy_au.0 1975842 120 + 2809422/160-280 UGUAGCUCAGCUGGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCACCGCCACUAUUUAUUAGUUGUAAAAUUAUAUUUAGGACCUUUAGCUCAGUUGGUUAG ..((((.(((((((((((((.((.((((......((....))..((((((...))))))...))))...(((..(((.((((....)))))))..))).))))))))).)))))))))). ( -41.00) >sy_sa.0 1550718 119 - 2516575/160-280 UGUAGCUCAGCUGGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCACCGCCACUAAUUAUUAGUUGU-AAAUUAUAUUUAGGACCUUUAGCUCAGUUGGUUAG ..((((.(((((((((((((.....(((....))).(((.(((.(((((......)))).).)))..))).........(((.(-((((...))))).)))))))))).)))))))))). ( -39.60) >sy_ha.0 1575664 120 - 2685015/160-280 UGUAGCUCAGCUGGCUAGAGCGUACGGUUCAUACCCGUGAGGUCGGGGGUUCGAUCCCCUCCACCGCCACUACUUAUUAGUUGUAGAAUUAUAAUUUGGACCUUUAGCUCAGUUGGUUAG ..((((.(((((((((((((.((.((((((((....))))(((.(((((......)))).).)))))).((((.........))))...........).))))))))).)))))))))). ( -40.60) >sp_ag.0 2132052 111 - 2160267/160-280 UGUAGCUCAGCUGGCUAGAGCGUCCGGUUCAUACCCGGGAGGUCGGGGGUUCGAUCCCCUCCGCCGCU-------AUUUAUUUU--AUUGAGACUUUGGACCUUUAGCUCAACUGGUUAG ..((((.(((.(((((((((.(((((((....)))(((..((..((((((...)))))).)).)))..-------.........--...........))))))))))).)).))))))). ( -40.00) >sp_mu.0 2007783 115 - 2030921/160-280 UGUAGCUCAGCUGGCUAGAGCGUCCGGUUCAUACCCGGGAGGUCGGGGGUUCGAUCCCCUUCGCCGCUAUAA---AUUAAUUAU--GUGGGUGUUUUGGACCUUUAGCUCAGCUGGUUAG ..((((.(((((((((((((.((((((...(((((((((.(((.(((((......)))))..))).))((((---.....))))--.))))))).))))))))))))).)))))))))). ( -52.60) >sp_pn.0 1691231 105 + 2038615/160-280 UGUAGCUCAGCUGGCUAGAGCGUCCGGUUCAUACCCGGGAGGUCGGGGGUUCGAUCCCCUUCGCCGCUA--------UAAUGAU--CUUG-----UCGGACCUUUAGCUCAGCUGGUUAG ..((((.(((((((((((((.((((((.((((.....((.(((.(((((......)))))..))).)).--------..)))).--....-----))))))))))))).)))))))))). ( -47.80) >consensus UGUAGCUCAGCUGGCUAGAGCGUACGGUUCAUACCCGGGAGGUCGGGGGUUCGAUCCCCUCCACCGCCACUA___AUUAAUUGU__AAUGAUAUUUUGGACCUUUAGCUCAGCUGGUUAG ..((((.(((((((((((((.((((((.......))))..(((.(((((......)))))..)))..................................))))))))).)))))))))). (-38.92 = -38.08 + -0.83) # Strand winner: reverse (1.00)

Generated by rnazCluster.pl (part of RNAz $RNAz::rnazVersion) on Thu Apr 6 18:54:19 2006