
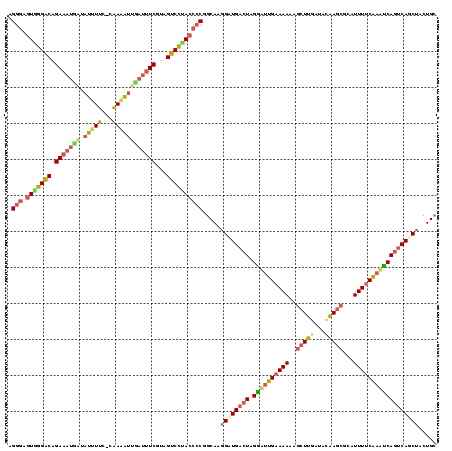
| Sequence ID | sy_au.0 |
|---|---|
| Location | 2,803,278 – 2,803,396 |
| Length | 118 |
| Max. P | 0.999983 |

| Location | 2,803,278 – 2,803,396 |
|---|---|
| Length | 118 |
| Sequences | 4 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 74.65 |
| Mean single sequence MFE | -41.04 |
| Consensus MFE | -33.00 |
| Energy contribution | -34.88 |
| Covariance contribution | 1.88 |
| Combinations/Pair | 1.38 |
| Mean z-score | -4.86 |
| Structure conservation index | 0.80 |
| SVM decision value | 4.69 |
| SVM RNA-class probability | 0.999939 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>sy_au.0 2803278 118 + 2809422 AGGGAGUGGGACAGAAAUGAUAUUUUCGCAAAAUUUAUUUCGUCGUCCCACCCCAGCAAGGUUGACUAGAAUUGAAAAAAGCUUGUUACAAGCGCAUUUUCGU-UCAGUCAACUACUGC .(((.(((((((.(((((((.((((.....)))))))))))...)))))))))).(((.((((((((.((((.(((((..(((((...)))))...)))))))-))))))))))..))) ( -46.80) >sy_ha.0 482537 119 + 2685015 AGGGAGUAGGACAGAAAUAAUAUUUUCUCAAAAUUUAUUUCGUAGUCCUACCCCGGCAAGGAUGACUAGGUUUGAAAAAAGCUUGAUUCAAGCGCAUUUUCAAAUCAGUCAGCUACUGA .(((.(((((((.(((((((.((((.....)))))))))))...)))))))))).....((.(((((.((((((((((..(((((...)))))...))))))))))))))).))..... ( -43.50) >sy_sa.0 2080833 118 + 2516575 AGGGAGUGGGACAGAAAUCAAAUUUUU-UAAUAGAGAUUUCGUAGUCCAACCCCGGCAAGGAUGACUAGGAUUGAAAAAAGCUUGAUAUAAGCGUAUUUUCAAUUCAGUCAUCUACUGC .(((.((.((((.((((((........-.......))))))...)))).))))).(((.((((((((.((((((((((..(((((...)))))...))))))))))))))))))..))) ( -43.46) >sp_pn.0 767243 116 - 2038615 AGAAAAUGAGGCAGAUUCGGUAACUCGAAGAGUUCGAUUUCAUCGUCUUACCCCUGCAACGAUGACUAGGUUUGAAAAAGA-UUGCUA-GAGCGCAUUU-CAAACCAUGCAGCAACUGC ..........((((....(((((..(((.(((......))).)))..))))).(((((..(....)..(((((((((....-.(((..-....))))))-)))))).)))))...)))) ( -30.40) >consensus AGGGAGUGGGACAGAAAUGAUAUUUUC_CAAAAUUGAUUUCGUAGUCCUACCCCGGCAAGGAUGACUAGGAUUGAAAAAAGCUUGAUACAAGCGCAUUUUCAAAUCAGUCAGCUACUGC .(((.(((((((.(((((((.(((((...))))))))))))...)))))))))).....((((((((.((((((((((..(((((...)))))...))))))))))))))))))..... (-33.00 = -34.88 + 1.88)


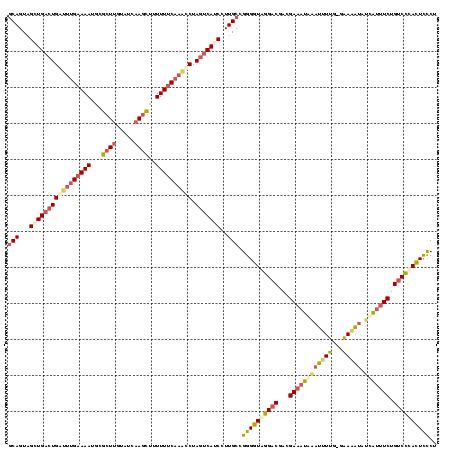
| Location | 2,803,278 – 2,803,396 |
|---|---|
| Length | 118 |
| Sequences | 4 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 74.65 |
| Mean single sequence MFE | -37.77 |
| Consensus MFE | -29.39 |
| Energy contribution | -31.82 |
| Covariance contribution | 2.44 |
| Combinations/Pair | 1.33 |
| Mean z-score | -4.30 |
| Structure conservation index | 0.78 |
| SVM decision value | 5.31 |
| SVM RNA-class probability | 0.999983 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>sy_au.0 2803278 118 + 2809422 GCAGUAGUUGACUGA-ACGAAAAUGCGCUUGUAACAAGCUUUUUUCAAUUCUAGUCAACCUUGCUGGGGUGGGACGACGAAAUAAAUUUUGCGAAAAUAUCAUUUCUGUCCCACUCCCU ((((..(((((((((-(.(((((...(((((...)))))..)))))..))).))))))).)))).(((((((((((.(((((....))))))((((......)))).)))))))))).. ( -41.70) >sy_ha.0 482537 119 + 2685015 UCAGUAGCUGACUGAUUUGAAAAUGCGCUUGAAUCAAGCUUUUUUCAAACCUAGUCAUCCUUGCCGGGGUAGGACUACGAAAUAAAUUUUGAGAAAAUAUUAUUUCUGUCCUACUCCCU ...((((.((((((.((((((((...(((((...)))))..))))))))..))))))...)))).((((((((((...(((((((..(((....)))..))))))).)))))))))).. ( -39.00) >sy_sa.0 2080833 118 + 2516575 GCAGUAGAUGACUGAAUUGAAAAUACGCUUAUAUCAAGCUUUUUUCAAUCCUAGUCAUCCUUGCCGGGGUUGGACUACGAAAUCUCUAUUA-AAAAAUUUGAUUUCUGUCCCACUCCCU ((((..((((((((.((((((((...((((.....))))..))))))))..)))))))).)))).(((((.((((...((((((...(((.-...)))..)))))).)))).))))).. ( -39.70) >sp_pn.0 767243 116 - 2038615 GCAGUUGCUGCAUGGUUUG-AAAUGCGCUC-UAGCAA-UCUUUUUCAAACCUAGUCAUCGUUGCAGGGGUAAGACGAUGAAAUCGAACUCUUCGAGUUACCGAAUCUGCCUCAUUUUCU ((((...(((((.((((((-((((((....-..))).-....)))))))))..........))))).(((((..(((.((........)).)))..)))))....)))).......... ( -30.70) >consensus GCAGUAGCUGACUGAUUUGAAAAUGCGCUUGUAUCAAGCUUUUUUCAAACCUAGUCAUCCUUGCCGGGGUAGGACGACGAAAUAAAUUUUG_GAAAAUAUCAUUUCUGUCCCACUCCCU (((...(((((((((((((((((...((((.....))))..)))))))))).)))))))..))).((((((((((...((((((((((((...))))).))))))).)))))))))).. (-29.39 = -31.82 + 2.44) # Strand winner: forward (0.98)


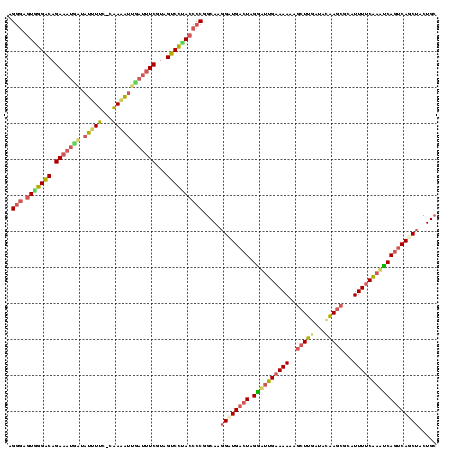
| Location | 2,803,278 – 2,803,396 |
|---|---|
| Length | 118 |
| Sequences | 4 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 74.65 |
| Mean single sequence MFE | -41.04 |
| Consensus MFE | -33.00 |
| Energy contribution | -34.88 |
| Covariance contribution | 1.88 |
| Combinations/Pair | 1.38 |
| Mean z-score | -4.86 |
| Structure conservation index | 0.80 |
| SVM decision value | 4.69 |
| SVM RNA-class probability | 0.999939 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>sy_au.0 2803278 118 + 2809422/0-119 AGGGAGUGGGACAGAAAUGAUAUUUUCGCAAAAUUUAUUUCGUCGUCCCACCCCAGCAAGGUUGACUAGAAUUGAAAAAAGCUUGUUACAAGCGCAUUUUCGU-UCAGUCAACUACUGC .(((.(((((((.(((((((.((((.....)))))))))))...)))))))))).(((.((((((((.((((.(((((..(((((...)))))...)))))))-))))))))))..))) ( -46.80) >sy_ha.0 482537 119 + 2685015/0-119 AGGGAGUAGGACAGAAAUAAUAUUUUCUCAAAAUUUAUUUCGUAGUCCUACCCCGGCAAGGAUGACUAGGUUUGAAAAAAGCUUGAUUCAAGCGCAUUUUCAAAUCAGUCAGCUACUGA .(((.(((((((.(((((((.((((.....)))))))))))...)))))))))).....((.(((((.((((((((((..(((((...)))))...))))))))))))))).))..... ( -43.50) >sy_sa.0 2080833 118 + 2516575/0-119 AGGGAGUGGGACAGAAAUCAAAUUUUU-UAAUAGAGAUUUCGUAGUCCAACCCCGGCAAGGAUGACUAGGAUUGAAAAAAGCUUGAUAUAAGCGUAUUUUCAAUUCAGUCAUCUACUGC .(((.((.((((.((((((........-.......))))))...)))).))))).(((.((((((((.((((((((((..(((((...)))))...))))))))))))))))))..))) ( -43.46) >sp_pn.0 767243 116 - 2038615/0-119 AGAAAAUGAGGCAGAUUCGGUAACUCGAAGAGUUCGAUUUCAUCGUCUUACCCCUGCAACGAUGACUAGGUUUGAAAAAGA-UUGCUA-GAGCGCAUUU-CAAACCAUGCAGCAACUGC ..........((((....(((((..(((.(((......))).)))..))))).(((((..(....)..(((((((((....-.(((..-....))))))-)))))).)))))...)))) ( -30.40) >consensus AGGGAGUGGGACAGAAAUGAUAUUUUC_CAAAAUUGAUUUCGUAGUCCUACCCCGGCAAGGAUGACUAGGAUUGAAAAAAGCUUGAUACAAGCGCAUUUUCAAAUCAGUCAGCUACUGC .(((.(((((((.(((((((.(((((...))))))))))))...)))))))))).....((((((((.((((((((((..(((((...)))))...))))))))))))))))))..... (-33.00 = -34.88 + 1.88)


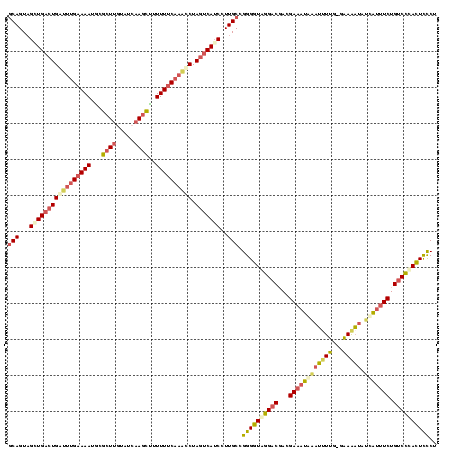
| Location | 2,803,278 – 2,803,396 |
|---|---|
| Length | 118 |
| Sequences | 4 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 74.65 |
| Mean single sequence MFE | -37.77 |
| Consensus MFE | -29.39 |
| Energy contribution | -31.82 |
| Covariance contribution | 2.44 |
| Combinations/Pair | 1.33 |
| Mean z-score | -4.30 |
| Structure conservation index | 0.78 |
| SVM decision value | 5.31 |
| SVM RNA-class probability | 0.999983 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>sy_au.0 2803278 118 + 2809422/0-119 GCAGUAGUUGACUGA-ACGAAAAUGCGCUUGUAACAAGCUUUUUUCAAUUCUAGUCAACCUUGCUGGGGUGGGACGACGAAAUAAAUUUUGCGAAAAUAUCAUUUCUGUCCCACUCCCU ((((..(((((((((-(.(((((...(((((...)))))..)))))..))).))))))).)))).(((((((((((.(((((....))))))((((......)))).)))))))))).. ( -41.70) >sy_ha.0 482537 119 + 2685015/0-119 UCAGUAGCUGACUGAUUUGAAAAUGCGCUUGAAUCAAGCUUUUUUCAAACCUAGUCAUCCUUGCCGGGGUAGGACUACGAAAUAAAUUUUGAGAAAAUAUUAUUUCUGUCCUACUCCCU ...((((.((((((.((((((((...(((((...)))))..))))))))..))))))...)))).((((((((((...(((((((..(((....)))..))))))).)))))))))).. ( -39.00) >sy_sa.0 2080833 118 + 2516575/0-119 GCAGUAGAUGACUGAAUUGAAAAUACGCUUAUAUCAAGCUUUUUUCAAUCCUAGUCAUCCUUGCCGGGGUUGGACUACGAAAUCUCUAUUA-AAAAAUUUGAUUUCUGUCCCACUCCCU ((((..((((((((.((((((((...((((.....))))..))))))))..)))))))).)))).(((((.((((...((((((...(((.-...)))..)))))).)))).))))).. ( -39.70) >sp_pn.0 767243 116 - 2038615/0-119 GCAGUUGCUGCAUGGUUUG-AAAUGCGCUC-UAGCAA-UCUUUUUCAAACCUAGUCAUCGUUGCAGGGGUAAGACGAUGAAAUCGAACUCUUCGAGUUACCGAAUCUGCCUCAUUUUCU ((((...(((((.((((((-((((((....-..))).-....)))))))))..........))))).(((((..(((.((........)).)))..)))))....)))).......... ( -30.70) >consensus GCAGUAGCUGACUGAUUUGAAAAUGCGCUUGUAUCAAGCUUUUUUCAAACCUAGUCAUCCUUGCCGGGGUAGGACGACGAAAUAAAUUUUG_GAAAAUAUCAUUUCUGUCCCACUCCCU (((...(((((((((((((((((...((((.....))))..)))))))))).)))))))..))).((((((((((...((((((((((((...))))).))))))).)))))))))).. (-29.39 = -31.82 + 2.44) # Strand winner: forward (0.98)


Generated by rnazCluster.pl (part of RNAz $RNAz::rnazVersion) on Thu Apr 6 18:59:27 2006