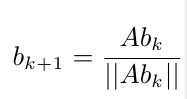Ergebnisse des Praktikums in der Graph Theorie
Reading and filtering the data

Als Programmiersprache haben wir JAVA gewält. Der Vorteil von JAVA ist die existenz zahlreicher Bibliotheken,
die uns ermöglicht haben unsere Ergebnisse zu vergleichen.
Um die Daten einzulesen wurde ein BufferedReader
verwendet. Dazu haben wir verglichen ob die Nutzung eines CSV-Parser Vorteile bringt. Der verwendete Parser (openCSV)
hat bei den relativ einfachen Dateien keine Vorteile gebracht.
Als Datenstruktur haben wir eine Adjazenzmatrix
ausgewählt, da diese nötig ist für folgenden Aufgaben
ist. Außerdem haben wir für die ersten Aufgaben, es auch
mit einer Kantenliste probiert.
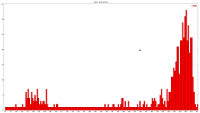
Verteilung der Knotengrade
| Theta |
Mensch |
Schimpanse |
| 0.05 |
 |
 |
Eigenvector centrality
Die Eigenvector Centrality habe wir erst mittels Apache.commons.Math package berechnet. Dieser Package konnte leider die Centrality nicht korrekt ermitteln.
Um die Eigenvector Centrality zu berechnen haben wir also die power iteration implementiert. Die power iteration beruht auf den Frobenius Theorem.
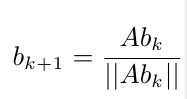
Distanzmatrix
Ausgehen von einer Adjazenzmatrix werden für jeden Knoten, die
direkten Nachbarn nach allen Nachbarn und ihre Entfernung (ausser
Knoten selbst) bis es keine Änderungen mehr gibt.
Graphvergleich mittels Zentratlitätswerten
Vorgehen
Zentralitäswert jeder Knoten wurde durch den Maximalen Wert geteilt,
um Werte zwischen 0-1 zu kriegen. Und dann der Vektor eines Graphen
den des anderen substrahiert.
Gene centrality
Gemeinsamkeit
| p=0.05 | p=0.01 |
|---|
| degree: | 90.34% | 85.85 % |
| eccentricity: | 85.90 % | 90.77 % |
| status: | 95.06 % | 87.60 % |
| centroid: | 89.88 % | 89.69 % |
| betweeness: | 95.27 % | 99.97 % |
| eigenvector: | 89.26 % | 85.46 % |
Unterschiede
| eccentricity: | 14.10 % | 09.23 % |
| status: | 4.94 % | 12.40 % |
| centroid: | 10.12 % | 10.31 % |
| betweeness: | 4.73 % | 0.03 % |
Direkten Graphvergleich
comparison of complete graph
p=0.05
| Human | Chimpanzee | Gemeinsam | Ergebnisse |
|---|
| nr of
edges: | 165777 | 169872 | 143073 | 74.2943046% |
| nr of
vertices: | 704 | 704 | 704 | 100% |
Gemeinsam
| p=0.05 | p=0.01 |
|---|
| nr of edges: | 143073 | 109819 |
| nr of vertices: | 704 | 704 |
| similarity (edges): | 74.29 % | 65.52 % |
| similarity (nodes): | 100 % | 100% |
comparison of cores
p=0.05
| Mensch | Schimpanse | Gemeinsam | Ergebnisse |
|---|
| k | 479 | 480 | | |
| nr of edges: | 120408 | 129968 | 98678 | 65.04 % |
| nr of vertices: | 491 | 511 | 445 | 79.89 % |
p=0.01
| Mensch | Schimpanse | Gemeinsam | Ergebniss |
|---|
| k | 426 | 430 | | |
| nr of edges: | 99611 | 100067 | 71882 | 56.25 % |
| nr of vertices: | 447 | 448 | 380 | 73.79 % |