Cluster 2
Familien
Das Cluster besteht aus zwei Familien mir-2 und mir-71. Zu mir-2 gehört ebenfalls mir-13. Beide zerfallen in Unterklassen.
Vorkommen
Beide Familien (mir-2 und mir-71) kommen ab den Protostomia vor. In den Arthropoda findet sich zudem die paraloge Unterfamilie mir-13.
Geschichte
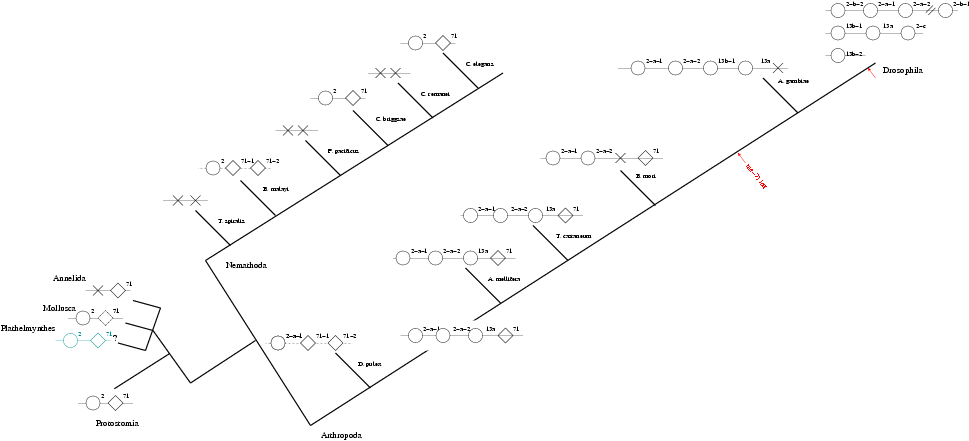
In den Protostomia befinden sich zunächst mir-2 und mir-71 in unmitellbarer Nähe. Im Zweig der Annelida ging die mir-2 verloren, bei Mollusca gibt es sowohl Verteter, die noch über bei miRNAs verfügen, als auch jene, die mir-71 verloren haben. Für die Plathelmynthen wurde mit S. mediterranea nur ein Vertreter betrachtet. Die miRBase-Annotatinen hierfür sind in Frage zu stellen, da viele angebliche miRNAs nicht zum Schema passten. Es lässt sich aber vemuten, dass auch dort mindestens ein mal mir-2 und mir-71 auftreten.
Es folgt eine deutlich Aufspaltung in Arthropoda und Nemathoda. Letztere behielten die Kostellation weitestgehend bei, oder verloren beide miRNAs wieder.
Eine Besonderheit ist bei B. malayi und D. pulex festzustellen. Beide verfügen über eine weitere Kopie der mir-71. Hinweise auf einen gemeinsamem Ursprung der Kopien konnten im Alignment nicht gefunden werden. Zudem treten nirgendwo sonst in den untersuchten Spezies wieder solche Kopien auf. Daher vermute ich, dass beide seperat voneinander in den jeweiligen Spezies entstanden sind. Mir-71-1 ist dabei jeweils die der ursrünglichen mir-71 am nahekommensten Kopie. Außerdem ist anzumerken, dass die Genome der beiden Spezies noch unzureichend genau sequenziert wurden, so dass das Vorkommen der miRNAs zwar bestätigt, ihre Anordnung und Anzahl jedoch nur geschätzt werden kann.
Bei den Arthropoda folgen nun einige veränderungen. Mit A. mellifera kommen zwei Kopien von mir-2 auf. Zu finden ist hier mir-2a-1 und mir-2a-2. Welche davon jedoch näher zur ursprünglichen mir-2 ist, ließ sich nicht feststellen. Die so hinzugekommene mir-13 gehört zur selben Familie wie mir-2. Ihrere Vertreter fallen im Alignment-Baum deutlich zusammen, so dass ein eventuelle Abstammung von mir-2a-1 oder 2 ebenfalls nicht festgemacht werden kann. Mit B. mori findet sich im folgenden eine Spezies, die mir-13a wieder verloren hat, die andere Kopie der mir-2 jedoch beibehielt.
Ab A. gambiae hingegen fehlt die mir-71. Dafür ist mir-13b-1 als Kopie von mir-13a hinzugekommen. Der Strang spaltet ich bei den Drosophilieden auf zwei Chromosomen auf. 2a-1 und 2a-2 finden sich zusammen mit einer weiteren Kopie auf einem eigenen Chromosom. Sehr weit entfernt davon findet sich auf dem selben Chromosom zudem mir-2b-1, dessen Position aufgrund der großen Entfernung jedoch nicht mit der der anderen drei in Zusammenhang gebracht werden kann. Mir-13b-1 und 13-a befinden sich mit einer weiteren Kopie von mir-2, mir-2c auf einem weiteren Chromosom. Schließlich gibt es mit mir-13b-2 es noch eine weitere Kopie der mir-13-1 auf einem extra Chromosom. Die Verteilung auf drei Chromosomen und die große Anzahl von Kopien lässt auf deutliche Duplikations- und Reorganisations-Ereignisse schließen.
Zusätzlich wurden die miRNA Positionen von mir-2b-2, mir-2a-1, mir-2a-2 und mir-2-b-1 über ein Alignment mit 12 Drosophilaspezies ausgeschnitten und aligniert. Es stellte sich klar heraus, dass die miRNAs bei allen Vertretern an den selben Stellen zu finden sind.
Files
