1) Bildliche Darstellung
|
|
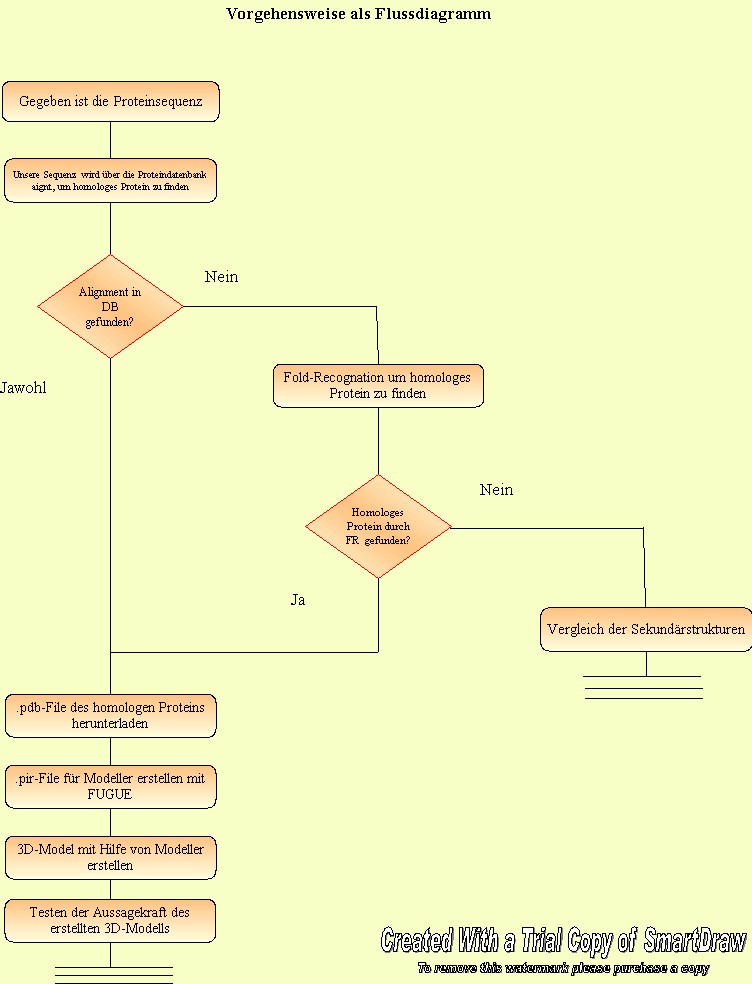
Schritt 1) Zunächst wird die gegebene Aminosäurestruktur gegen viele andere Strukturen aus einer Proteindatenbank alignt. Das Alignment gibt dann Auskunft darüber, ob in dieser Datenbank bereits eine ähnliche, homologe Aminosäuresequenz gespeichert ist. [Tool: blastp; http://www.ncbi.nlm.nih.gov/BLAST/ ; National Center for Biotechnology Information] zurück zum Flowchart Schritt 2.1) Wurde ein guter Alignmentscore zwischen der zu untersuchenden Aminosäuresequenz und einer Sequenz aus der Datenbank ereicht, kann man davon ausgehen, dass es sich dabei um homologe Proteine handelt. Anhand einer weiteren Datenbank ist es nun möglich für das soeben gefundene homologe Protein die Tertiärstruktur, gespeichert in einem .pdb-File, herunterzuladen. [Tool: Protein Data Bank; http://www.rcsb.org/pdb/; Research Collaboratory for Structural Bioinformatics (RCSB)] zurück zum Flowchart Schritt 2.2) Wurde kein Allignment mit einem guten Alignment-Score ermittelt, so wird versucht mittels Fold Recognation eine passende Struktur zu finden. Die Aminosäuresequenz wird hierbei gegen bereits bekannte 3D-Strukturen in einer Datenbank geprüft, bis ein Protein mit einer zu unserer Sequenz passenden Struktur gefunden wird. Das Ergebnis ist dann wieder ein homologes Protein, dessen Tertiärstruktur man aus der Protein Data Bank erhält. [Tool: FUGUE; http://www-cryst.bioc.cam.ac.uk/fugue/ [Tool: 3dpssm; zurück zum Flowchart Schritt 2.3) Wurde weder durch das Alignen in (Schritt 1), noch durch die Fold-Recognation (Schritt 2.2) ein homologes Protein gefunden, so bleibt nur die Auswertung der Sekundärstrukturen. [Tool: ********************* zurück zum Flowchart Schritt 3) Nun wird diese .pdb-Datei dazu genutzt ein Alignment zwischen der zu analysierenden Aminosäuresequenz und der Struktur des homologen Proteins zu erzeugen. Dies geschieht mit dem Internet-Tool Fugue. Ergebnis des Alignmnts ist ein .pir-File, welches für den nächsten Schritt benötigt wird. [Tool: FUGUE; http://www-cryst.bioc.cam.ac.uk/fugue/ ; University of Cambridge, Department of Biochemistry] zurück zum Flowchart Schritt 4) Im folgenden berechnet das Programm "Modeller" ein 3D Modell der Tertiärsruktur für die zu untersuchende Aminosäuresequenz. Zur Berechnung benötigt das Programm drei Dateien. Neben der Datei "model.top" , die Konfigurationszwecken dient sind dies noch zwei Strukturdaten enthaltende Dateien. Zum einen wird das in Schritt 2a erstellte .pir-File benötigt und zum anderen das .pdb-File mit der Tertiärstruktur des homologen Proteins. Anhand dieses Strukturmodells wird nun für die zu analysierende Sequenz die Tertiärstruktur gebildet. Man erhält die Tertiärstruktur in einem .pdb-File. [Tool: Modeller; http://structbio.vanderbilt.edu/comp/soft/modeller/; Vanderbilt University; Ceter fr Structural Biology] zurück zum Flowchart Schritt 5) Um die Aussagekraft der gewonnenen Tertiärstruktur zu prüfen wird das erstellte Modell nun noch zwei Prüfungen unterzogen. Das Programm "Profiler" berechnet aus den beiden Tertiärstrukturfiles, dem Erstellten und dem Homologen, ihren RMS-Wert (mittlerer quadratischer Fehler). Dieser gibt dann nach folgendem Schlüssel Auskunft über die Korrektheit des erstellten 3D-Modells: RMS-Wert < 10 3D-Modell ist brauchbar RMS-Wert > 10 3D-Modell ist unbrauchbar In unserer Auswertung ziehen wir diese Grenze jedoch nicht so eng, sondern betrachten RMS-Werte von bis zu 14 als gut. [Tool: Profiler] zurück zum Flowchart Schritt 6) Ein weiteres Tool berechnet nun noch Energiekurven für das erstellte und das homologe Model. Daraus wird ersichtilich, wie stabil das erstellte Modell ist. Des geschieht anhand sogenannter knowledge-based potentials. [Tool: Prosa II; http://lore.came.sbg.ac.at:8080/CAME/CAME_EXTERN/PROSA ; Center of Applied Molecular Engineering, Institute of Chemestry and Biochemistry, University of Salzburg] zurück zum Flowchart |